Cancer cell | ecDNA利用其超级增强子驱动相分离形成核内凝聚体,高效激活致癌转录
在癌细胞的细胞核中,除了23对染色体,还潜藏着一类游离的环状DNA分子:染色体外环状DNA(extrachromosomal DNA,ecDNA)。这些脱离染色体独立存在的DNA环,携带着扩增数十倍甚至上百倍的癌基因,是癌症基因组中的"超级武器"【1,2】。研究发现,近半数胶质母细胞瘤和多种高侵袭性癌症都携带ecDNA【3】,它们的存在往往预示着肿瘤更凶险、患者生存期更短。ecDNA在细胞分裂时随机分配给子代细胞【4】,导致每个癌细胞携带的ecDNA数量各不相同,这种不公平分配加剧了肿瘤异质性,使癌症更难治疗【5】。
科学家们发现,ecDNA不仅仅通过增加癌基因拷贝数来促癌,它们还具有一种更隐秘的能力——充当"移动增强子"【6】。这些环状DNA携带的超级增强子区域,可以跨越染色体疆界,与远处的染色体基因建立联系,像遥控器一样远程激活这些基因的表达。研究表明,ecDNA倾向于在细胞核中聚集成簇【7】,形成高度活跃的转录中心【8,9】。然而,ecDNA究竟如何实现这种"远程操控"?它们在细胞核中如何组织?这种调控模式在不同癌症中是否具有普遍性?破坏这种调控能否抑制肿瘤生长?

【DOI】10.1016/j.ccell.2025.08.008
【发表时间】2025年9月18日
【IF】一区44.5
【单位/通讯作者】华盛顿大学Chia-Lin Wei
【关键词】ecDNA;核凝聚体/相分离;超级增强子;MED1;移动增强子;染色质相互作用;三维基因组
这项研究首次阐明ecDNA作为"移动增强子"调控癌症基因组的精确分子机制。研究团队通过单分子染色质互作分析、表观基因组学和CRISPR表观编辑等前沿技术,在三种癌症模型中证实:ecDNA携带的超级增强子可招募MED1等转录因子,通过液-液相分离形成核凝聚体,这些凝聚体充当转录枢纽,与数千个染色体基因建立远程互作,反式激活71%的已知癌基因。此外,研究发现破坏ecDNA超级增强子活性或解聚MED1凝聚体,可显著抑制癌细胞增殖并诱导凋亡,为开发靶向ecDNA的新型癌症治疗策略提供了重要靶点和概念验证。
研究思路/技术路线
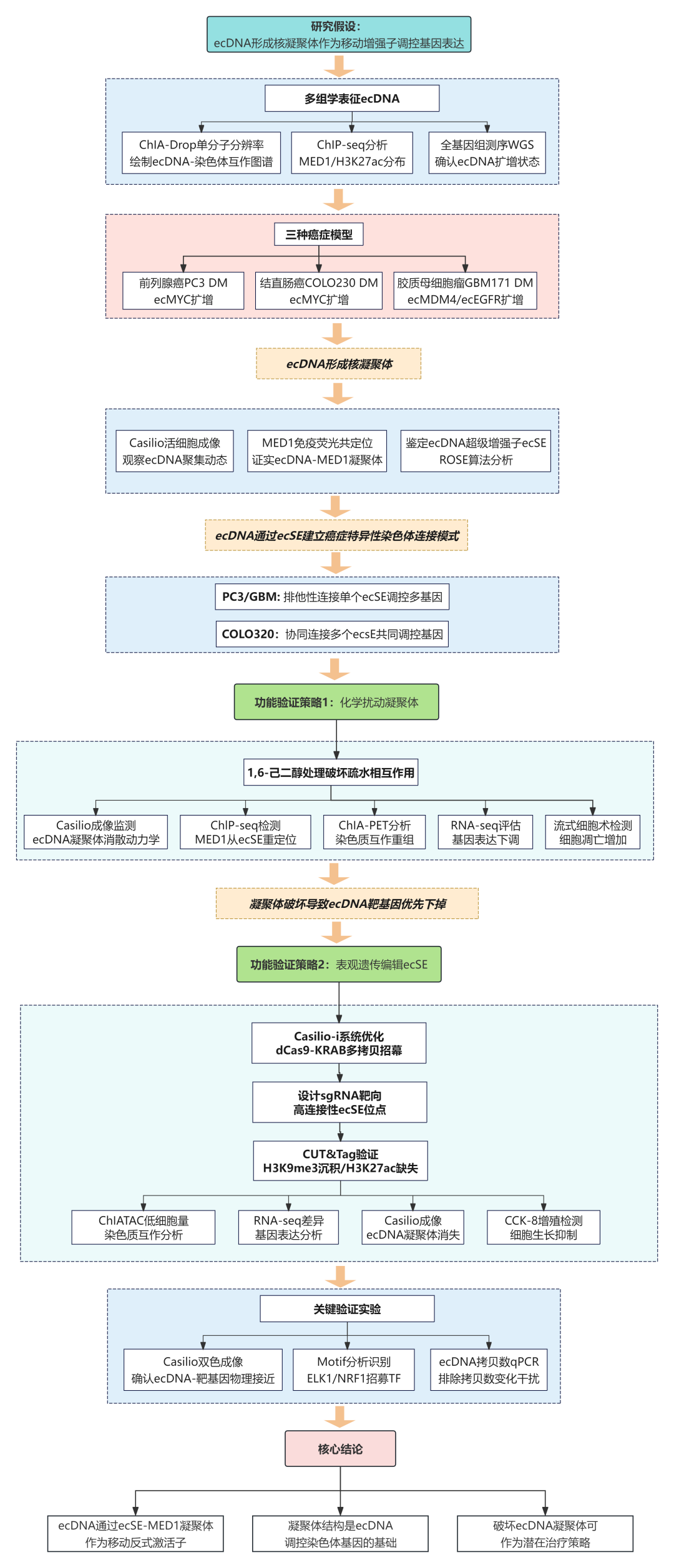
关键研究结果
1、ecDNA通过MED1介导形成核凝聚体并建立多重染色质互作网络

研究利用ChIA-drop技术在三种癌症模型(前列腺癌PC3 DM、结直肠癌COLO320 DM和胶质母细胞瘤GBM171 DM)中系统绘制了ecDNA-染色体互作图谱。ChIA-drop利用微流控技术将单个染色质复合物分配到独立液滴中进行条形码标记,实现了单分子分辨率的多重染色质接触分析。结果显示,三种癌症模型中分别鉴定出14,810、25,876和3,775个ecDNA-染色体接触位点,这些接触位点高度富集MED1结合和H3K27ac活性标记。
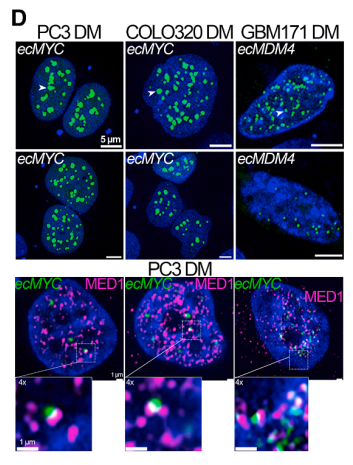
通过Casilio活细胞成像技术验证,ecDNA在细胞核内形成离散的荧光斑点,这些斑点与MED1蛋白免疫荧光信号高度重叠,证实ecDNA聚集体是MED1核凝聚体的组成部分。
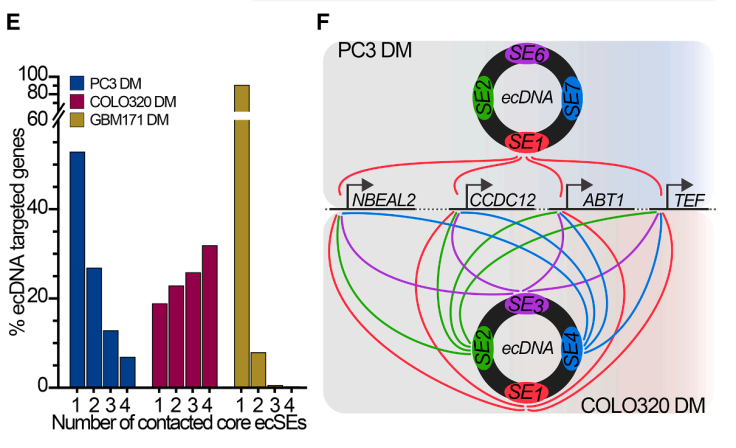
在所有三种癌症中共鉴定出2,299个核心染色体靶基因被ecDNA连接,其中71%的已知癌基因(来自COSMIC和NCG数据库的3,447个癌基因)是ecDNA的互作靶标。不同癌症模型展现出特异性的ecDNA连接模式:在PC3 DM和GBM171 DM中,单个ecDNA超级增强子(ecSE)可独立调控多个基因;而在COLO320 DM中,94%的靶基因同时连接到多个ecSE,呈现协同激活模式。
2、ecDNA超级增强子特异性介导染色质互作和基因激活
通过整合UMI策略的ChIP-seq分析校正拷贝数偏差后,研究在三种癌症中分别鉴定出13、23和24个ecSE。这些ecSE的MED1结合强度比典型染色体增强子高一个数量级,且在全局超级增强子中排名靠前。染色质互作分析显示,仅少数高连接性ecSE作为枢纽,汇聚了绝大多数ecDNA介导的染色质接触,而这些ecSE在染色体同源位点几乎不产生类似的远程互作。
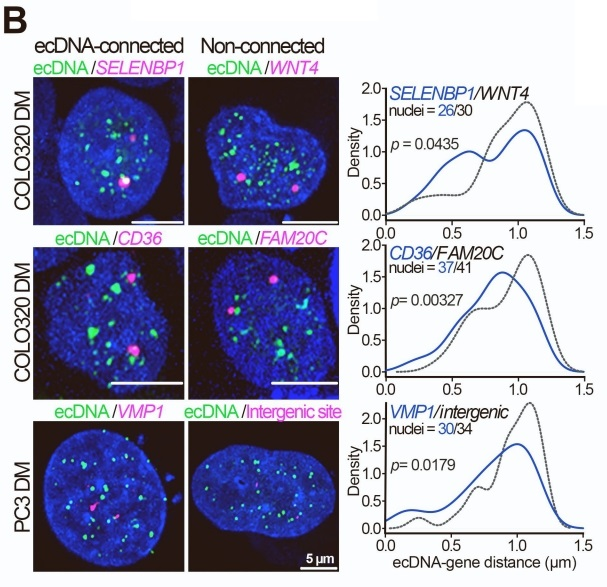
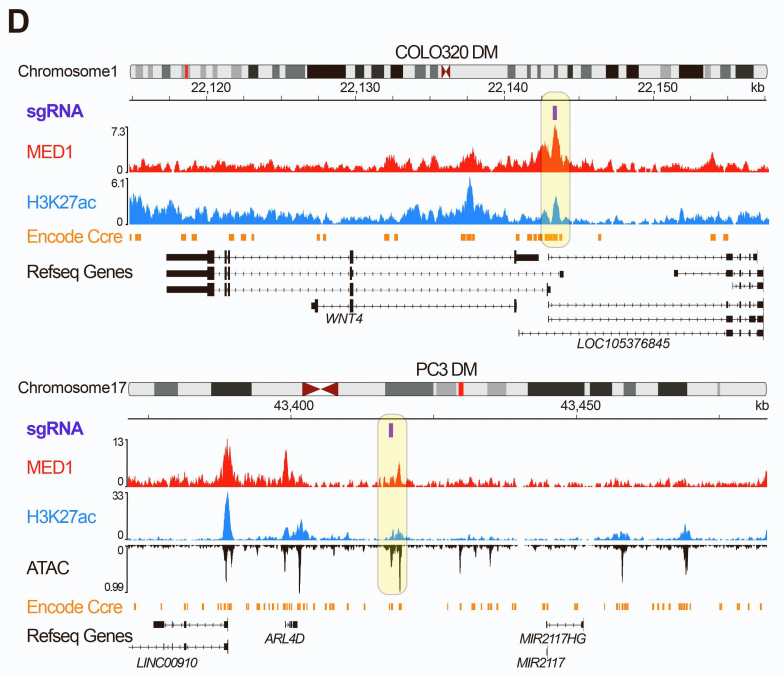
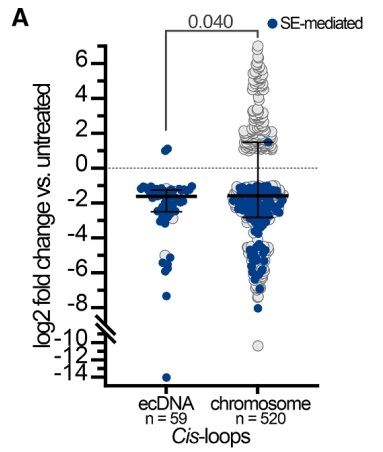
HOMER motif分析揭示,ecDNA-染色体接触区域的MED1结合位点显著富集ELK1和NRF1转录因子结合基序(富集倍数分别为2.3和4.7),提示这些转录因子参与招募MED1到ecDNA染色质复合物。通过Casilio-imaging技术对ChIA-drop鉴定的靶位点进行正交验证,发现ecDNA与靶基因的空间距离显著小于随机对照位点(Wilcoxon秩和检验p<0.05),确认了ecDNA-染色体接触的特异性。
3、快速解聚ecDNA凝聚体并重塑染色质结构

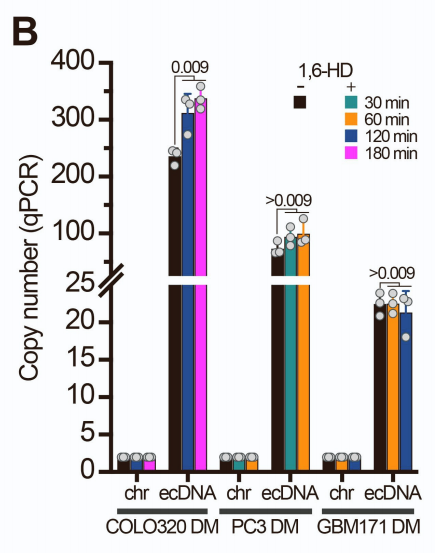
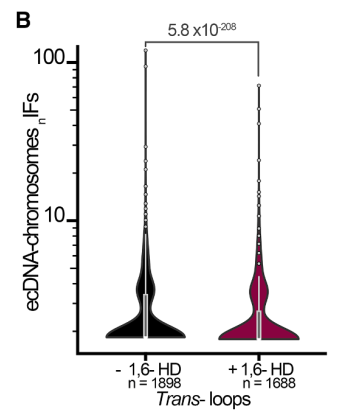
为验证ecDNA聚集体的凝聚体特性,研究使用1,6-己二醇(1,6-HD)处理细胞并通过Casilio活细胞成像追踪ecDNA动态变化。结果显示,1,6-HD处理后ecDNA荧光斑点信号迅速消失,而同一染色体上ecDNA起源位点的荧光信号不受影响。不同癌症模型的ecDNA凝聚体对1,6-HD的响应动力学存在差异:PC3 DM和GBM171 DM细胞中ecDNA斑点在1小时内完全消失,而COLO320 DM细胞中需要155分钟后仍有部分信号残留(A)。qPCR验证确认1,6-HD处理不改变ecDNA拷贝数(B)。
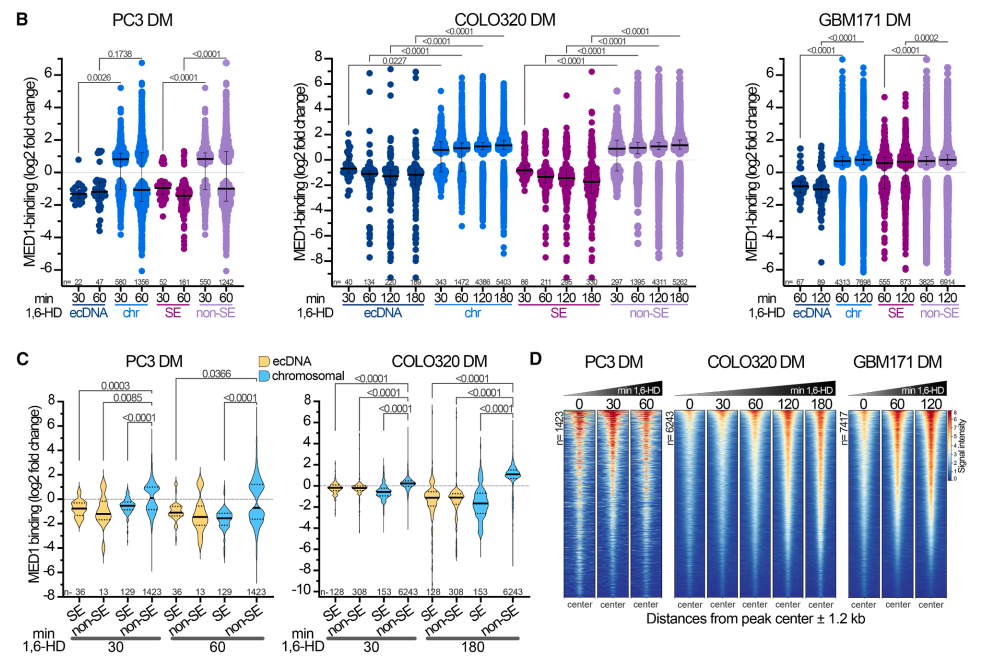
ChIP-seq分析结果显示,1,6-HD处理后ecDNA上的MED1结合显著下降,且下降幅度显著高于染色体超级增强子和非超级增强子区域(B,C)。ChIA-drop分析进一步揭示,1,6-HD处理导致ecDNA-染色体互作网络快速瓦解,ecDNA介导的染色质接触数量大幅减少(D)。这些结果表明,MED1凝聚体是维持ecDNA聚集和染色质互作网络的结构基础。
4、CRISPR表观沉默ecSE阻断靶基因激活
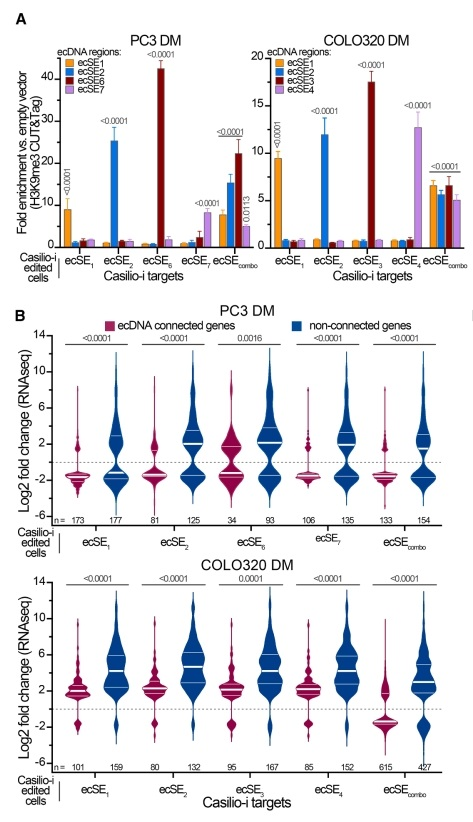
为建立ecDNA增强子与靶基因表达的因果关系,研究采用Casilio-i系统(CRISPR介导的表观沉默技术)特异性失活ecSE。该系统利用dCas9-KRAB融合蛋白和多条sgRNA靶向ecSE区域,诱导局部异染色质化而不改变DNA序列。在PC3 DM细胞中,针对ecSE1(ecDNA上连接性最高的超级增强子)设计了8条sgRNA进行表观沉默。
CUT&Tag分析结果显示,Casilio-i处理后ecSE1区域的H3K27ac活性标记显著下降,而染色体同源位点不受影响,证实了靶向沉默的特异性。
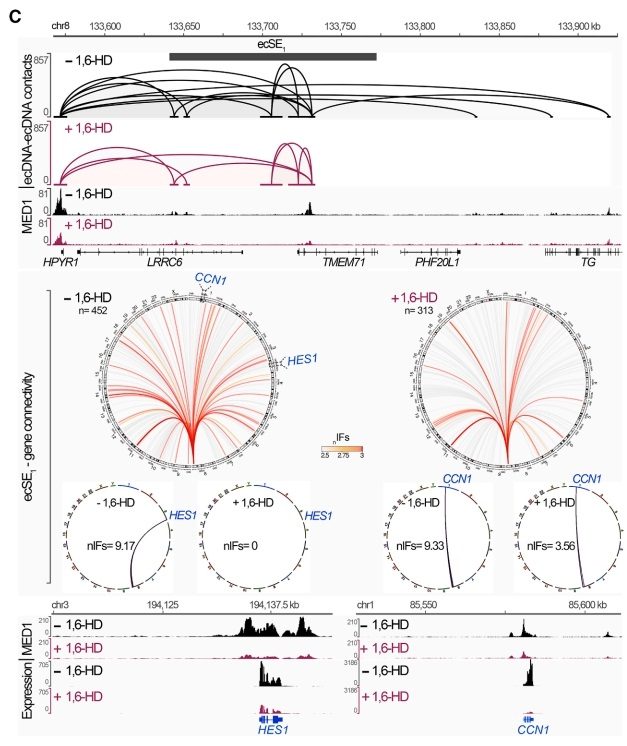
RNA-seq分析表明,ecSE1沉默导致其直接连接的染色体靶基因表达下降40-60%,包括多个癌症相关基因如NBEAL2、CCDC12等。
ChIA-drop分析进一步证实,ecSE1沉默后其介导的ecDNA-染色体互作显著减少,而其他未被靶向的ecSE的互作网络基本保持不变。这些结果直接证明了ecDNA超级增强子对靶基因转录激活的因果作用。
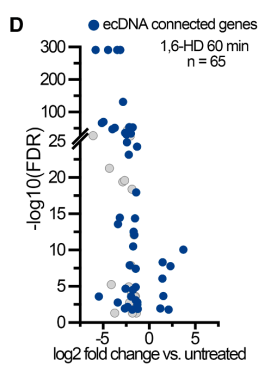
5、破坏ecDNA凝聚体或失活增强子抑制癌细胞增殖并诱导凋亡
研究通过功能实验评估了干预ecDNA凝聚体或增强子活性对癌细胞行为的影响。1,6-HD处理在所有三种癌症模型中均导致细胞增殖显著减少50%以上(CCK-8增殖实验),并诱导细胞凋亡增加2-3倍(Annexin V/PI流式分析)。
类似地,Casilio-i介导的ecSE1表观沉默在PC3 DM细胞中同样显著抑制细胞增殖并促进凋亡。
转录组分析结果显示,1,6-HD或Casilio-i处理后,ecDNA靶基因的表达普遍下调,尤其是参与细胞周期、DNA复制和癌症信号通路的基因。GSEA富集分析结果表明,下调基因显著富集在MYC靶基因、E2F靶基因和G2/M检查点等促增殖通路。Western blot验证了关键癌基因如MYC、MDM4和EGFR的蛋白水平在处理后显著降低。这些结果证实,破坏ecDNA凝聚体结构或失活其超级增强子可有效抑制癌基因表达网络,从而抑制肿瘤细胞生长。
6、ecDNA超级增强子活性维持凝聚体形成并促进肿瘤细胞增殖
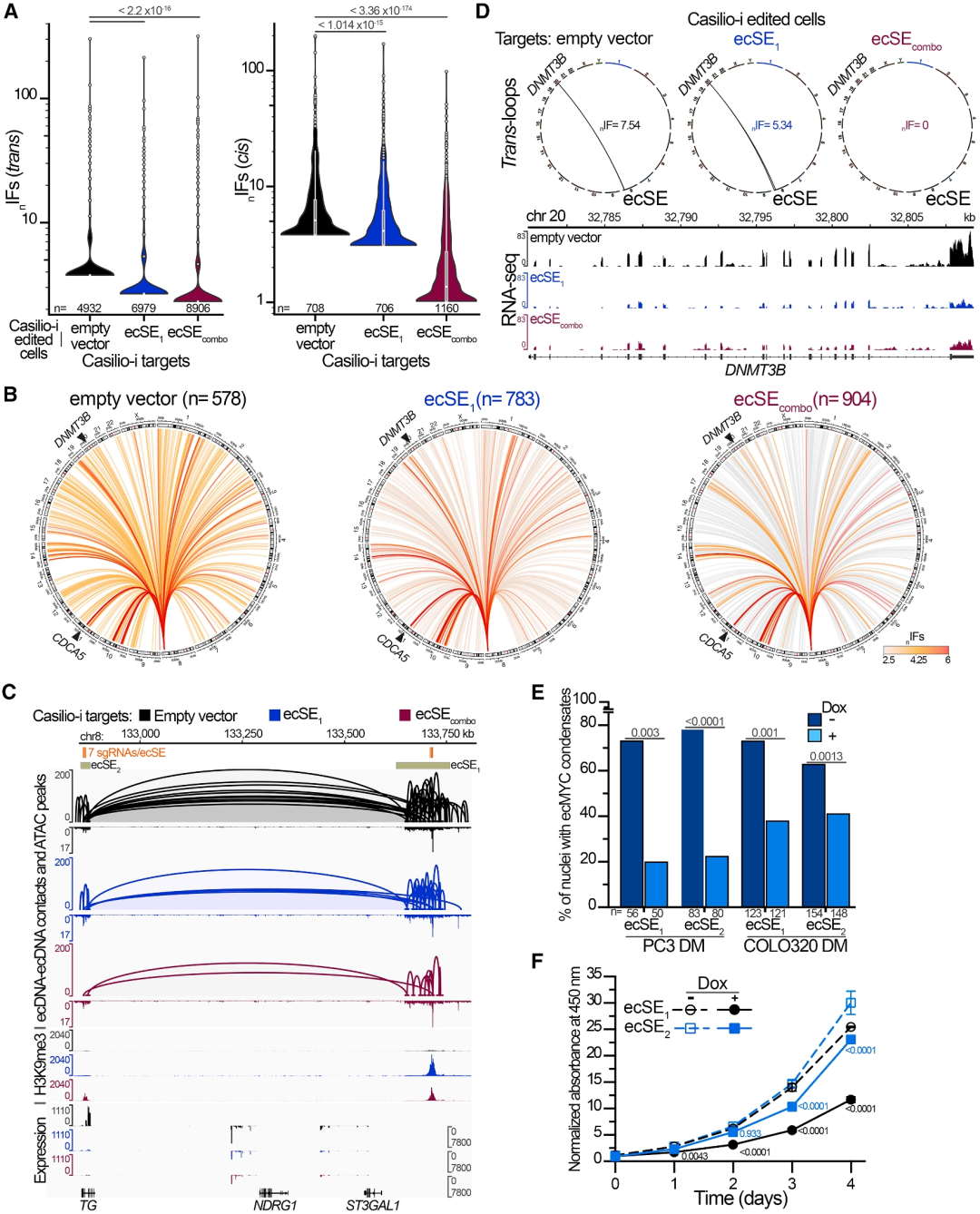
为验证ecSE活性对ecDNA凝聚体形成的重要性,研究通过活细胞成像观察了Casilio-i处理对ecDNA凝聚体的影响。在稳定表达靶向ecSE1或ecSE2的sgRNA的PC3 DM和COLO320 DM细胞中,Dox诱导后含有ecMYC荧光斑点的细胞核数量显著减少,减少幅度高达4倍(E)。qPCR验证确认这种荧光斑点的消失并非由于ecDNA分子的降解,因为Casilio-i处理不影响ecDNA拷贝数(C)。
ChIATAC分析证实,ecSE沉默后ecDNA介导的顺式(ecDNA间)和反式(ecDNA-染色体间)互作频率显著降低(A)。以DNMT3B和CDCA5为例,当所有四个ecSE被沉默后,这些ecSE1靶基因失去了与ecDNA的所有互作,其表达分别下降60%和72%(B-D)。
功能实验显示,ecSE1或ecSE2沉默显著抑制PC3 DM细胞增殖,在4天的对数生长期内增殖率降低超过50%,其中ecSE1扰动的抑制效应更为显著(图5F)。转录组分析表明,ecSE沉默主要影响参与DNA复制、染色质组织调控和核糖体生物合成的基因功能。
这些结果表明,ecDNA超级增强子的活性是维持ecDNA凝聚体形成和促进肿瘤细胞增殖的关键因素。通过破坏ecSE-MED1结合,可以有效瓦解ecDNA凝聚体结构,阻断其反式激活功能,从而抑制肿瘤细胞生长。
研究意义与机制模型
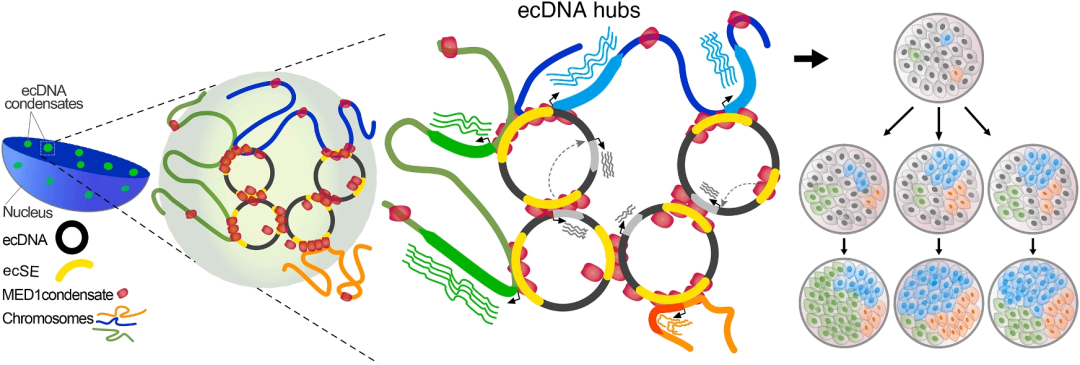
该研究通过整合ChIA-Drop、Casilio双色成像、表观编辑和化学扰动等技术,建立了ecDNA-MED1凝聚体驱动癌基因转录的结构-功能模型:ecDNA通过ecSE招募MED1形成相分离凝聚体,在核内建立转录活性区室;这些凝聚体作为枢纽与特定染色体基因建立多重接触,协同激活包括癌基因在内的靶基因网络;破坏凝聚体或沉默ecSE均可瓦解这一调控架构,导致靶基因表达下调和细胞凋亡。该机制揭示了ecDNA如何通过重塑三维基因组结构驱动肿瘤异质性和演化,为开发针对ecDNA增强子的癌症治疗策略提供了理论基础。
参考文献
[1] Verhaak RGW, Bafna V, Mischel PS. Extrachromosomal oncogene amplification in tumour pathogenesis and evolution. Nat Rev Cancer. 2019;19(5):283-288. doi:10.1038/s41568-019-0128-6
[2] Nathanson DA, Gini B, Mottahedeh J, et al. Targeted therapy resistance mediated by dynamic regulation of extrachromosomal mutant EGFR DNA. Science. 2014;343(6166):72-76. doi:10.1126/science.1241328
[3] Kim H, Nguyen NP, Turner K, et al. Extrachromosomal DNA is associated with oncogene amplification and poor outcome across multiple cancers. Nat Genet. 2020;52(9):891-897. doi:10.1038/s41588-020-0678-2
[4] Turner KM, Deshpande V, Beyter D, et al. Extrachromosomal oncogene amplification drives tumour evolution and genetic heterogeneity. Nature. 2017;543(7643):122-125. doi:10.1038/nature21356
[5] L'Abbate A, Macchia G, D'Addabbo P, et al. Genomic organization and evolution of double minutes/homogeneously staining regions with MYC amplification in human cancer. Nucleic Acids Res. 2014;42(14):9131-9145. doi:10.1093/nar/gku590
[6] Zhu Y, Gujar AD, Wong CH, et al. Oncogenic extrachromosomal DNA functions as mobile enhancers to globally amplify chromosomal transcription. Cancer Cell. 2021;39(5):694-707.e7. doi:10.1016/j.ccell.2021.03.006
[7] Hung KL, Yost KE, Xie L, et al. ecDNA hubs drive cooperative intermolecular oncogene expression. Nature. 2021;600(7890):731-736. doi:10.1038/s41586-021-04116-8
[8] Koche RP, Rodriguez-Fos E, Helmsauer K, et al. Extrachromosomal circular DNA drives oncogenic genome remodeling in neuroblastoma. Nat Genet. 2020;52(1):29-34. doi:10.1038/s41588-019-0547-z
[9] Morton AR, Dogan-Artun N, Faber ZJ, et al. Functional Enhancers Shape Extrachromosomal Oncogene Amplifications. Cell. 2019;179(6):1330-1341.e13. doi:10.1016/j.cell.2019.10.039
- - - 推荐阅读 - - -

Nature | 大脑如何变老?基因组-转录组变化洞察人脑衰老的演化
研究的核心结论是,健康大脑的衰老并非源于神经元功能身份的丧失,而是一场由基因“使用成本”驱动的、普遍性的细胞基础维护能力下降。
2025-10-11

Science | 靶基因如何限制其增强子的作用范围?基因调控新发现
基因调控新发现
2025-09-26

创新组合测序方法揭示转录因子特殊调控机制
转录因子是哺乳动物细胞中一类重要的基因调控元件,很多疾病的发生都与转录因子功能失调有关。2015年,美国麻省总医院及哈佛医学院研究者联合在Science发文,公布了他们关于转录因子与基因调控元件中DNA及RNA之间
2020-06-23
