Epi™ PEmax先导编辑工具

项目简介
传统基因编辑技术(如CRISPR-Cas9)依赖切断DNA双链进行编辑。这种“切割-修复”模式如同创伤性手术,不仅效率受限,还会引入不可预知的突变,带来不确定性与潜在风险。
先导编辑(Prime Editing,PE)则开创了全新的基因编辑范式。Epi™ PEmax无需切断DNA双链,却能像“分子尺笔”一般,直接精准地实现碱基替换、插入或删除。该方法从源头上避免了DNA断裂的副作用,使编辑结果更纯净、安全,尤其在iPS细胞、原代细胞等难编辑类型中优势显著,可为您提供基因型更明确、更可靠的细胞模型。
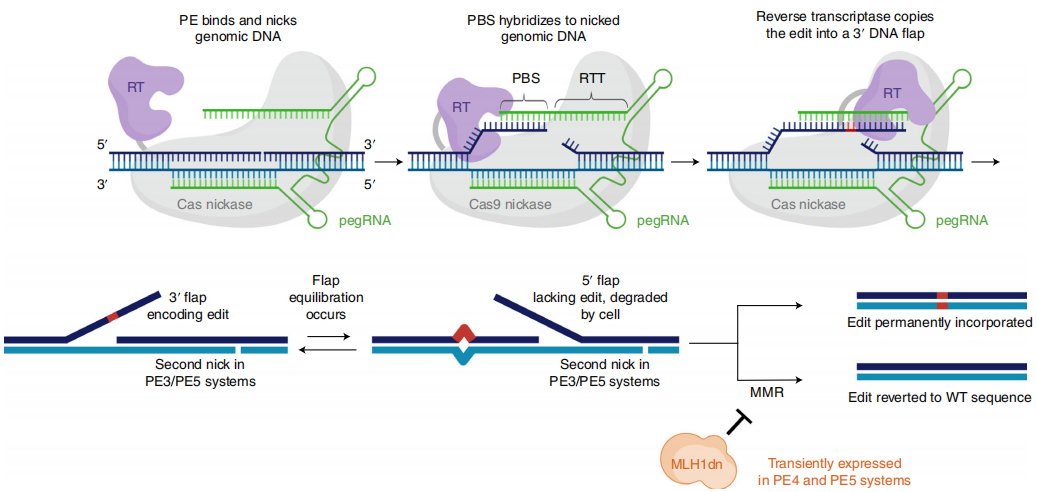
PE原理示意图[1]
机制原理
先导编辑是一种高效的基因编辑方法,它巧妙结合了Cas9蛋白和逆转录酶,通过pegRNA(先导编辑引导RNA)精准改写DNA序列,而不需切割双链。
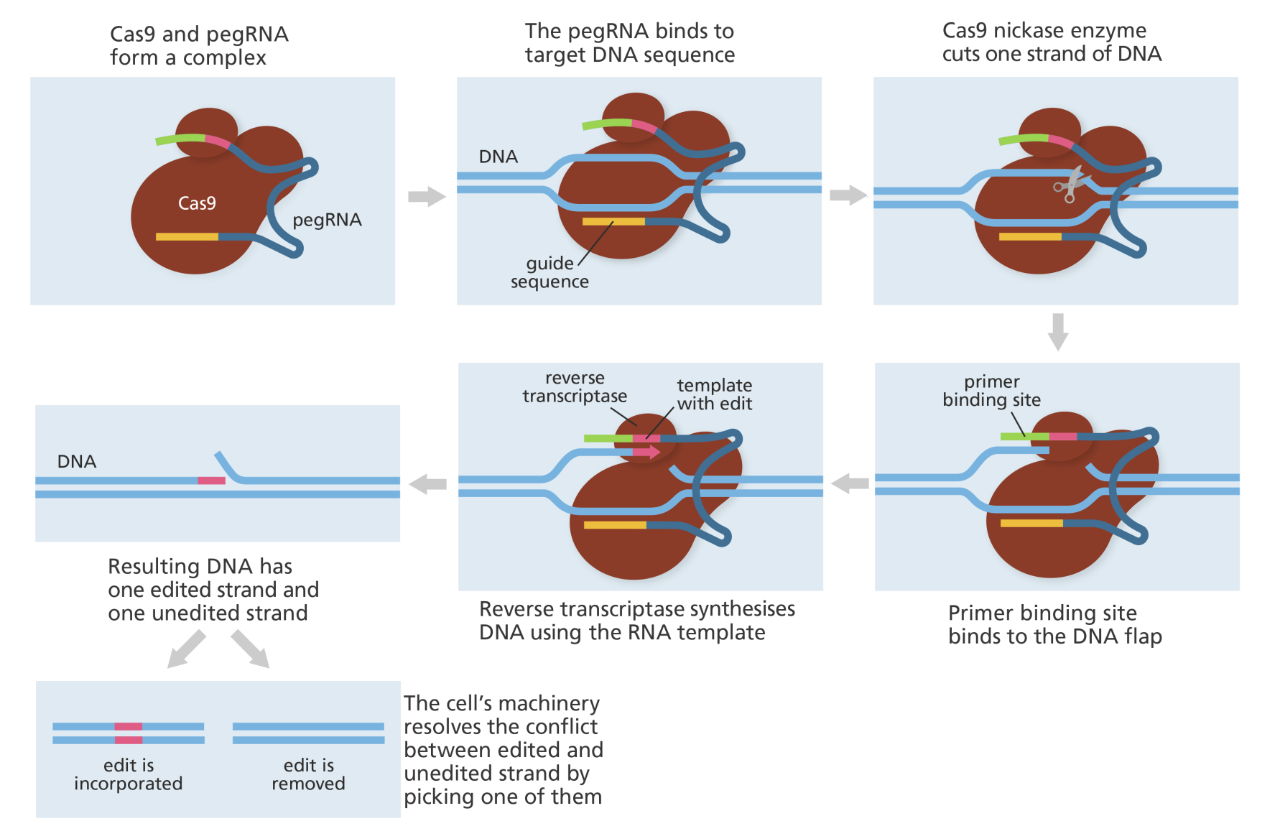
PE技术流程(via:Wellcome Sanger Institute)
流程:Cas9蛋白在pegRNA指引下锁定目标DNA,产生单链切口,暴露3'末端。随后,pegRNA的引物结合位点与切口退火,逆转录酶以此为起点,根据pegRNA模板合成包含编辑内容的新DNA链。最后,通过细胞天然修复机制,新序列整合进基因组,实现碱基替换、插入或删除。通过加入MLH1dn,从而抑制MMR,可以有效保护并固定先导编辑的产物,进而有效提升编辑效率。
这一机制如同精准的基因编辑器,指哪打哪,避免了传统方法的随机错误,编辑效率高、安全性强,适用于多种细胞类型。
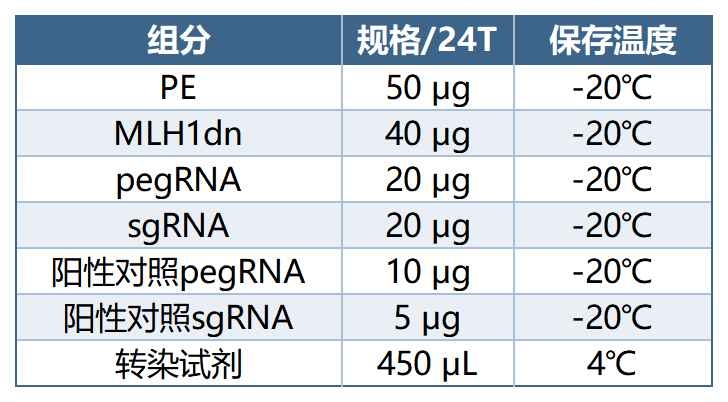
产品规格
核心优势
我们的先导编辑技术,基于经典系统,对核心的Cas9蛋白与逆转录酶进行了优化,从而实现了对目标序列更高精确的改写:
1.卓越精确度:通过专有pegRNA设计算法,可以将编辑成功率提升至传统方法的2.6倍以上,并大幅度降低副产物,确保结果高度可预测[2]。
2.广泛编辑能力:支持12种单碱基转换、小片段插入/删除(1-30 bp),适用于复杂基因组区域。
3.增强编辑能力:通过优化MLH1dn抑制机制,能够有效地提高编辑效率[1]。
1.模型构建:在iPSC中引入疾病突变,创建个性化疾病模型,用于机制探索。
2.功能研究:精确修改蛋白质域,验证氨基酸位点的作用,如信号通路调控。
3.调控元件分析:编辑非编码区(如增强子),量化对转录的影响,支持表观遗传学研究。
4.药物开发:生成稳定细胞株,进行高通量筛选,加速靶点验证。
应用场景
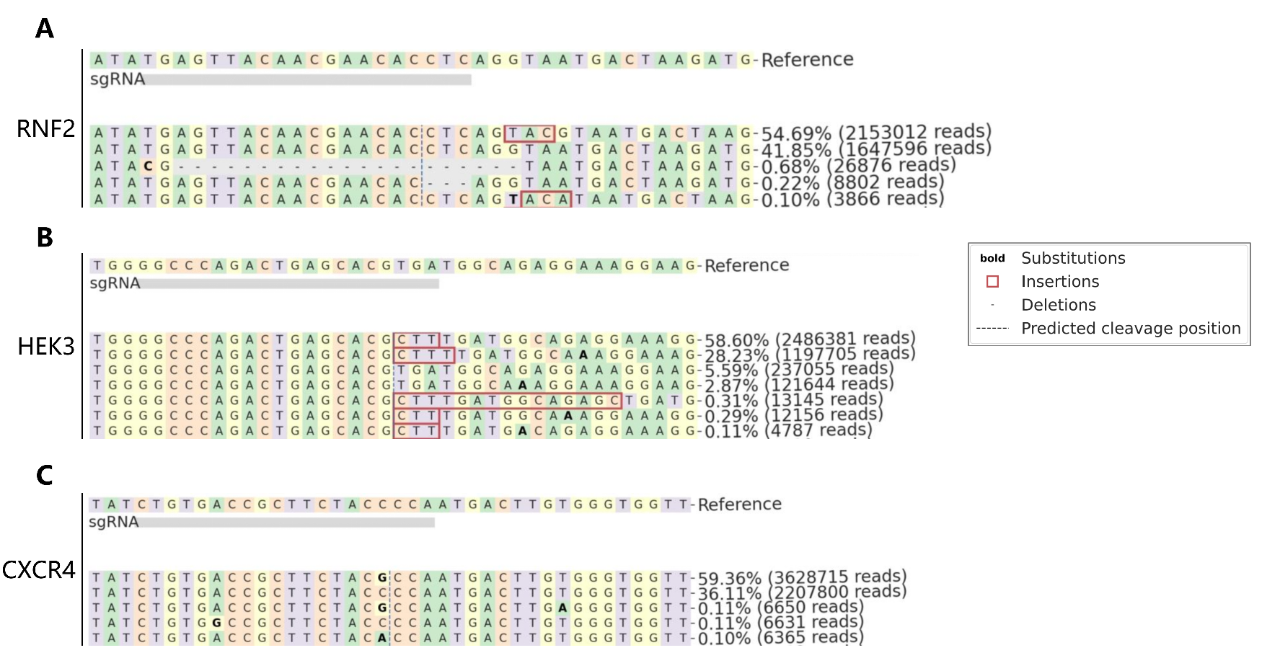
Epi™ PEmax的二代测序结果基因组比对
Epi™ PEmax实测数据
A:
基因名称:RNF2
编辑类型:插入TAC
编辑效率检测:RNF2基因插入TAC碱基的编辑成功率可达54.69%
B:
基因名称:HEK3
编辑类型:插入CTT
编辑效率检测:HEK3基因插入CTT碱基的编辑成功率可达58.6%
C:
基因名称:CXCR4
编辑类型:单碱基G替换C
编辑效率检测:CXCR4基因单碱基G替换C的编辑成功率可达59.36%
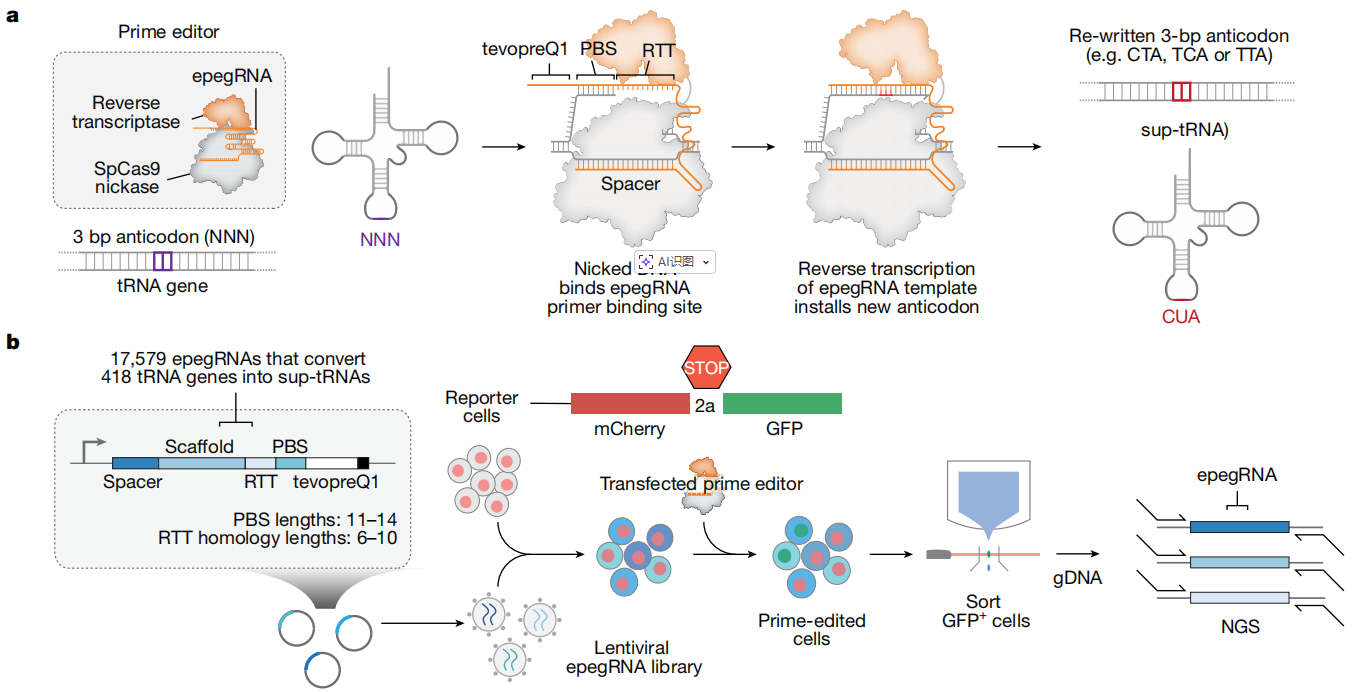
文章使用先导编辑技术的目的是通过精准编辑基因组中的冗余tRNA基因,将其永久转化为高效抑制性tRNA(sup-tRNA),从而通读由无义突变引起的多种遗传病的提前终止密码子,实现一种疗法治疗多种疾病的“疾病无关性”策略。
相关研究案例
1.通用基因编辑疗法:
Nature:应用先导编辑安装抑制性tRNA实现疾病无关性的基因组编辑[3]

在Batten病、Tay-Sachs病及Hurler综合征小鼠模型等多种疾病背景下,成功恢复了20%-70%的酶活性或全长蛋白表达,几乎完全逆转了疾病相关的病理积累,且全基因组范围未检测到脱靶效应,证明了其高效性与安全性。
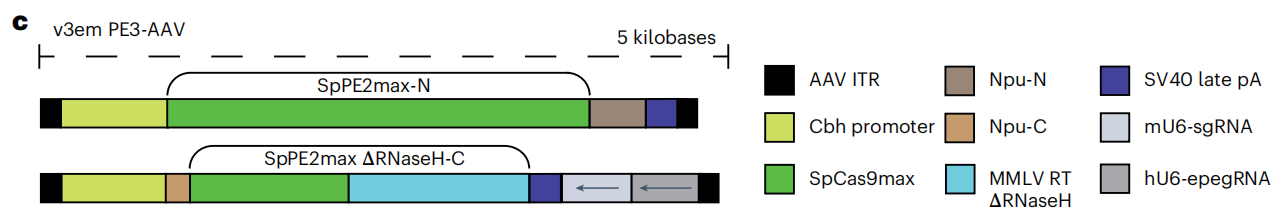
这项研究使用先导编辑技术去系统性地识别并解决腺相关病毒(AAV)体内递送先导编辑器的瓶颈(如蛋白质表达量、pegRNA稳定性和DNA修复),开发出新型的双AAV载体系统(v3em PE-AAV),以实现对成年小鼠重要器官的高效基因编辑。
2.体内递送优化
Nat Biotechnol:应用双AAV系统实现小鼠多器官高效先导编辑[4]
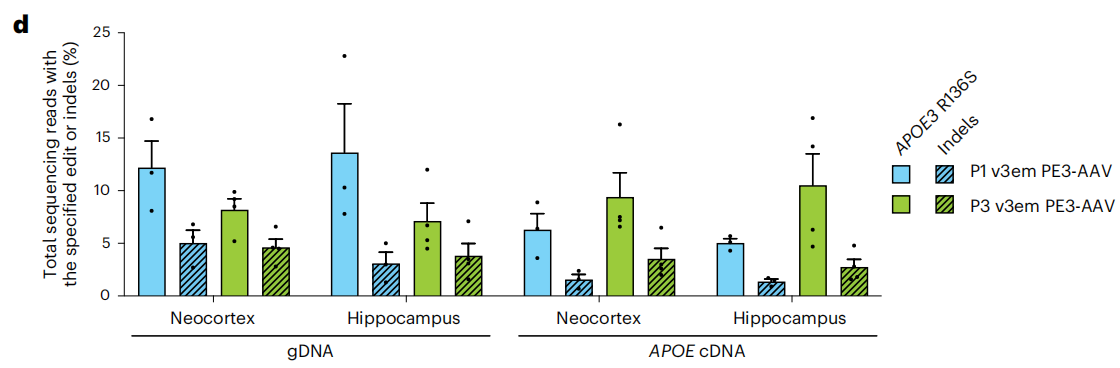
优化后的双AAV系统(v3em PE-AAV)在肝脏中实现了46%的编辑效率,在心脏中的效率也提升至9.1%。研究进一步应用此系统,成功在体内安装了与阿尔茨海默病(APOE Christchur ch突变)和冠状动脉疾病(Pcsk9保护性突变)相关的基因突变,且全基因组范围分析未检测到脱靶编辑,证明了其高效性与安全性。
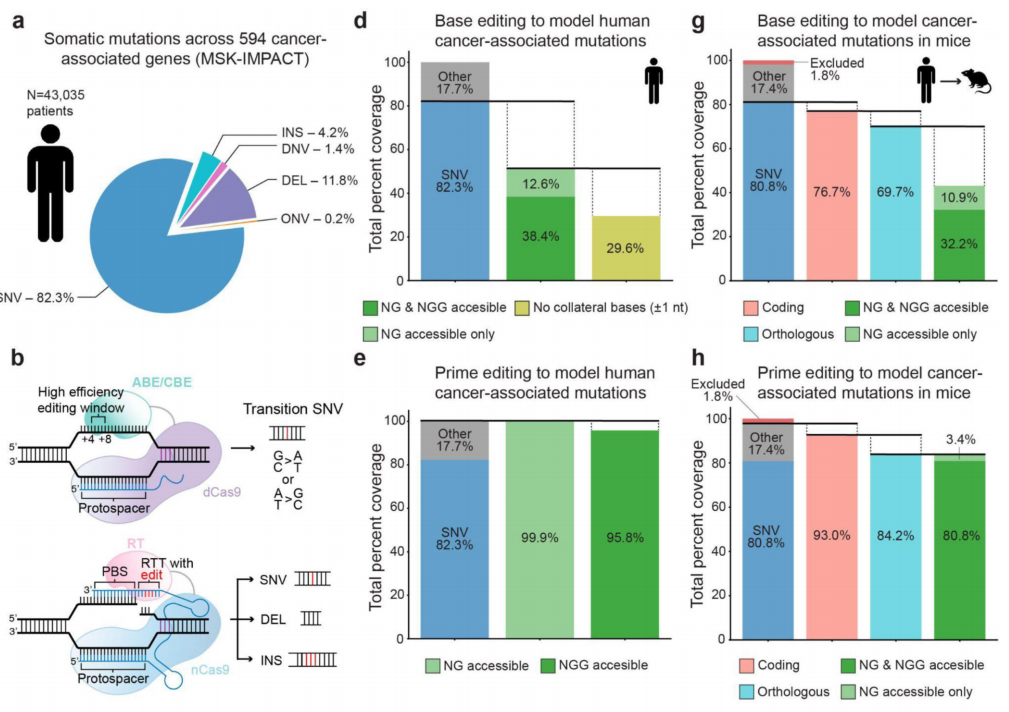
作者分析人类癌症基因组数据,旨在量化PE相较于碱基编辑(BE)在模拟癌症相关突变方面的理论优势,结果显示PE理论上能覆盖绝大多数(>95%)的突变类型,特别是在鼠类中保守的编码区突变。
3.基因编辑动物模型
Nat Biotechnol:构建条件性表达先导编辑器的小鼠模型以在体内模拟广泛的体细胞突变[5]
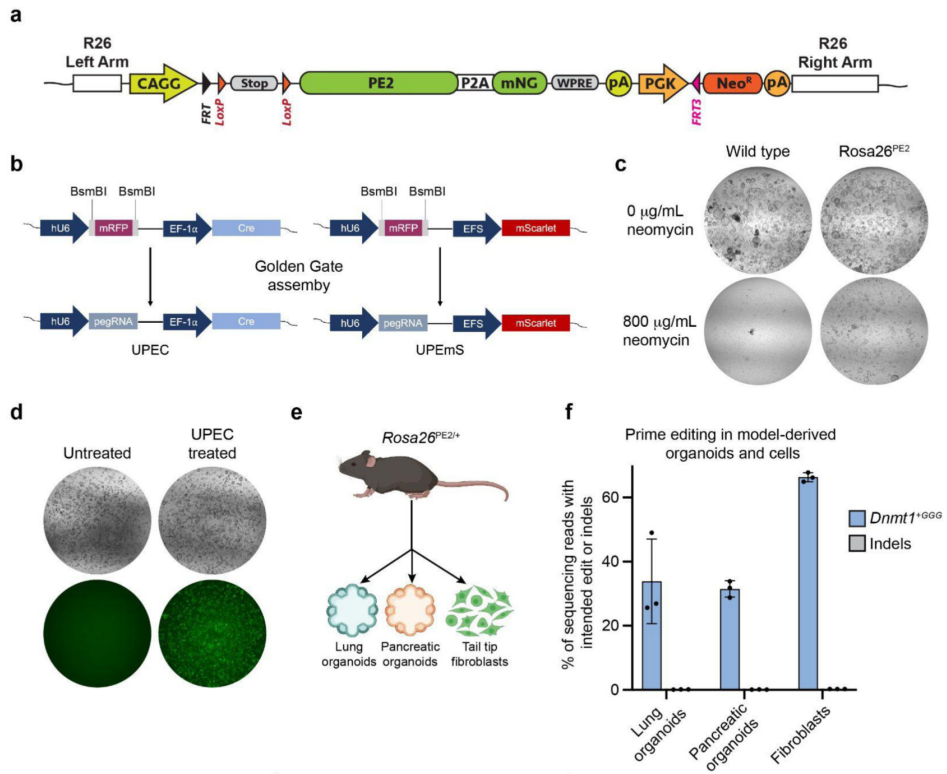
通过构建并验证一种Cre诱导型Rosa26-PE2基因敲入小鼠模型,旨在建立一个无需外源递送大分子编辑器的体内多功能编辑平台,结果证实该模型能在多种原代细胞、类器官及小鼠肝脏中实现高效、精确的基因编辑。此外,还通过体外PE精确安装了与癌症相关的Kras和Trp53突变,目的是验证该模型对特定功能突变的编辑能力与纯度,结果显示编辑效率高、副产物少,且突变具有预期的生物学效应(如药物敏感性)。
参考文献
[1]Doma, Jordan L et al. “Designing and executing prime editing experiments in mammalian cells.” Nature protocols vol. 17,11 (2022): 2431-2468.
[2]Anzalone, Andrew V et al. “Search-and-replace genome editing without double-strand breaks or donor DNA.” Nature vol. 576,7785 (2019): 149-157.
[3]Pierce SE, Erwood S, Oye K, et al. Prime editing-installed suppressor tRNAs for disease-agnostic genome editing. Nature. 2025;648(8092):191-202.
[4]Davis JR, Banskota S, Levy JM, et al. Efficient prime editing in mouse brain, liver and heart with dual AAVs. Nat Biotechnol. 2024;42(2):253-264.[5]Ely ZA, Mathey-Andrews N, Naranjo S, et al. A prime editor mouse to model a broad spectrum of somatic mutations in vivo. Nat Biotechnol. 2024;42(3):424-436.
