Active Ribo-seq:活跃翻译组测序技术

项目简介
Active Ribo-seq:活跃翻译组测序技术
常规Ribo-seq技术可以检测转录本上核糖体的分布、翻译活性,是阐述翻译机制的经典研究工具,但无法区分翻译中的活跃核糖体和非活跃的核糖体。表观生物为了满足生物医学领域对翻译组测序技术的更高需求,在国内率先引进意大利IMMAGINA Biotechnology公司RiboLace技术,推出活跃翻译组测序 Active Ribo-seq 服务。这是一种基于生物素修饰嘌呤霉素(3P复合物)分离活跃核糖体的技术,无需抗体和标签,所需样本量比传统Ribo-seq减少90%以上,可快速高效地绘制全转录组范围的活跃核糖体图谱,达到单碱基分辨率。
应用与优势
技术应用(联合longRNA-seq)
1. 检测转录本上活跃核糖体的分布、翻译活性
2. 推测翻译起始位点、ORF 位置
3. 确定蛋白翻译效率
4. 探究翻译调控和基因表达情况
5. 鉴定新蛋白 / 新短肽
技术优势
1. 识别处于翻译转态的RNA分子,准确描绘翻译中的核糖体图谱
2. 样本量要求更低,可低至单细胞水平
3. 高精度的活跃核糖体图谱,达到单碱基分辨率
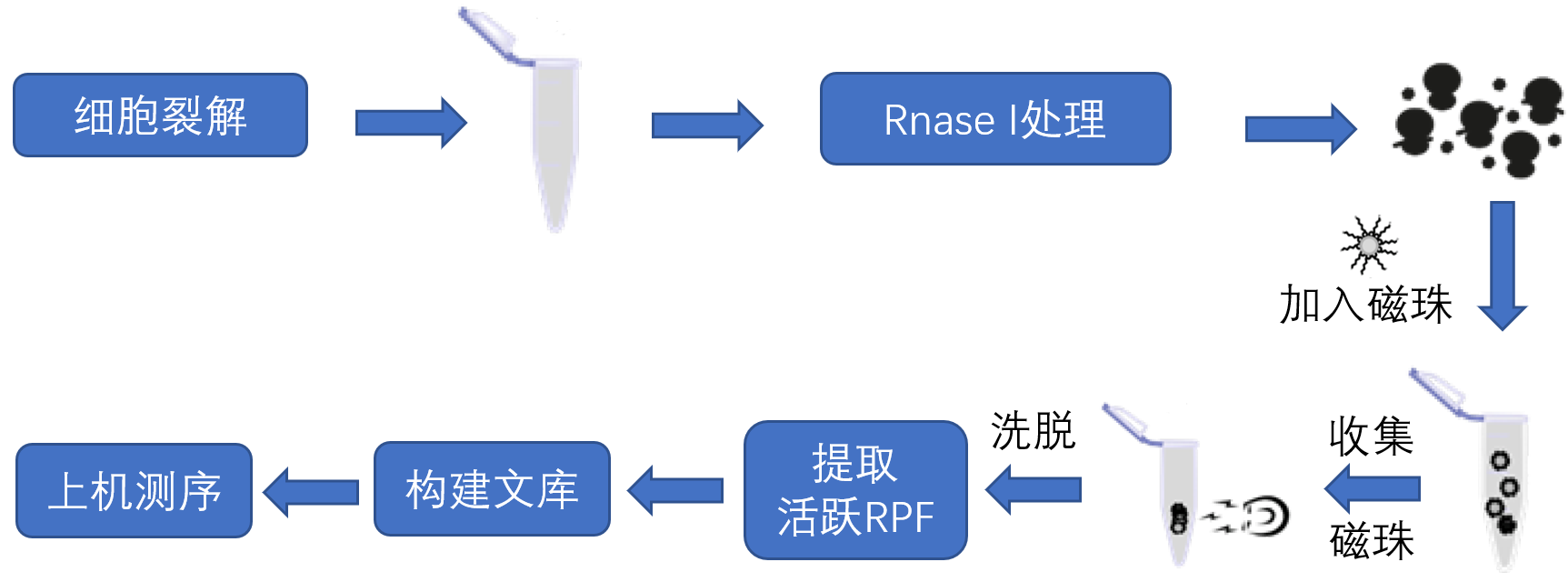
技术流程
送样要求
≥ 1×10⁶个细胞/样本
细胞
≥ 50 mg/样本
组织
仅限人、大小鼠
样本物种
样品分组
1. 常规要求至少 2 组样品,包括对照组和实验组(临床样本为正常人组和患者组)
2. 每个样品均进行翻译组和转录组测序
3. 样本数建议:3 VS 3
分析内容
Active Ribo-seq
1. 全基因组翻译活性分析
2. 基因翻译效率计算
3. 差异翻译效率基因分析
4. 差异翻译效率基因 GO 分析
5. 差异翻译效率基因 KEGG 分析
6. 起始密码子预测(包括非 ATG 起始)
7. ORF(开放阅读框)位置预测
8. 新蛋白 / 新短肽鉴定
9. 密码子使用频率差异分析
10. lncRNA / circRNA 编码能力预测
对照 longRNA-seq 分析
1. 数据质控
2. 基因比对与统计
3. mRNA 差异表达分析
4. lncRNA 差异表达分析
5. 差异基因 GO 分析
6. 差异基因 KEGG 分析
7. circRNA 差异表达分析
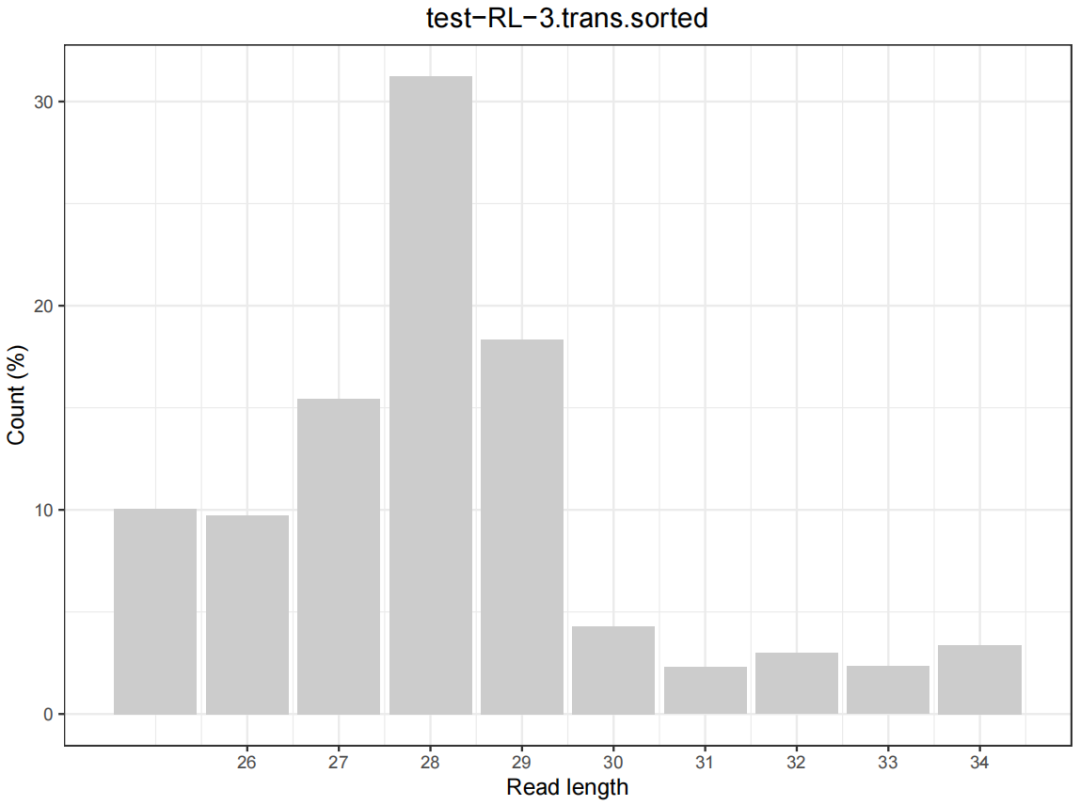
图1. RiboLace Ribo-seq 比对到基因组的 read 的长度分布情况
表观生物实测数据
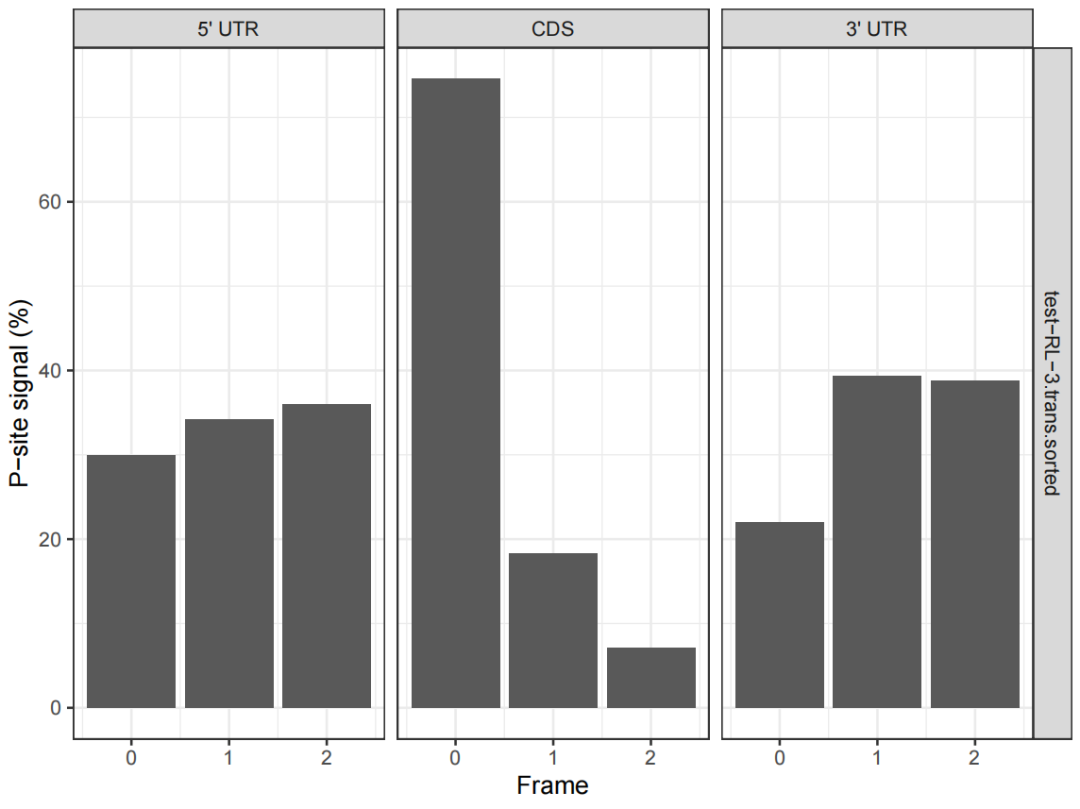
图2. P-site 信号在 5'UTR、CDS、3'UTR 区间的分布
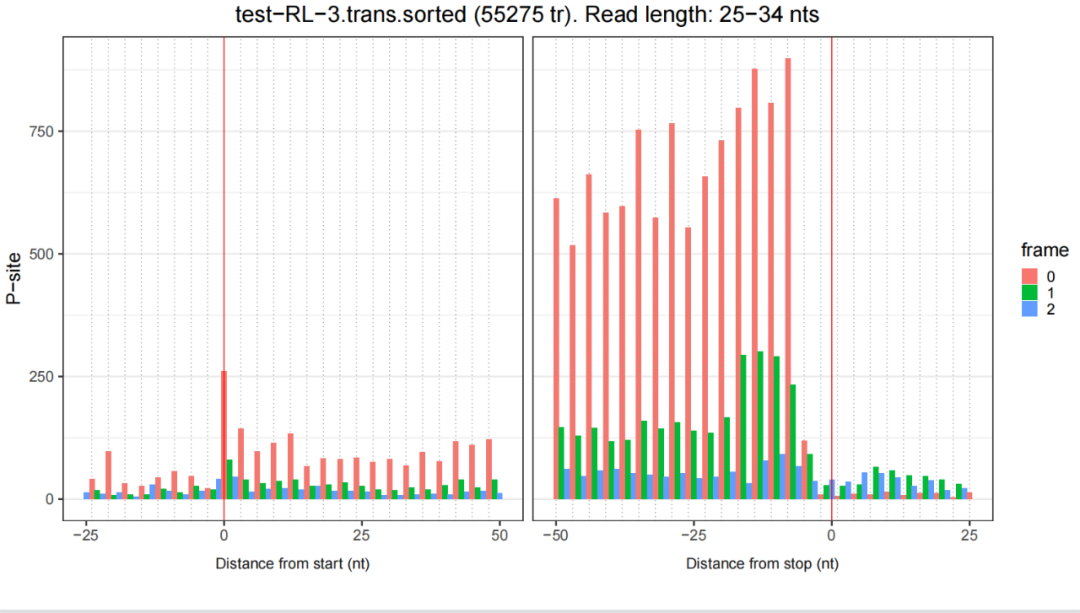
图 3. RiboLace 呈现典型的周期性 3-nt 分布
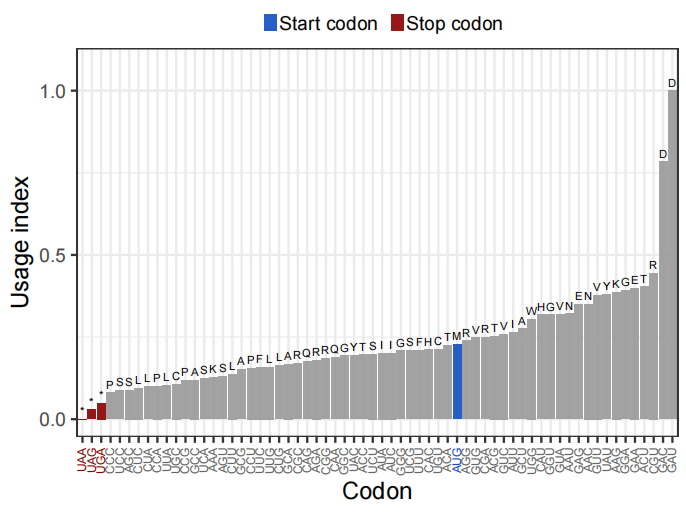
图 4. 不同密码子使用频率分析

图5. RiboLace和转录组测序联合分析热图揭示高翻译效率的基因含有特定的RBP motif
参考案例
Nat Commun:单细胞转录组和翻译组学跨组学联合揭示人卵母细胞成熟的潜在机制[1]
将转录组和翻译组跨组学联合分析比单独使用转录组更准确,特别是在哺乳动物卵母细胞这类受翻译调节控制的细胞类型。在这项研究中,研究者结合了RiboLace和转录组测序,揭示了小鼠和人类卵母细胞之间不同的翻译表达模式,并阐述了人类卵母成熟过程中从细胞质到细胞核的时空顺序调节,还发现了OOSP2诱导因子在人卵母细胞成熟中的作用。针对单卵母细胞RiboLace和转录组测序分析进一步阐明,OOSP2通过翻译调节诱导包括了小GTP酶的特异信号通路。
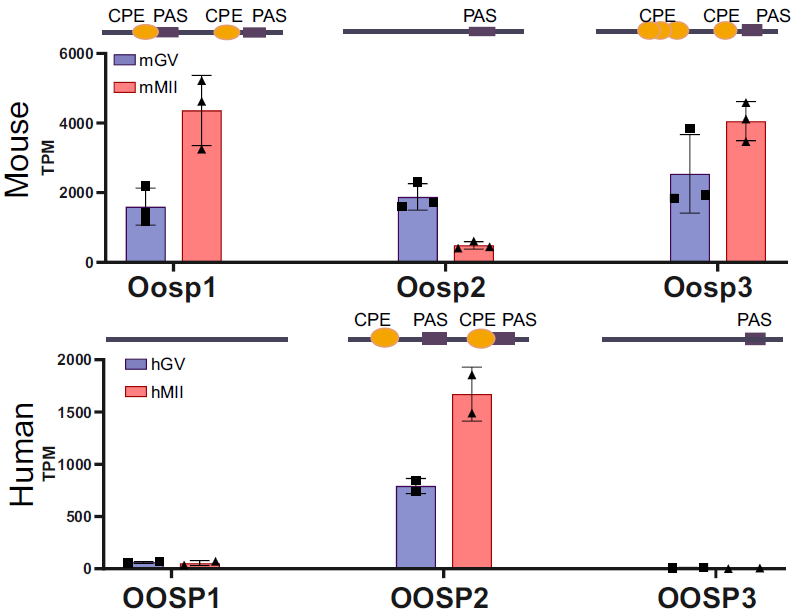
图6.人和小鼠的GV和MII卵母细胞中OOSP基因家族的翻译表达情况,3’UTR中的CPE元件和PAS信号与翻译表达密切相关
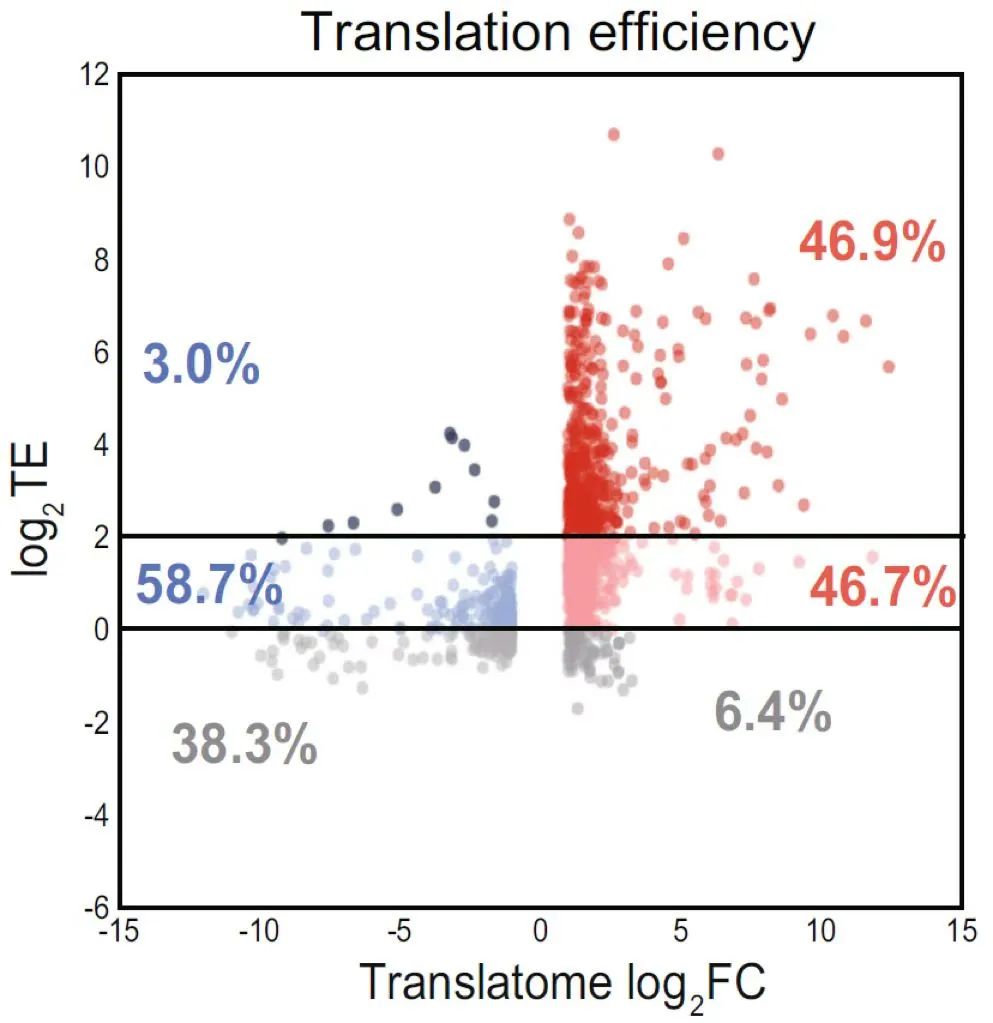
图7. 单细胞RiboLace与转录组测序,OOSP2处理后的翻译效率差异火山图
参考文献
[1] Hu W, Zeng H, Shi Y, et al. Single-cell transcriptome and translatome dual-omics reveals potential mechanisms of human oocyte maturation. Nat Commun 2022 Aug 30;13(1).
[2] Zhang C, Wang M, Li Y, et al. Profiling and functional characterization of maternal mRNA translation during mouse maternal-to-zygotic transition. Sci Adv 2022 Feb 04;8(5)
[3] JaegerAM, Stopfer LE, Ahn R, et al. Deciphering the immunopeptidome in vivo reveals new tumour antigens. Nature 2022 Jul;607(7917)
