m1A-seq
项目简介
N1-methyladenosine(m1A)是一种重要的RNA转录后修饰,是RNA分子腺嘌呤第1位氮原子上的甲基化修饰,首次报道是在50多年前【1】。2016 年,何川等人【2】发现m1A对甲基化mRNA的翻译具有促进作用,存在于多种不同的真核细胞基因转录本中,包括酵母菌到哺乳动物,使我们重新认识m1A。越来越多的研究发现,m1A不仅影响局部RNA结构、蛋白-RNA相互作用以及5’UTR的翻译,还存在于tRNA、rRNA、mRNA和线粒体转录本【3】,在维持这些ncRNA的正确结构和功能上发挥重要的作用【4】。m1A修饰的相关研究初露峥嵘,提示我们这个新的 RNA 甲基化修饰有着巨大的研究意义。表观生物一直专注于表观遗传学研究领域,推出 m1A-seq 整体服务,绘制全转录组 m1A 甲基化修饰图谱帮助客户揭示 m1A 修饰的未知功能与机制。
送样要求
样本物种: 仅限人、大小鼠物种,其他物种需评估
≥ 100 μg/ 样本
总RNA
≥ 3 X107 个细胞 / 样本
细胞数量
≥ 100 mg/ 样本
组织
分析内容
基本分析内容
1. 原始数据过滤及质控
2. m1A 的基本特征
• Peak 在基因元件的分布
• Reads 在基因元件的分布
• Peak 关联基因的特征
3. 参考基因组比对
4. Reads 在染色体上的分布
5. Peak Calling 分析
6. Peak 可视化 差异 Peak 分析
7. Peak 统计分析 差异 Peak 关联基因 GO,KEGG 分析
8. Motif 分析
高级分析内容
1. RNA-seq 表达差异与 m1A 修饰差异关联分析
2. m1A 修饰差异与 GWAS 数据库关联分析
3. 翻译组 Ribo-seq 翻译差异与 m1A 修饰差异关联分析
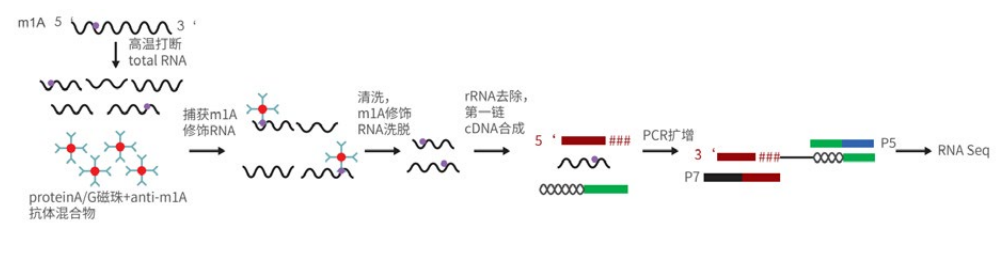
技术原理
将RNA片段化,利用m1A抗体进行免疫沉淀实验。洗脱RNA片段,反转录成cDNA,进行高通量测序。
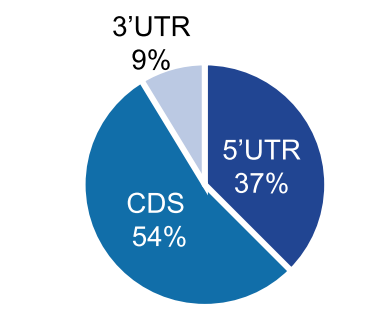
图1. Peaks 结构百分比图【2】
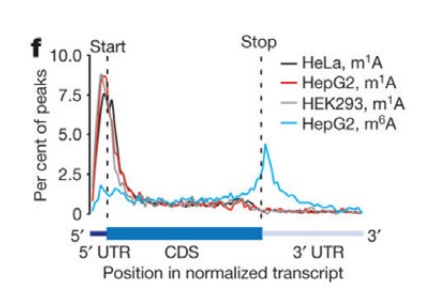
图 2. m1A修饰位点富集于AUG起始密码子附近【2】
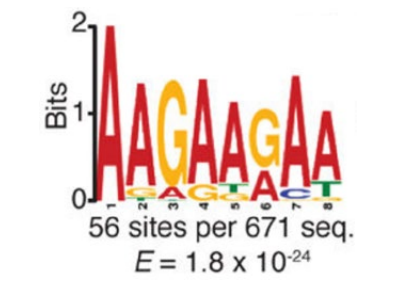
图3. m1A修饰motif分析【2】

图4. 某特定基因的m1A修饰区域【2】
客户文章
PNAS:RNA 修饰通过 ATP5D 调控肿瘤细胞糖酵解 [8]
研究者通过测序和功能研究证实,ATP5D 是 ATP 合成酶最重要的亚单位之一,参与 m1A 去甲基酶 ALKBH3 调节的癌症细胞糖酵解。m1A 修饰的 ATP5D 外显子1,通过增加与 YTHDF1/eRF1 复合物的结合来负调控其翻译延伸,从而促进核糖体复合物释放 mRNA。m1A 还调节 E2F1 的 mRNA 稳定性,E2F1 直接与 ATP5D 启动子结合,启动它的转录。利用dm1 ACRISPR 系统,靶向 ATP5D m1A,去甲基化,显著提高了 ATP5D 的表达和癌症细胞的糖酵解。体内实验证明 m1A/ATP5D 在肿瘤生长和癌症进展中的作用。此研究揭示了 mRNA m1A 修饰和细胞代谢的联系,对这些相互作用的进一步理解,对癌症治疗至关重要。
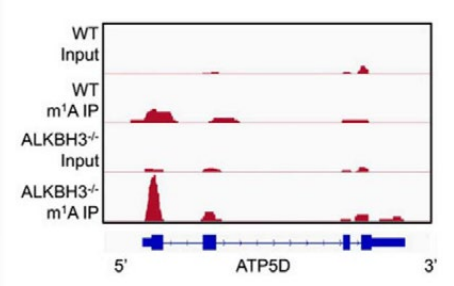
图5. m1A-seq显示野生组和ALKBH3-/-HeLa细胞ATP5D基因CDS区的m1A富集峰
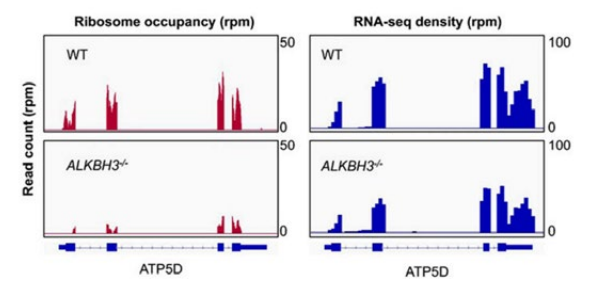
图6. 两组细胞的ATP5D基因的mRNA表达水平(RNA-seq) 与核糖体足迹(Ribo-seq)
参考文献
[1] Maki RA, Brown JL, Cummings DJ. Transfer RNA methyltransferase activity in paramecium aurelia. Biochim Biophys Acta. 1976;425(3):334-341. doi:10.1016/0005-2787(76)90260-4
[2] Dominissini D, Nachtergaele S, Moshitch-Moshkovitz S, et al. The dynamic N(1)-methyladenosine methylome in eukaryotic messenger RNA. Nature. 2016;530(7591):441-446. doi:10.1038/nature16998 [3]Zhang C, Jia G. Reversible RNA Modification N1-methyladenosine (m1A) in mRNA and tRNA. Genomics Proteomics Bioinformatics. 2018;16(3):155-161. doi:10.1016/j.gpb.2018.03.003
[4] Roundtree IA, Evans ME, Pan T, He C. Dynamic RNA Modifications in Gene Expression Regulation. Cell
. 2017;169(7):1187-1200. doi:10.1016/j.cell.2017.05.045
[5] Wu Y, Chen Z, Xie G, et al. RNA m1A methylation regulates glycolysis of cancer cells through modulating ATP5D. PNAS. 2022;119(28):e2119038119. doi:10.1073/pnas.2119038119
