Hi-C
项目简介
Hi-C
Hi-C(High-throughput chromosome conformation capture),即高通量染色体捕获技术,可在全基因组范围研
究 DNA 的相互作用,描绘高分辨率的染色质三维结构图谱,得到 A/B compartment、拓扑相关结构域(TAD)、染色质
环(loop)三个层级的信息。作为表观遗传学科研服务的国内先驱,表观生物可为客户提供 Hi-C 技术与表观多组学研究
手段联合分析的整体研究思路,从三维表观遗传组学层面深入研究疾病发生发展的机制。
技术应用
1. 研究染色质片段之间的相互作用
2. 建立基因组三维折叠模型
3. 适用于基因组组装
4. 与 RNA-seq 联合分析,研究染色质三维构象对RNA 表达的调控
5. 与 ChIP-seq 联合分析,解析调控元件、转录因子与染色质相互作用的关系
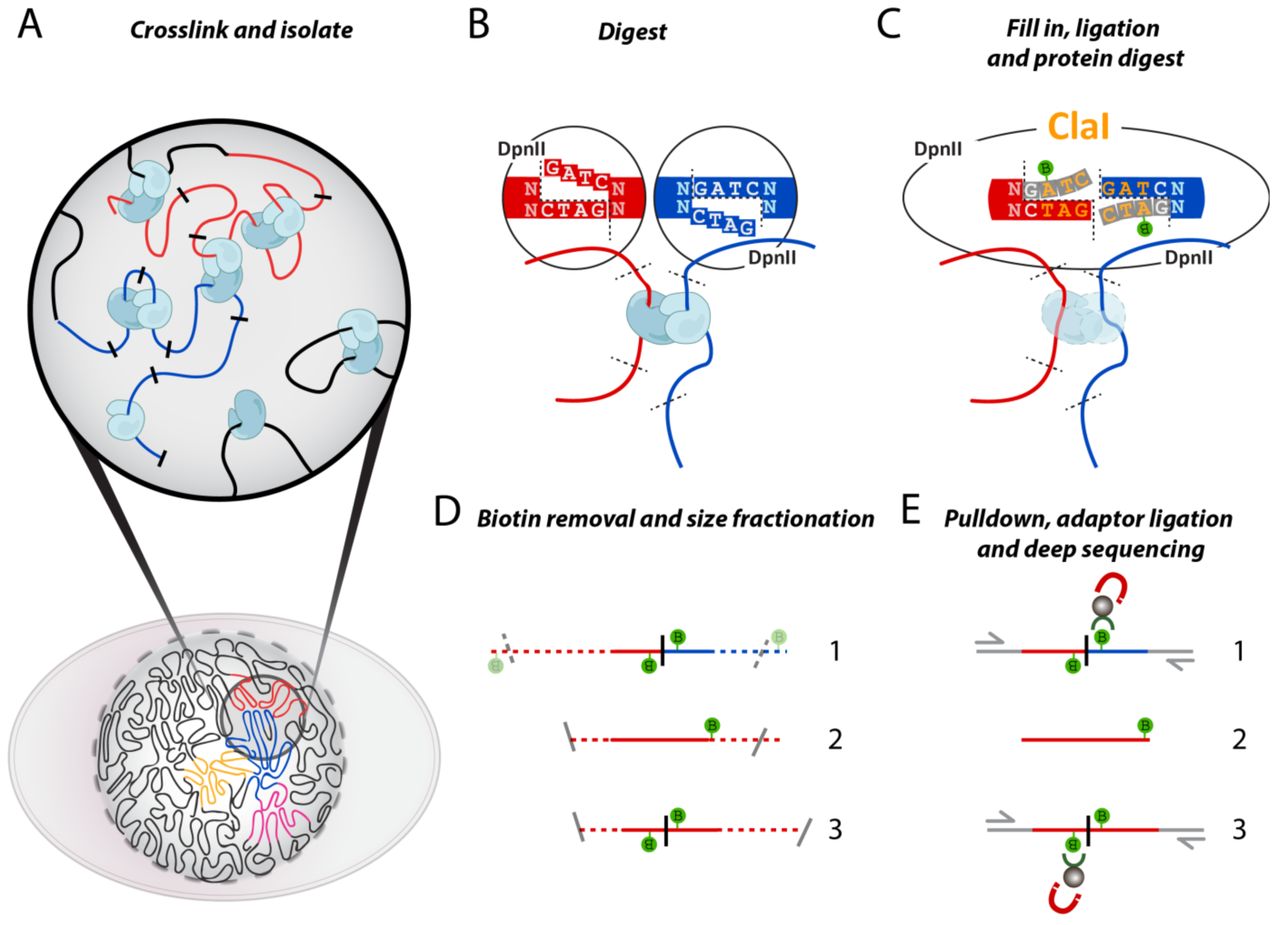
图 1-3. Hi-C 技术原理
技术原理
A, 甲醛固定细胞;B. DpnII 消化 DNA,识别 GATC,生成 5'-GATC 粘性末端;C. 用 dNTPs 和生物素 -14-dATP 填平 5'
粘性末端,成为平端。连接平端,生成一个新的限制性酶切位点(Clal),可用于评估填平效率。连接之后,交联逆转,
去除 DNA 中的蛋白质;D. 去除未连接端的生物素。DNA 被切成 200~300bp 的片段,适用于双端测序;E. 用链霉亲和素
磁珠富集连接产物,再加接头。
送样要求
样本类型:
1. 细胞:≥ 5×10^6 个细胞/样本;2. 组织:≥ 100mg/ 样本
需要先用甲醛交联固定
分析内容
1. 数据过滤
1.1 原始数据
1.2 数据过滤
1.3 测序数据质量分布
1.4 测序数据碱基分布
2. 基因组比对分析
3. 比对到酶切片段
4. 顺反相互作用比例
5. 染色体相互作用
5.1 染色体间相互作用
5.2 染色体内的相互作用
6. 结构分析
6.1 A/B compartments
6.2 TAD calling
6.3 Loop 鉴定及差异分析
6.4 三维建模
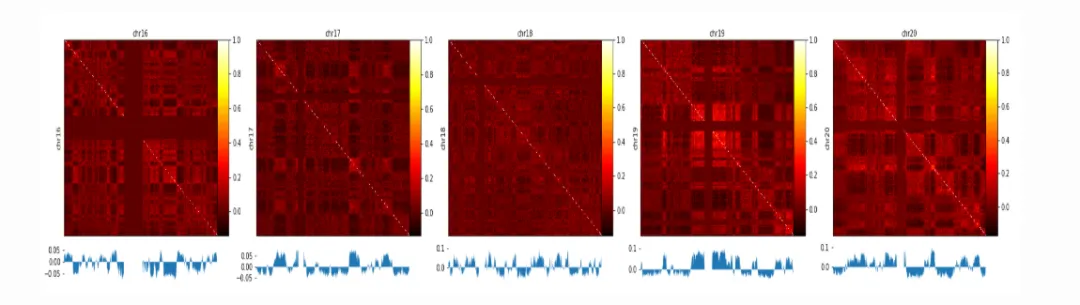
图1. A/B compartment分析
分析示例

图2. TAD鉴定
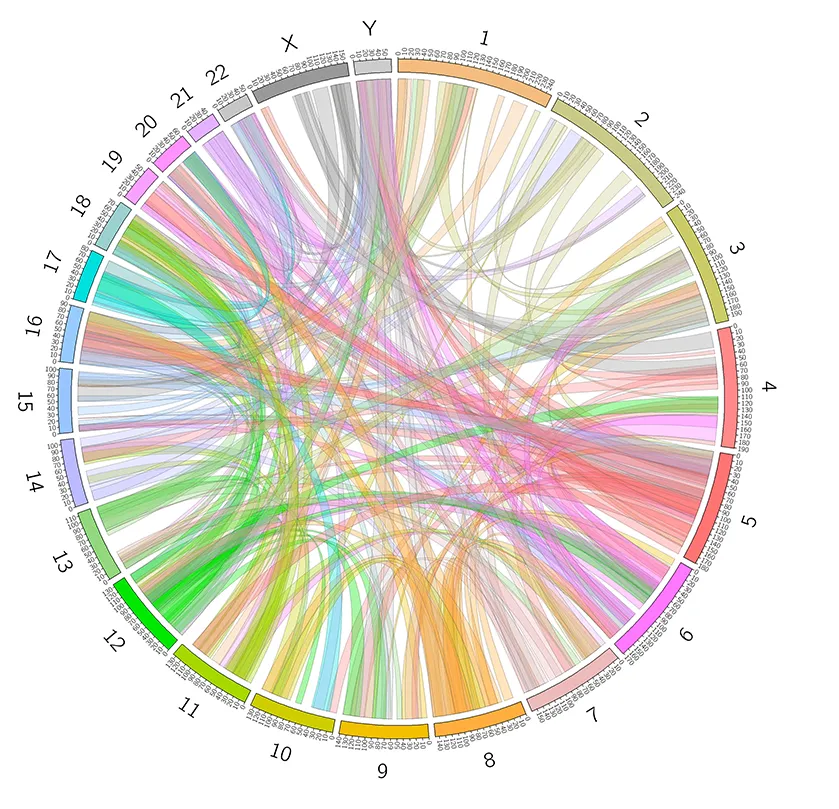
图3. Loop鉴定
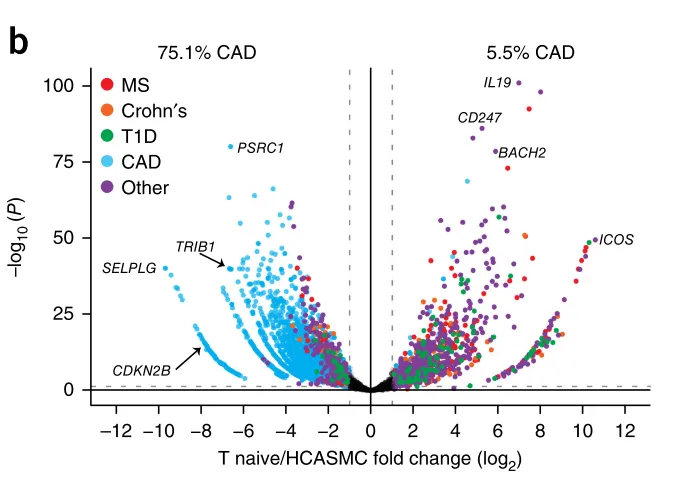
图4. Loop差异分析
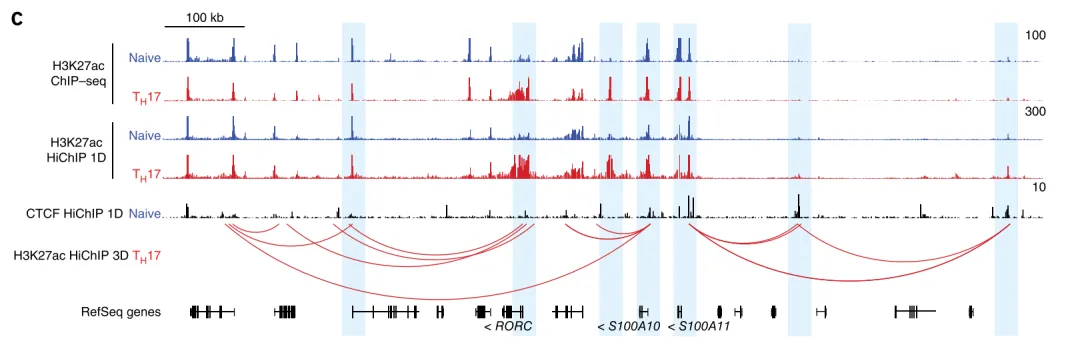
图5. SNP与Loop关联分析(感兴趣基因绘图)
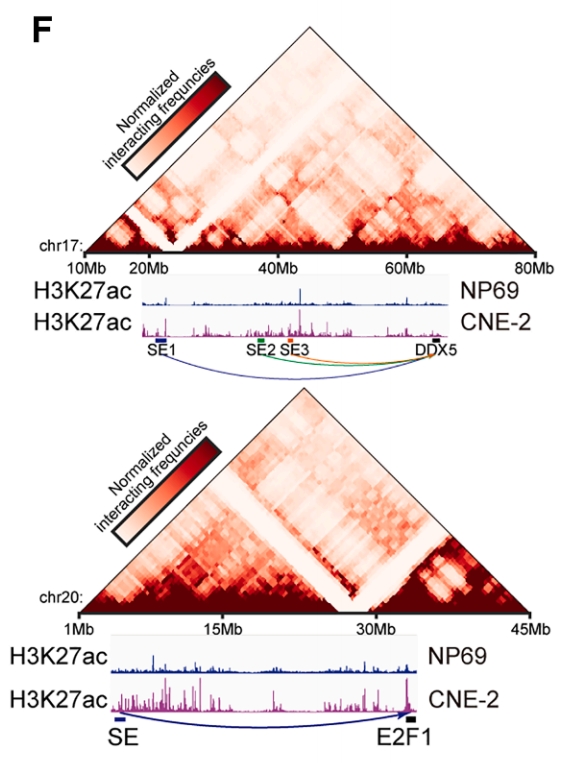
图9. 通过Hi-C分析鉴定的CNE2细胞中SE区域与DDX5和E2F1的TAD区域之间的长程环状相互作用,以及CNE2和NP69的H3K27ac ChIP信号
客户文章
Cell Rep Med: DDX5超级增强子通过促进ADAM10转录激活血管生成拟态形成和鼻咽癌转移的机制研究(DOI: 10.1016/j.xcrm.2025.102146)
该研究揭示了抗血管生成药物(AADs)耐药的重要机制——代偿性血管生成拟态(VM)形成。研究团队采用DIA质谱蛋白质组学技术筛选出ADAM10作为促进VM形成的关键蛋白,发现其在转移性鼻咽癌患者外泌体中显著富集。通过CUT&Tag测序分析H3K27ac修饰识别超级增强子,结合Hi-C技术解析三维基因组结构,发现DDX5和E2F1两个转录因子通过远程环状相互作用被超级增强子激活,进而上调ADAM10表达。体内外实验证实,IM联合尼妥珠单抗能够同时抑制血管生成和VM形成,有效克服AADs耐药性,抑制鼻咽癌转移并改善预后。该研究为理解抗血管生成治疗耐药机制提供了新的表观遗传学视角,并提出了潜在的联合治疗策略。(CUT&Tag和Hi-C由表观生物提供)
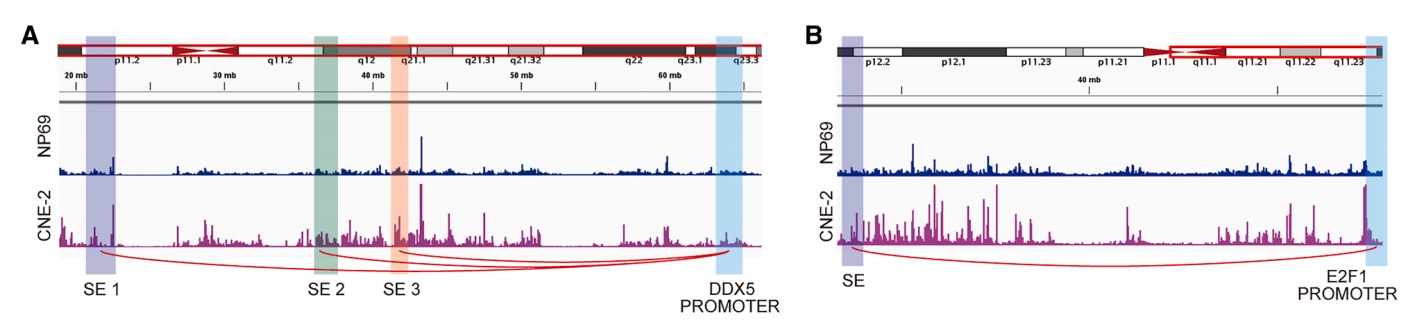
图10. 染色质长程环状相互作用在SE区域与DDX5 (A)和E2F1 (B)启动子之间的相互作用
