rPRO-seq全新首发,新生RNA测序新标杆!流程加速,更低样本量
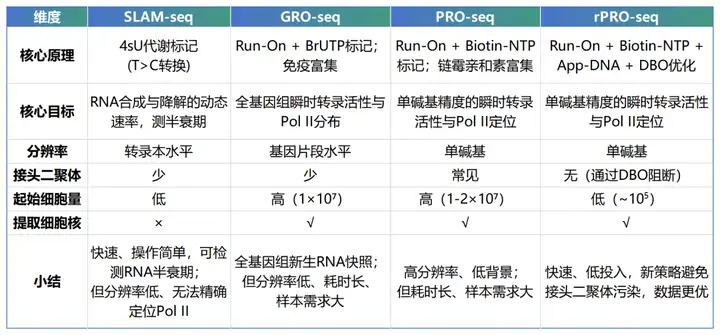
转录调控是基因表达的核心环节,传统RNA-seq技术往往只能检测稳态的RNA,而难以捕捉那些半衰期极短、不稳定或低丰度的转录本(如eRNA、lncRNA)。为了精准解析启动子近端RNA聚合酶II的暂停与释放、转录起始与延伸的精细调控,以及增强子转录活动等关键生物学过程,先后诞生了GRO-seq、SLAM-seq和PRO-seq等技术,经历一场从“稳态”到“动态”的深刻变革。其中PRO-seq可实现单碱基分辨率,但存在耗时耗力、样本需求高等局限。
新生RNA(nascent RNA)是指正在被RNA聚合酶转录、尚未完成加工的RNA分子。它直接反映基因在特定时间点的实时转录活性,不受RNA剪接、稳定性或降解等转录后过程的影响。

rPRO-seq(Rapid Precision nuclear run-on sequencing)是PRO-seq的全新升级版本,在保持单碱基分辨率优势的同时,通过独特的接头设计和流程优化,极大地缩短了建库时间(~15 h),实现更低起始细胞量,解决了接头二聚体问题,显著提升建库效率和数据质量。适用于临床样本、稀有细胞类型。
技术对比
技术流程
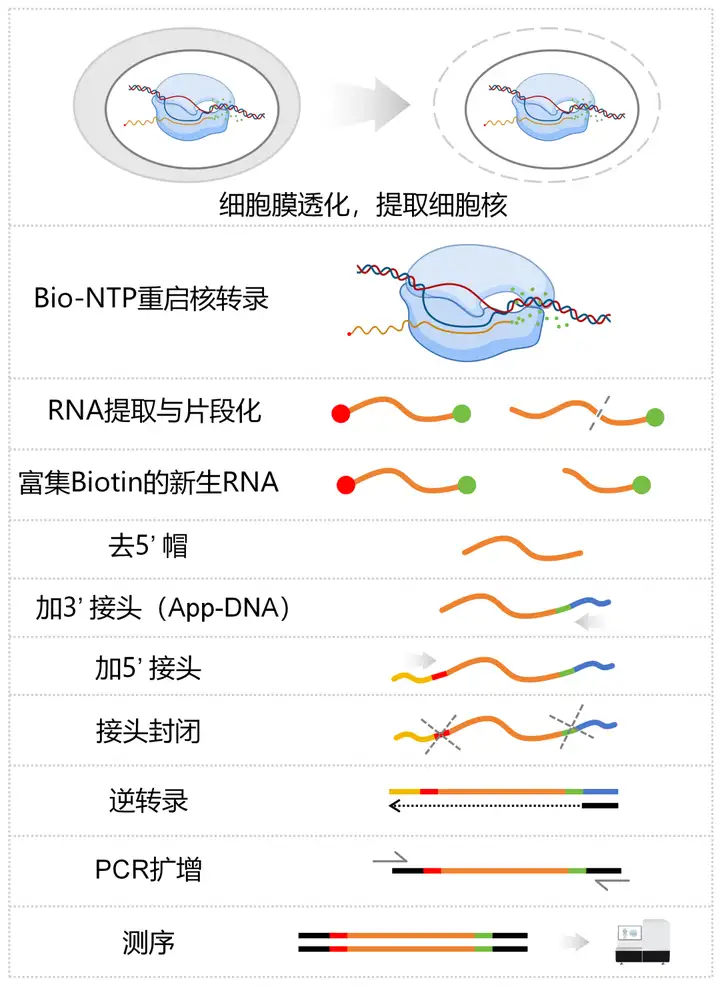
为什么选择rPRO-seq?
相较于传统的PRO-seq,rPRO-seq:
📉实验周期大幅缩短
建库时间从4-5 d压缩至~15 h;加速项目周期,降低RNA降解风险;
📉起始细胞量大幅降低
最低仅需~10⁵个细胞(传统需1-2×10⁷),显著拓展了技术适用范围;
📈数据有效率大幅提升
App-DNA+DBO策略提高连接效率、避免接头二聚体生成,数据质量显著提升。
应用场景
1.转录调控机制研究
-
启动子近端暂停分析:单碱基精度定位RNA Pol II暂停位点,解析暂停释放调控机制;
-
增强子功能研究:高灵敏度检测eRNA,绘制增强子-启动子调控网络。
2. 表观遗传调控研究
-
染色质状态与转录偶联:结合ChIP-seq、CUT&Tag、ATAC-seq等数据,解析转录因子结合、组蛋白修饰或染色质开放程度与转录活性之间的关联;
-
稳态RNA水平:对比新生RNA(生产速率)与稳态RNA(库存量),计算RNA的降解速率或合成效率,揭示转录后调控机制;
-
三维基因组:联合HiChIP、Hi-C数据,将远端增强子(Enhancer)的转录信号与目标基因(Promoter)的转录信号通过物理接触关联起来,解析远距离调控机制;
-
非编码RNA功能:检测上游反义转录、增强子RNA、长链非编码RNA等不稳定转录本。
3. 疾病机制与药物研发
-
肿瘤转录依赖性评估:快速评估如BET抑制剂等表观药物的直接转录靶点;
-
药物响应动力学:区分药物的直接转录效应与继发性适应反应;
-
神经发育障碍研究:如研究揭示关键基因在神经元中调控神经发育相关基因的关键作用。
4. 发育与干细胞生物学
深入剖析胚胎干细胞、组织干细胞或稀有祖细胞(如造血祖细胞)在分化、自我更新过程中的瞬时转录重编程机制。
送样要求
样本类型:细胞,≥ 1×10⁵个细胞/样本;活率>85%
样本物种:仅限人、大小鼠,其他物种需评估
更多信息请咨询客服或当地销售
数据分析
分析内容:
| 标准分析 |
|
1. 去接头、低质量reads 2. Reads基因组比对 3. 转录活跃区域识别 4. 启动子和基因体活性区域分类 5. 各区域read counts计算统计 6. Reads在转录起始位点(TSS)附近的meta分析 7. Traveling Ratio分析:计算转录延伸效率(启动子/基因体信号比) 8. Traveling Matrix分析:二维分类转录状态变化(四象限分析) 9. 差异表达分析10. GO/KEGG通路富集分析 |
| 高级分析 |
| eRNA分析 |
| 个性化分析(评估收费) |
分析结果示例(完整详见Demo报告):
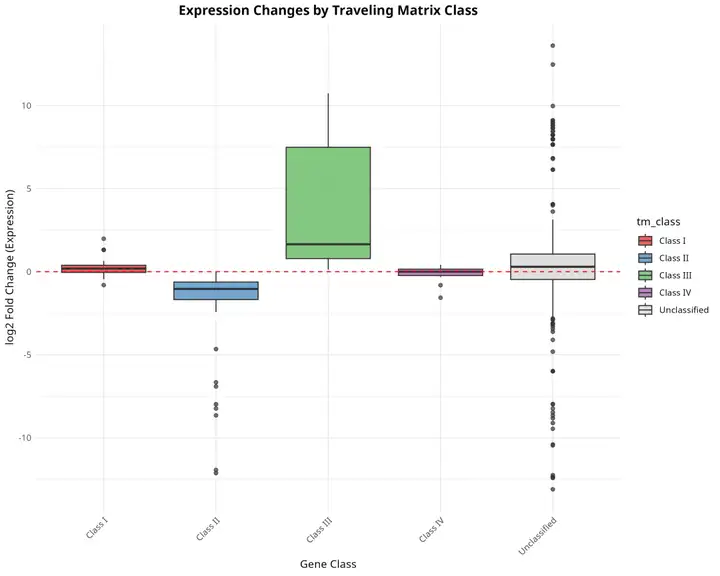
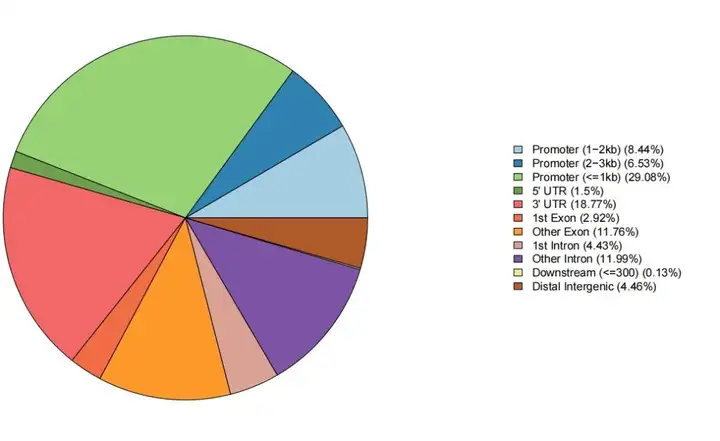
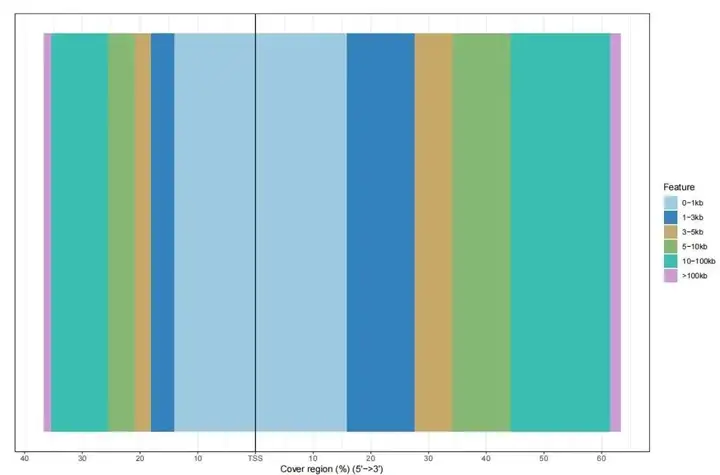
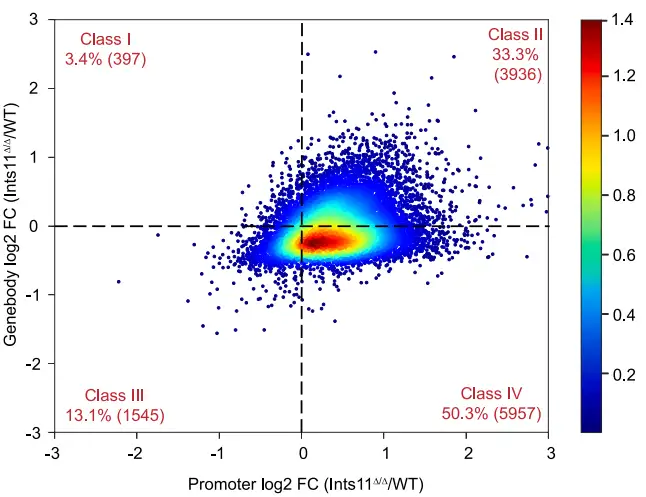
Class I: 转录暂停(启动子信号增加,基因体减少);Class II: 转录抑制(启动子和基因体信号都减少);Class III: 转录激活(启动子和基因体信号都增加);Class IV: 转录暂停释放(启动子信号减少,基因体增加)
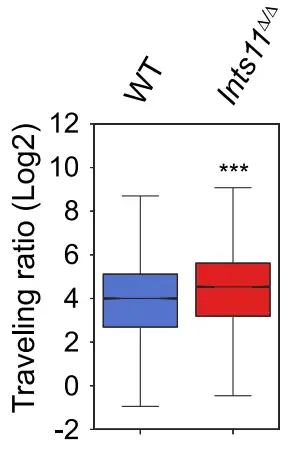
相关案例
案例一:

标题:Enhancing transcriptome mapping with rapid PRO-seq profiling of nascent RNA(1)
期刊:Molecular Cell(16.6)
发表时间:2025.08
研究使用Ints11敲除小鼠模型,从50万个稀有造血祖细胞进行rPRO-seq分析。INTS11缺失导致3525个基因差异表达,下调基因富集于信号通路和mRNA加工。转录矩阵(TM)显示50%基因启动子聚合酶密度增加而基因体降低,反映暂停增强和延伸受损;90%增强子RNA转录延长,证实Integrator作为启动子近端终止复合物的功能。
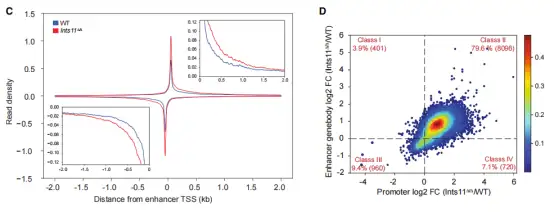
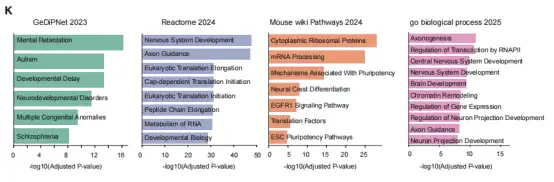
此外,将小鼠胚胎干细胞分化为神经元,通过INTS11-dTAG系统急性降解INTS11后进行rPRO-seq。结果显示神经元基因尤其是长基因(>20kb)出现聚合酶暂停和延伸缺陷。下调基因富集于神经发育障碍通路(神经元分化、突触组织、轴突发育等),直接靶基因包括自闭症风险基因SHANK3、突触相关基因NLGN/NRXN和谷氨酸受体GRIN2A/2B等,在转录层面阐明INTS11突变致病机制。
案例二:

标题:Glucocorticoid receptor suppresses GATA6-mediated RNA polymerase II pause release to modulate classical subtype identity in pancreatic cancer(2)
期刊:GUT(25.8)
发表时间:2025.06
测序方法:RNA-seq、ATAC-seq、ChIP-seq、PRO-seq、leChRO-seq、HiChIP、scFFPE-seq;公共数据(scRNA-seq、RNA-seq)
多组学整合研究思路:
该研究首先整合患者scRNA-seq、PDX和细胞系bulk RNA-seq鉴定经典亚型标志物CDH17/LGALS4,并通过多重免疫荧光和scFFPE-seq在患者组织验证;随后利用荧光报告系统进行药物筛选发现GR激动剂,结合mRNA-seq、ATAC-seq和H3K27ac ChIP-seq确认其抑制经典亚型的转录组和表观效应,并通过生物信息学预测和siRNA筛选锁定GATA6为关键调控因子;进而通过多层次ChIP-seq(GATA6、GR、HNF4A、Pol II total/pSer2/pSer5)定位基因组结合位点,PRO-seq揭示GATA6通过促进Pol II暂停释放(而非招募)调控经典亚型转录的独特机制,HiChIP解析增强子-启动子三维互作关系;最后用dCas9-KRAB在GATA6结合的增强子处人工诱导暂停,通过leChRO-seq验证增强子暂停导致靶基因启动子暂停增加和表达下降的因果关系;同时对比ΔNp63在基底型中通过促进Pol II招募和稳定E-P互作的不同调控模式,形成从患者发现到分子机制、从观察性研究到干预性验证、从二维基因组到三维结构的完整研究闭环。
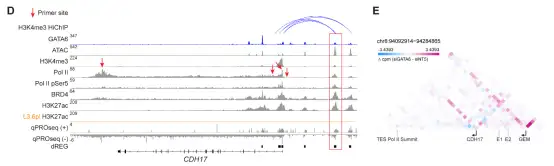
核心逻辑:
表观基因组学(染色质可及性+组蛋白修饰)→ 转录组学(稳态mRNA-seq)→ 转录动态(PRO-seq发现暂停释放机制) → 三维基因组学(HiChIP确认E-P互作)→ 因果验证(dCas9-KRAB + leChRO-seq证明增强子-启动子功能联系)
案例三:

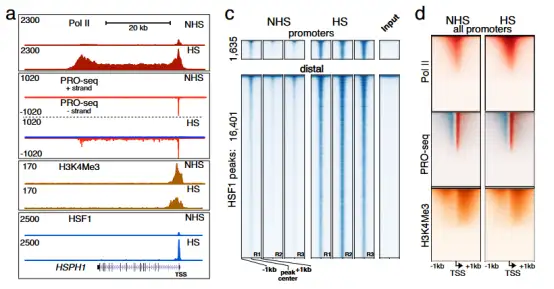
标题:Transcriptional responses of cancer cells to heat shock-inducing stimuli involve amplification of robust HSF1 binding(3)
期刊:NC
发表时间:2023.11
测序方法:ChIP-seq、PRO-seq、ATAC-seq
多组学整合研究思路:
研究通过两种不同刺激(热休克和砷处理)诱导两个远缘癌细胞系(MCF-7和K562)的热休克反应,利用ChIP-seq追踪HSF1结合位点的动态变化,结合PRO-seq实时监测新生RNA转录活性,并用ATAC-seq评估染色质可及性变化。通过跨条件、跨细胞系的比较分析,研究发现:HSF1的全基因组结合模式主要由细胞类型决定而非刺激类型,其结合位点在基础状态下已预先建立,激活过程是信号强度依赖的均匀放大而非位点重新分布;HSF1结合与转录激活在全基因组水平存在广泛解偶联,共同的HSF1结合可导致不同的转录结果,提示转录因子组合调控的复杂性;HSF1优先结合已开放的染色质区域,但在远端非功能元件处可诱导适度的染色质开放,提示其在表观遗传重塑中的潜在作用。
案例四:

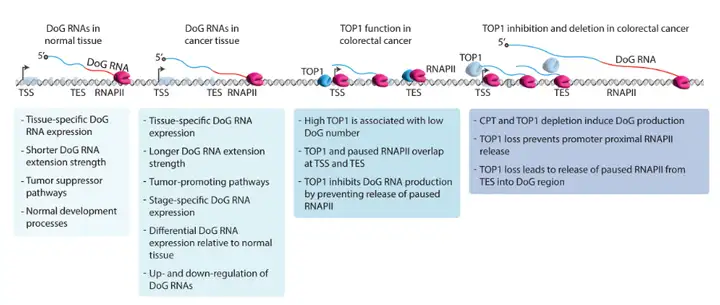
标题:Downstream-of-gene (DoG) transcripts contribute to an imbalance in the cancer cell transcriptome(4)
期刊:SCIENCE ADVANCES(12.5)
发表时间:2024.07
测序方法:RNA-seq、PRO-seq、ChIP-seq、TOP1-seq
研究通过分析TCGA公共数据库中三种主要癌症(乳腺癌、结肠癌、肝癌)的RNA-seq数据,系统鉴定了肿瘤特异性DoG RNA图谱,发现其表达失调与患者预后显著相关,并与肿瘤促进/抑制通路功能富集相关联;
结合RNA-seq、ChIP-seq(RNAPII和TOP1)及PRO-seq数据,在结肠癌细胞模型中解析了TOP1通过调控RNA聚合酶II在TSS和TES的暂停/释放动态来抑制DoG RNA产生的分子机制;
接着通过TOP1-seq技术区分催化活性TOP1与总TOP1的基因组分布差异,结合药物干预(CPT处理)和基因敲低实验,证实TOP1的催化活性依赖和非依赖功能在DoG RNA调控中的双重作用,最终将DoG RNA失调与结肠癌治疗靶点(TOP1抑制剂CPT的临床疗效)建立因果联系。该策略从癌症转录组异常特征的临床关联到转录调控机制的分子解析,为理解非编码RNA在肿瘤发生中的作用提供了证据。
1、Cingaram PKR, Beckedorff F, Yue J, et al. Enhancing transcriptome mapping with rapid PRO-seq profiling of nascent RNA. Mol Cell. 2025;85(15):2988-3001.e4.
2、Ekstrom TL, Rosok RM, Abdelrahman AM, et al. Glucocorticoid receptor suppresses GATA6-mediated RNA polymerase II pause release to modulate classical subtype identity in pancreatic cancer. Gut. 2025;74(7):1112-1124. Published 2025 Jun 6.
3、Dastidar SG, De Kumar B, Lauckner B, et al. Transcriptional responses of cancer cells to heat shock-inducing stimuli involve amplification of robust HSF1 binding. Nat Commun. 2023;14(1):7420. Published 2023 Nov 16.
4、Abe K, Maunze B, Lopez PA, et al. Downstream-of-gene (DoG) transcripts contribute to an imbalance in the cancer cell transcriptome. Sci Adv. 2024;10(27):eadh9613.
- - - 推荐阅读 - - -

RIC-seq: RNA与RNA相互作用
2026-01-15
PAR-CLIP-seq:描绘RNA-蛋白质相互作用图谱
PAR-CLIP-seq(Photoactivatable Ribonucleoside-Enhanced Crosslinking and lmmunoprecipitation sequencing)即基于光活化核糖核苷的交联和免疫沉淀测序技术[1],将具有光活性的核糖核苷类似物4sU掺入到新转录的RNA中,再通过分析碱基T-C转换位点,可在全转录组范围内研究RNA与蛋白的相互作用,揭示靶向RBP的RNA结合位点。
2026-01-15

RADICL-seq
RADICL-seq(RNA And DNA Interacting Complexes Ligated and sequenced)是一种探索 RNA 与染色质相互作用 的新技术,绘制细胞核内 RNA 与染色质的相互作用图谱,能够鉴定不同类转录本的基因组覆盖模式,以及细胞特异性的 RNA- 染色质相互作用。利用 RADICL-seq 可以在 RNA 与基因组靶标区域相互作用时捕获 RNA,并对捕获的每个 RNA 进行 全面定位。
2026-01-15
