SELECT-m6A单位点修饰定量检测试剂盒

项目简介
SELECT(single-base elongation-and ligation-based qPCR amplification method)[1]检测技术,即单碱基延长和连接的qPCR扩增技术,研发团队为北京大学贾桂芳课题组,只需三小时就能完成低丰度转录本中单碱基分辨率的m6A检测,无需抗体富集就能够针对性地对特定位点进行 m6A修饰定量,样本间修饰丰度差异分析以及鉴定目标位点是否存在m6A修饰。表观生物与北京大学签订SELECT-m6A修饰定量检测技术转让(专利实施许可)协议,获得全球范围内实施SELECT-m6A修饰定量检测技术成果转化的授权,为客户提供多款SELECT检测试剂盒,填补市场上特定位点m6A 常规检测技术的空白,协助研究者鉴定m6A相关蛋白的作用靶点,深入挖掘m6A修饰的作用机制,有望为RNA m6A修饰分子标志物的发现带来新突破。
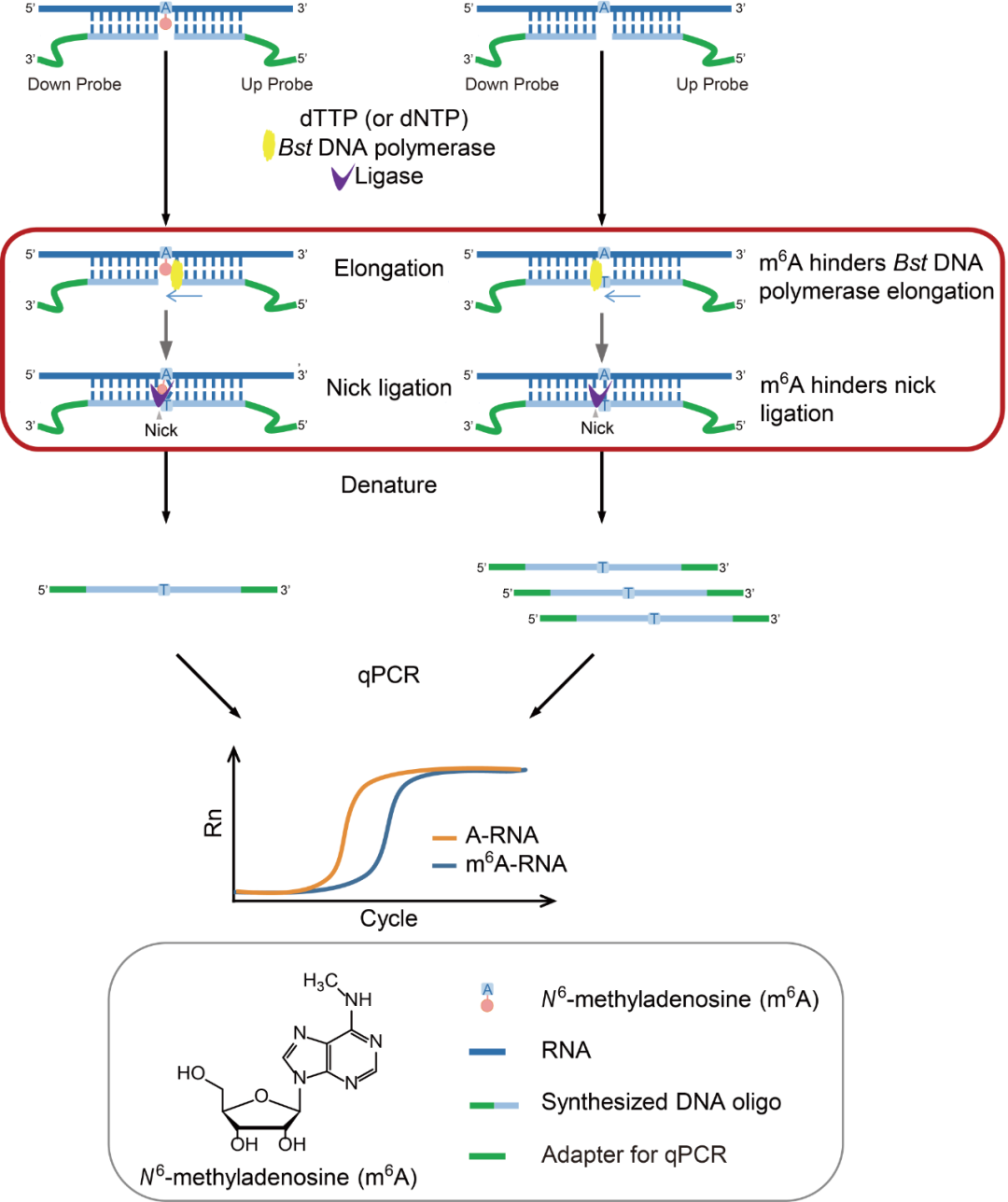
技术原理
技术应用
1. 验证样本间特定位点m6A修饰丰度差异
2. 检测RNA目标位点是否存在m6A修饰
3. 对特定位点的m6A修饰丰度进行绝对定量
4. 鉴定m6A修饰相关蛋白的作用靶点
技术优势
1. 荧光定量PCR进行精确定量,达到单碱基分辨率
2. 无需抗体IP,无需同位素标记,降低成本,可操作性高
3. 灵敏度高,总RNA所需样本低至1μg
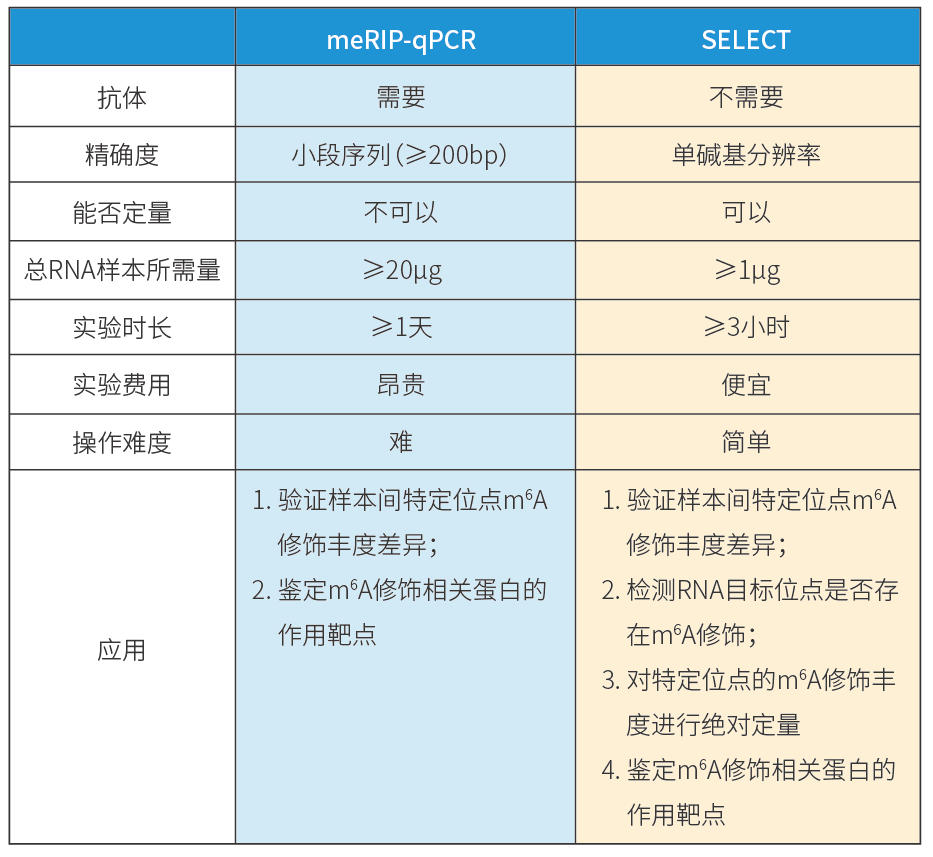
技术对比
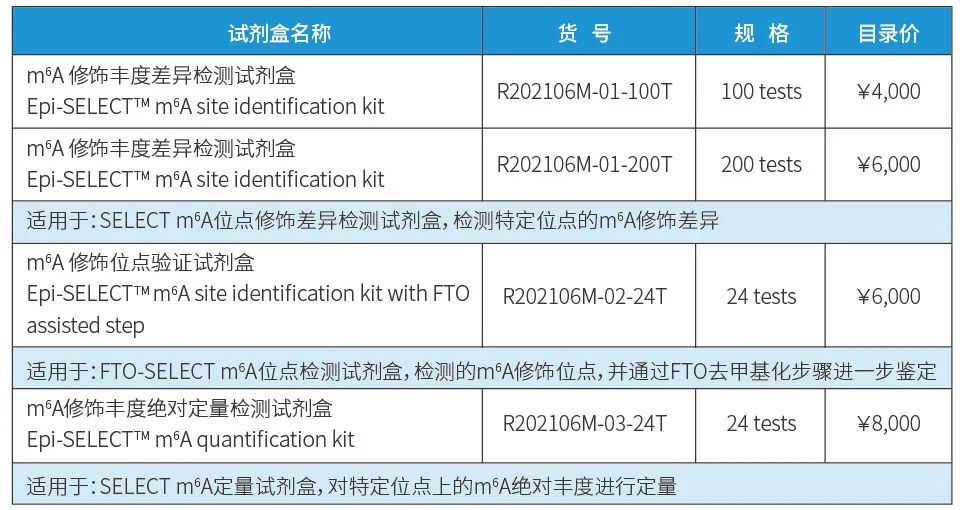
试剂盒信息
应用案例
1. 检测RNA目标位点是否存在m6A修饰;
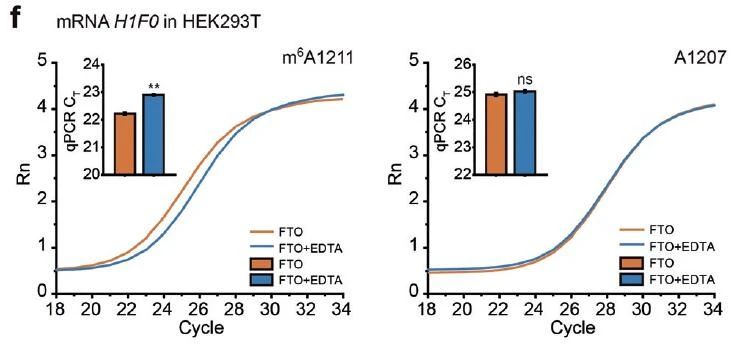
图1. qPCR扩增曲线和CT值柱形图显示,HeLa细胞lncRNA MALAT1 (e)以及HEK293T细胞mRNA H1F0(f)的m6A位点和非m6A位点[1]
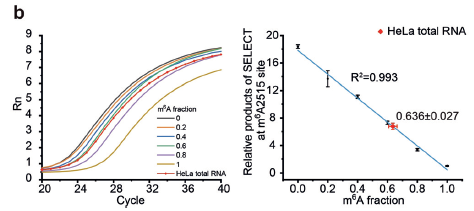
图2. 利用系列浓度梯度的已知m6A成分标准品制作标准曲线,然后对特定位点上的m6A修饰成分进行定量[1]
2. m6A对特定位点的m6A修饰丰度进行绝对定量
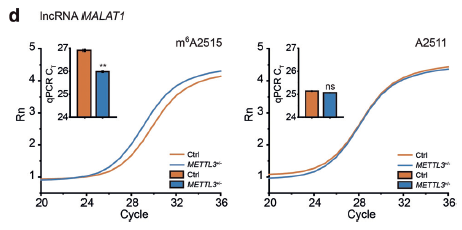
图2. 利用系列浓度梯度的已知m6A成分标准品制作标准曲线,然后对特定位点上的m6A修饰成分进行定量[1]
3. 鉴定m6A修饰相关蛋白(甲基化酶、去甲基化酶、识别蛋白等)的作用靶点
图3. SELECT分析 lncRNA MALAT1和以及METTL3+/-细胞(对照组)的m6A2515和A2511(input对照)位点,荧光扩增曲线和qPCR CT值柱状图显示lncRNA MALAT1的2515位点是METTL3的一个靶点[1]
客户文章
1. Adv Sci:CRISPR/dCas13(Rx)介导的RNA m6A动态编辑系统增强棉花抗旱性研究[2]
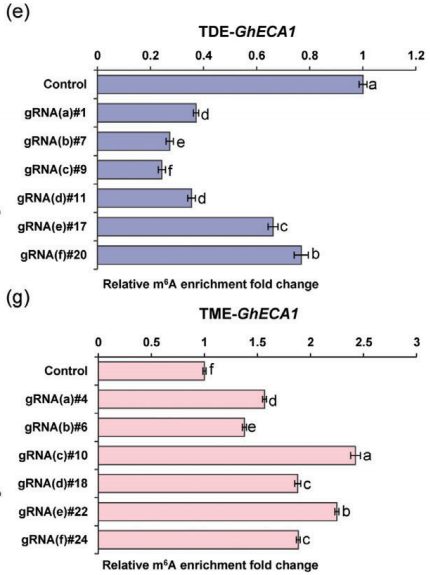
这项研究开发了基于CRISPR/dCas13(Rx)的可编程RNA m6A编辑工具(TME和TDE),通过融合甲基转移酶GhMTA或去甲基酶GhALKBH10,实现对植物内源转录本GhECA1和GhDi19特定m6A位点的动态修饰。
研究中使用SELECT试剂盒验证GhECA1转录本3' UTR区域和GhDi19转录本5' UTR区域的m6A修饰效率:TDE编辑器使GhECA1的m6A水平降低24%-76%,并导致其mRNA丰度下降;而TME编辑器使GhECA1的m6A富集增加1.37-2.51倍,并上调其表达。同时,在GhDi19的5' UTR区域,TDE显著降低m6A水平(32%-76%)并促进转录,而TME提高m6A水平(1.43-2.52倍)抑制表达。
图4. 3e和3g分别展示了TDE(去甲基化编辑器)和TME(甲基化编辑器)对GhECA1转录本3' UTR区域m6A水平的调控效果
![2. Acta Pharmacol Sin:METTL14介导pri-miR-5099的m6A甲基化驱动心肌梗死中心肌细胞焦亡的分子机制[3]](http://openapi.whaleng.com/file-server/ep/2025/06/743ce975f4184f09aed533a74d819ab4.png)
2. Acta Pharmacol Sin:METTL14介导pri-miR-5099的m6A甲基化驱动心肌梗死中心肌细胞焦亡的分子机制[3]
这篇文章揭示了METTL14介导的m6A甲基化通过调控pri-miR-5099加工促进心肌细胞焦亡的分子机制。研究应用SELECT试剂盒对pri-miR-5099的m6A甲基化位点进行了特异性检测。通过FTO去甲基化酶处理和EDTA对照实验,比较两组样本的qPCR循环阈值差异,证实了pri-miR-5099序列中存在特异性m6A修饰位点。
该检测为后续机制研究提供了关键证据,证明METTL14介导的m6A甲基化增强了DGCR8对pri-miR-5099的识别,从而促进其加工为成熟miR-5099-3p。这一发现将RNA表观遗传修饰与心肌梗死病理过程联系起来,揭示了METTL14/miR-5099-3p/ELF1调控轴在心肌细胞焦亡中的核心作用,为心肌保护提供了新的治疗靶点。
图5. 通过表观生物SELECT试剂盒(Epi-SELECT m6A Site Identification Kit with FTO-assisted step)验证pri-miR-5099序列中是否存在特异性m6A甲基化位点
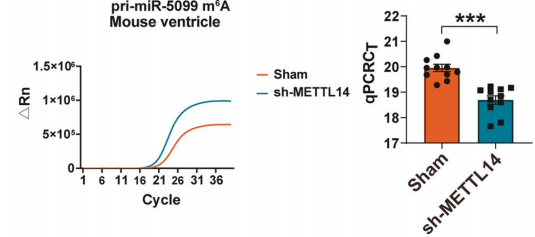
图6. 在敲低METTL14的小鼠心肌组织中,利用SELECT技术检测pri-miR-5099的m6A甲基化水平
![3. Nat Commun:基于RNA m6A修饰的吸收电离辐射剂量估算biomarker[4]](http://openapi.whaleng.com/file-server/ep/2025/06/293d0ed75d4e42df91f7d3b8b4aebc8e.png)
3. Nat Commun:基于RNA m6A修饰的吸收电离辐射剂量估算biomarker[4]
这项研究首次发现了基于RNA m6A修饰的新型辐射暴露剂量biomaker,并建立了基于该修饰的人体辐射暴露剂量预测模型。
为了证实Ncoa4 m6A水平对IR的响应曲线,研究者使用了单碱基分辨率、不依赖于抗体的m6A检测方法SELECT。结果显示,四个靶位点中的三个(如图3)在照射后7天和14天都表现出显著升高的m6A水平,并且升高的范围与照射剂量明显相关。
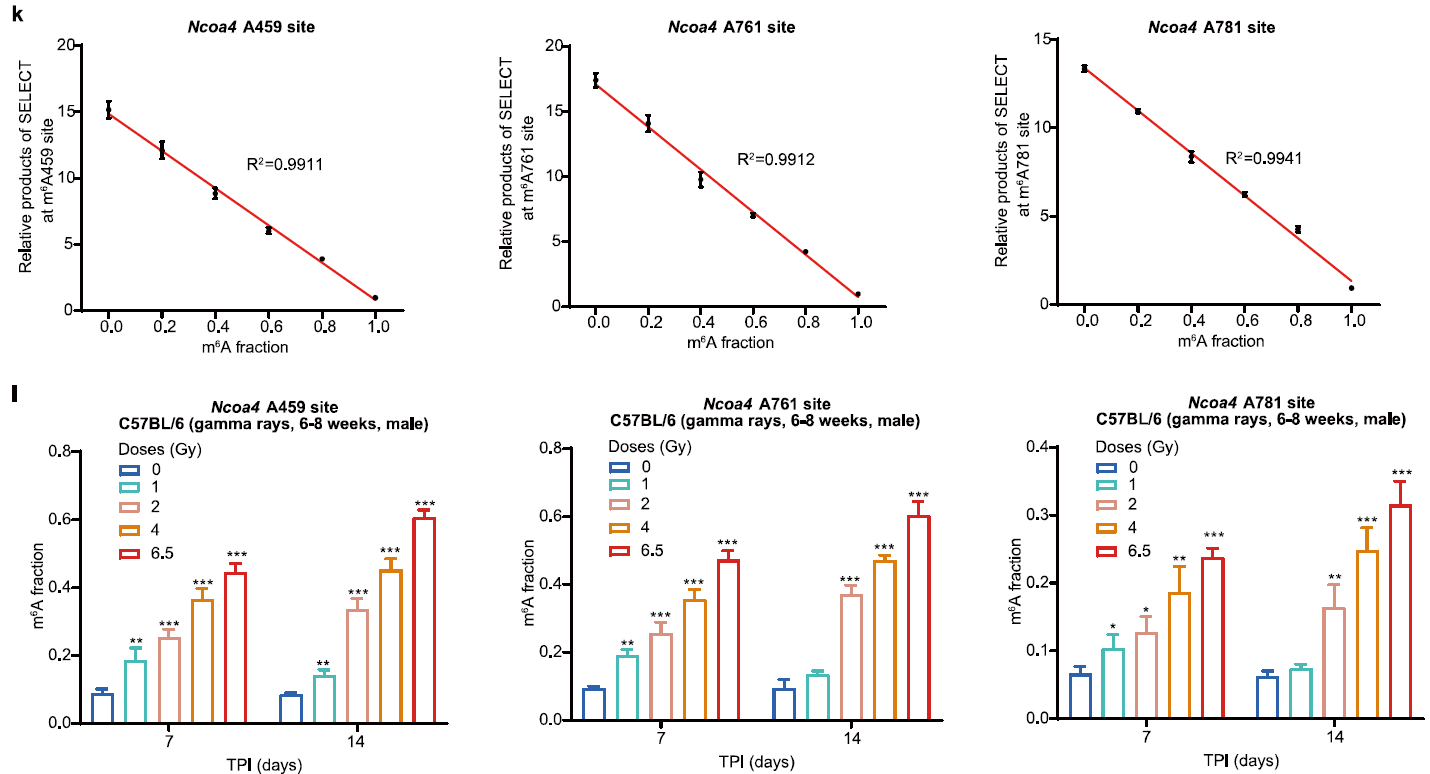
图7. 研究者使用表观生物提供的SELECT试剂盒来进一步评估m6A在Ncoa4转录本4个位点对IR的反应模式,结果显示其中三个在照射后7天和14天都显示出m6A水平显著升高,并且升高的范围与暴露剂量明显相关,与meRIP-qPCR结果一致
![4. Cell Rep:m6A 修饰的长链非编码RNA 调控神经元发育及其机制[5]](http://openapi.whaleng.com/file-server/ep/2025/06/eb37dbf010094c01a3e8b31d012e591f.png)
4. Cell Rep:m6A 修饰的长链非编码RNA 调控神经元发育及其机制[5]
这项研究揭示了长链非编码RNA (lncRNA) Dubr 的m6A修饰通过稳定YTHDF1/3 复合体以及介导mRNA 翻译调控神经元轴突生长和迁移及其机制。
图8. 研究者使用表观生物提供的SELECT-m6A 修饰单位点定量检测试剂盒,鉴定Dubr m6A甲基化位点,qPCR显示,FTO处理显著减少ND7/23细胞RNA裂解液的4 个Dubr位点的m6A修饰水平,ND7/23细胞RNA裂解液的4个Dubr位点,表明Dubr m6A修饰位点的特异性,所以说,Dubr保守区域的4个motif是它们的m6A修饰所必须的
参考文献与SELECT已发表文章(部分):
[1] Xiao Y, Wang Y, Tang Q, et al. An Elongation- and Ligation-Based qPCR Amplification Method for the Radiolabeling-Free Detection of Locus-Specific N6 -Methyladenosine Modification. Angew Chem Int Ed Engl. 2018 Dec 3;57(49):15995-16000.
[2] Yu L, Alariqi M, Li B, et al. CRISPR/dCas13(Rx) Derived RNA N6-methyladenosine (m6A) Dynamic Modification in Plant. Adv Sci (Weinh). 2024;11(39):e2401118. doi:10.1002/advs.202401118(SELECT 客户文章)
[3] Yu H, Li QS, Guo JN, et al. METTL14-mediated m6A methylation of pri-miR-5099 to facilitate cardiomyocyte pyroptosis in myocardial infarction. Acta Pharmacol Sin. Published online February 12, 2025. doi:10.1038/s41401-025-01485-y(SELECT 客户文章)
[4] Chen, H., Zhao, X., Yang, W. et al. RNA N6-methyladenosine modification-based biomarkers for absorbed ionizing radiation dose estimation. Nat Commun 14, 6912 (2023).(SELECT 客户文章)
[5] Huang J, Jiang B, Li GW, et al. m6A-modified lincRNA Dubr is required for neuronal development by stabilizing YTHDF1/3 and facilitating mRNA translation. Cell Rep. 2022 Nov 22;41(8):111693. (SELECT 客户文章)
[6] Zhang S, Lv C, Niu Y, et al. RBM3 suppresses stemness remodeling of prostate cancer in bone microenvironment by modulating N6-methyladenosine on CTNNB1 mRNA. Cell Death Dis. 2023 Feb 7;14(2):91.(SELECT 客户文章)
[7] Tan L, Qin Y, Xie R, et al. N6-methyladenosine-associated prognostic pseudogenes contribute to predicting immunotherapy benefits and therapeutic agents in head and neck squamous cell carcinoma. Theranostics 2022;12(17).(SELECT 客户文章)
[8] Chen X, Wang Y, Wang JN, et al. m6A modification of circSPECC1 suppresses RPE oxidative damage and maintains retinal homeostasis. Cell Rep. 2022 Nov 15;41(7):111671. (SELECT 客户文章)
[9] Sheng R, Meng W, Zhang Z, et al. METTL3 regulates cartilage development and homeostasis by affecting Lats1 mRNA stability in an m6A-YTHDF2-dependent manner. Cell Rep. 2024;43(8):114535. doi:10.1016/j.celrep.2024.114535(SELECT 客户文章)
[10] Zhou J, Tang J, Zhang C, et al. ALKBH5 targets ACSL4 mRNA stability to modulate ferroptosis in hyperbilirubinemia-induced brain damage. Free Radic Biol Med. 2024;220:271-287. doi:10.1016/j.freeradbiomed.2024.05.014(SELECT 客户文章)
[11] Cai B, Ma M, Yuan R, et al. MYH1G-AS is a chromatin-associated lncRNA that regulates skeletal muscle development in chicken. Cell Mol Biol Lett. 2024 Jan 4;29(1):9. (SELECT 客户文章)
[12] Li L, Cheng H, Zhou Y, et al. METTL3 regulates autophagy of hypoxia-induced cardiomyocytes by targeting ATG7. Cell Death Discov. 2025;11(1):37. Published 2025 Feb 1. doi:10.1038/s41420-025-02320-3 (SELECT 客户文章)
[13] Liu K, Wang X, Wang J, et al. N 6-methyladenosine modifications stabilize phosphate starvation response-related mRNAs in plant adaptation to nutrient-deficient stress. Nat Commun. 2025;16(1):4093. Published 2025 May 1. doi:10.1038/s41467-025-59331-y(SELECT 客户文章)
