PA-Ψ-seq

项目简介
假尿嘧啶(Pseudouridine,简称 Ψ)是 RNA 上常见的修饰类型,在 rRNA、tRNA 等 ncRNA 上的生物学功能已得到
广泛研究与报道,但由于缺乏在 mRNA 上检测 Ψ 修饰的技术,我们对 mRNA 的 Ψ 修饰功能尚不清楚。
表观生物 PA-Ψ-seq[1](photo-crosslinking-assisted Ψ sequencing,光交联辅助 Ψ 修饰测序),基于抗体对 Ψ修饰进行精确定位,达到单核苷酸分辨率。
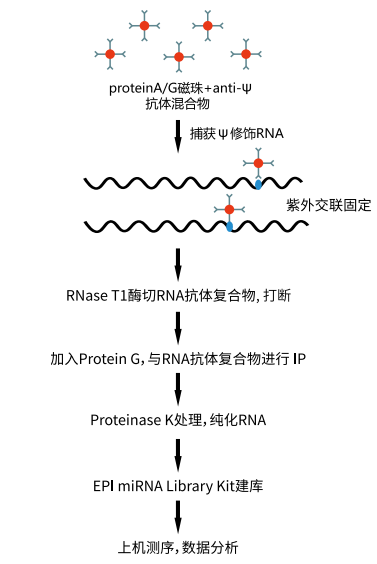
图 1. PA-Ψ-seq 技术流程
技术原理
将 RNA 片段化,利用假尿嘧啶抗体进行免疫沉淀实验。洗脱 RNA 片段,反转录成 cDNA,进行高通量测序。
应用与优势
技术应用
1. 绘制全转录组的 Ψ 修饰图谱
2. 研究 Ψ 修饰对各种生理过程的调控机制
3. 研究肿瘤等疾病的 Ψ 修饰异常表达
技术优势
1. 光辅助技术将残基交联至同源抗体,特异性高
2. 分辨率高,达到单碱基水平
2. 分析内容丰富,多年 RNA 修饰技术经验
送样要求
样本物种: 仅限人、大小鼠,其他物种需评估
≥ 300 μg/ 样本,RIN 值≥ 6
总RNA
≥ 300 μg/ 样本,RIN 值≥ 6
细胞
≥ 150 mg/ 样本
组织
分析内容
基本分析
1. 测序原始 reads 去接头 , 质量控制 (OC)
2. 参考基因组比对 (Mapping)
3. 富集区域鉴定 (PeakCalling)
4. 富集区域注释 (PeakAnno)
5. mRNA 修饰分析
6. 富集区域 metagene 图、pie 图、venny 图
7. Motif 分析
8. Peak 基因 GO 和 KEGG 分析
9. 差异 Peak 分析 ( 即差异 RNA 修饰 mRNA)
10. 差异 Peak 基因 GO 和 KEGG 分析
高级分析
1. 差异 RNA 修饰热图 (Heatmap)
2. 关联分析 ( 与 RNA-seq 关联分析或多组学关联分析 )
3. 关联分析热图 (Heatmap)
4. IGV 峰图 ( 附 5 个基因 )
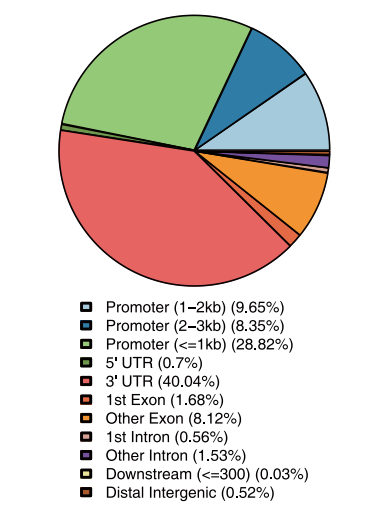
图 2. 注释类型饼图
表观生物实测数据
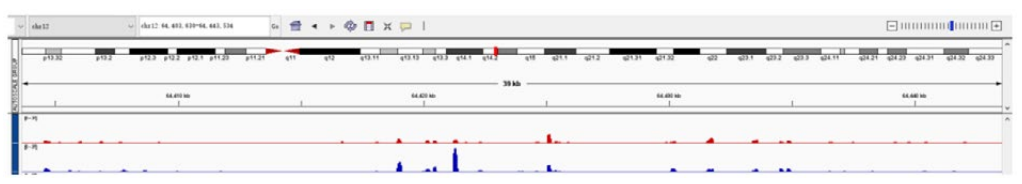
图 3. IGV 基因峰图
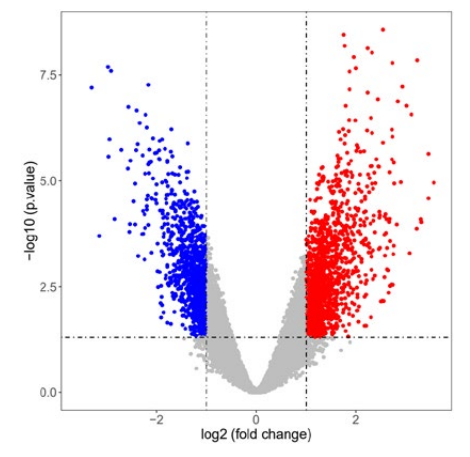
图 4. 差异 Peaks 火山图
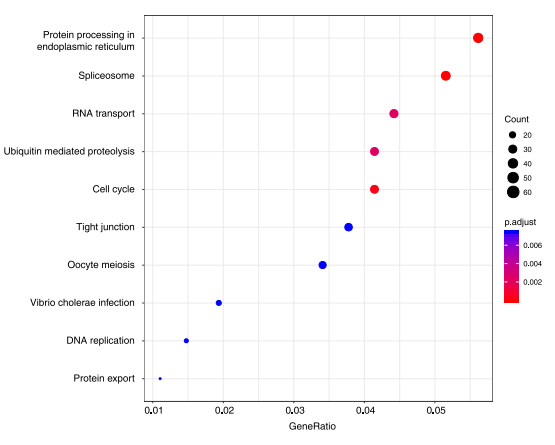
图 5. GO 差异 KEGG 分析气泡图
样本设置:
研究主要使用结直肠癌细胞系HCT116、DLD-1、HT-29和对照细胞HeLa,通过siRNA敲低PUS7或反义寡核苷酸耗竭7SK进行实验处理,每组设置2-3个生物学重复。同时分析了TCGA和GTEx数据库中的结直肠癌临床样本数据。
使用的测序方法:
采用假尿嘧啶测序(BID-seq)在单碱基分辨率检测核RNA的假尿苷修饰(鉴定363个Ψ位点);PAR-CLIP-seq捕获PUS7直接结合的RNA靶标(185个靶标);KAS-seq监测全基因组Pol II转录动态;mRNA-seq分析转录组变化(1000+差异基因);LC-MS/MS定量Ψ修饰水平。
关键结果:
研究发现PUS7催化7SK snRNA第250位尿苷的假尿苷化修饰(Ψ水平从72%降至48%),该修饰稳定7SK-P-TEFb复合物。PUS7缺失导致P-TEFb从7SK释放,Pol II Ser2磷酸化增加40%,促进转录延伸。转录组分析显示转录因子KLF6和凋亡基因DDIT3表达上调,介导细胞凋亡。功能实验证实PUS7敲低显著增强结直肠癌细胞对5-FU化疗的敏感性。利用dCas13b-PUS7系统特异性增加7SK Ψ250修饰可逆转上述表型,确证了因果关系。研究揭示PUS7-7SK轴是调控转录延伸的关键机制,为结直肠癌治疗提供了新靶点。
应用案例
Nat Commun: PU S7通过调控7SK假尿苷化调节RNA聚合酶II转录延伸并影响结直肠癌化疗敏感性 [2]
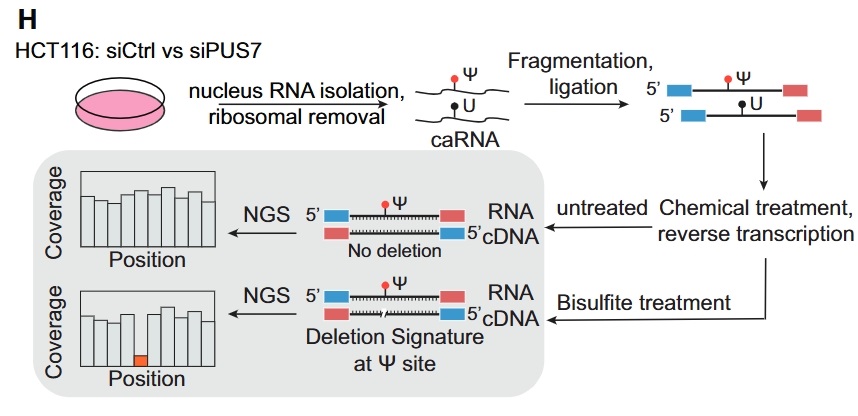
图 6. Ψ 测序 利用反转录过程中Ψ位点产生的缺失信号, 实现单碱基分辨率的全转录组Ψ位点检测和定量
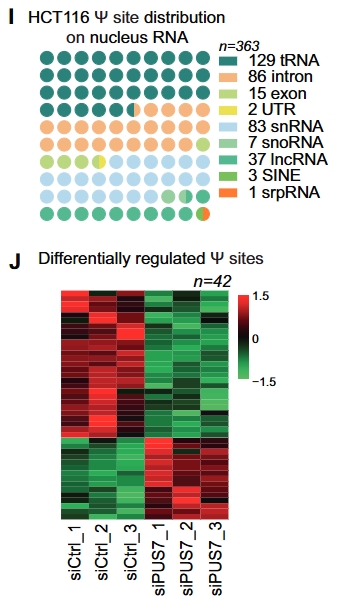
图 7. I)在HCT116细胞核RNA上共鉴定出363个Ψ位点;J)PUS7敲低后,42个位点的Ψ水平发生显著变化,其中25个位点Ψ水平下降
参考文献
[1] Martinez Campos C, Tsai K, Courtney DG, Bogerd HP, Holley CL, Cullen BR. Mapping of pseudouridine residues on cellular and viral transcripts using a novel antibody-based technique. RNA. 2021;27(11):1400-1411.
[2] Zhao Y, Sun HL, Li W, et al. Pseudouridylation of 7SK by PUS7 regulates Pol II transcription elongation. Nat Commun. 2025;16(1):9595. Published 2025 Oct
