NCB: 一种新的m6A阅读器!可促进mRNA翻译和稳定性
m6A是在真核细胞mRNA中最普遍存在的修饰,需由阅读器 (reader) 识别 (比如YTH结构域蛋白),调控mRNA的命运。近日,中山大学杨建华教授团队、辛辛那提大学陈建军教授以及芝加哥大学何川教授团队等合作,发现IGF2BP蛋白 (胰岛素样生长因子2 mRNA 结合蛋白) 是一种独特的m6A阅读器,它不像YTH结构域家族蛋白那样促进mRNA降解,而是使mRNA更稳定!咱们来看看他们是如何发现这种新reader的:
研究者通过两个方法,筛选m6A结合蛋白:1. 使用甲基化的单链RNA诱饵 (ss-m6A,共有序列为GG(m6A)CU),对照组为未甲基化的RNA (ss-A),进行RNA pull-down和质谱分析;2. 利用自主研发的一个新计算流程:基于已发表的RBP CLIP-seq (紫外交联免疫共沉淀测序) 数据库和已知的m6A修饰位点,筛选出潜在m6A结合蛋白。
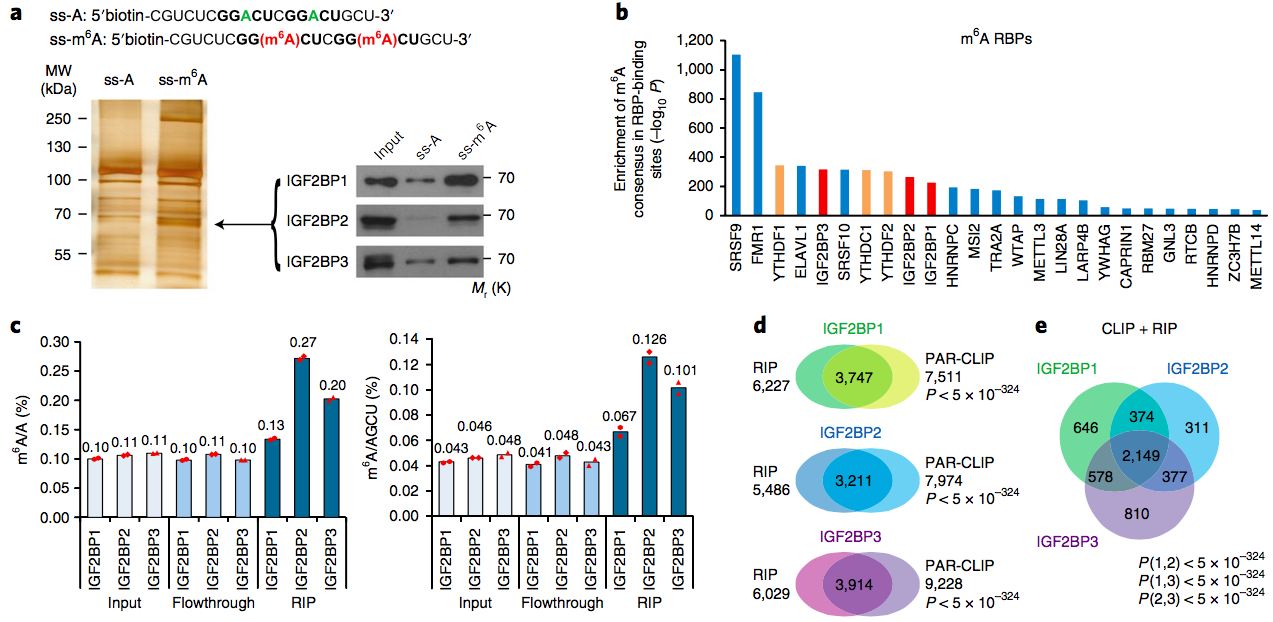
a.质谱分析表明3种IGF2BP蛋白与ss-m6A结合。b.计算结果表明这3种IGF2BP在112个显著的RBP里面排前15。c. FLAG-IGF2BP上富集有m6A修饰。d. 对比RIP-seq结果和已发表的PAR-CLIP数据,找到3种IGF2BP蛋白的高可信度靶基因。
通过这种方法,研究者鉴定得IGF2BP为m6A结合蛋白。
发现IGF2BP能使靶基因更稳定
为探究IGF2BP的作用,研究者进行了功能缺失实验,即敲降IGF2BP,进行RNA-seq,发现靶基因的表达因此减少:
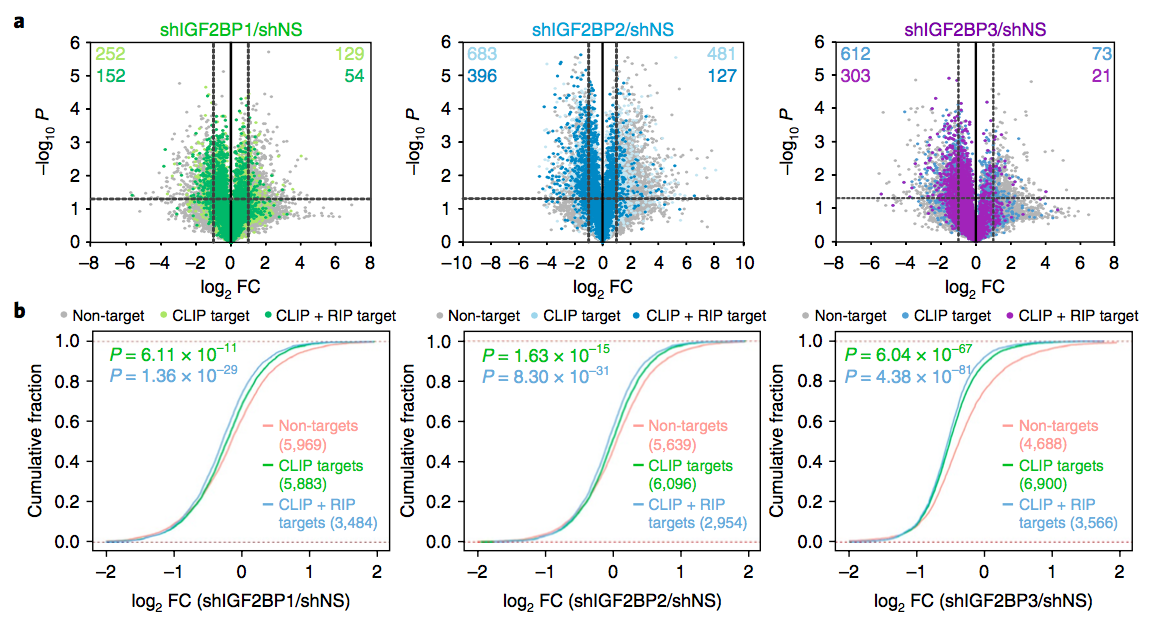
CLIP组靶基因 (CLIP数据所示的靶基因) 表达被抑制,特别是CLIP+RIP组靶基因 (RIP-seq与CLIP数据重叠的靶基因)。
IGF2BP的这个功能否受胞内m6A水平影响呢?降低m6A水平,常用的方法是敲降甲基转移酶METTL3或METTL14 ;研究者敲降HepG2细胞的METTL14后,进行m6A-seq和RNA-seq,发现有1,516个基因的m6A修饰相应减少,其中有418个基因的mRNA水平下降,而IGF2BP的高可信靶基因都在此列,下调显著。IGF2BP和METTL14的关联表明IGF2BP与m6A调控的基因相关。
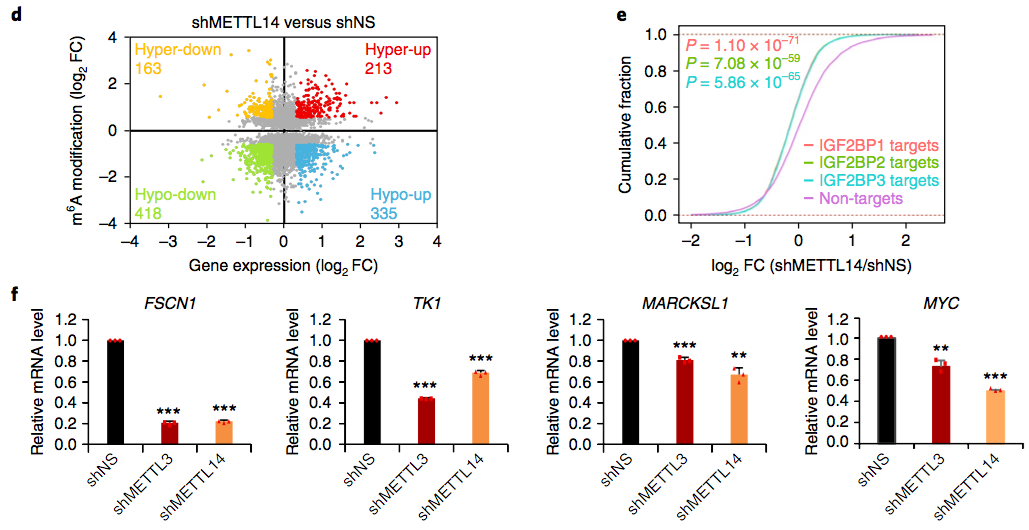
d. HepG2细胞敲降METTL14后的m6A水平与基因表达水平,两者均下降的418个基因即为m6A-Hypo-down组;e. shMETTL14细胞中IGF2BP高可信靶基因的mRNA log2 FC累积频数;f. METTL3或METTL14沉默后FSCN1、TK1、MARCKSL1 和MYC mRNA水平。
研究者敲降了细胞的IGF2BP3,检测mRNA稳定性,发现IGF2BP3和CLIP的高可信度靶基因的中位半衰期都显著缩短了将近一半!
还有哪些因子共同增强mRNA的稳定性呢?研究者对IGF2BP2复合物进行了pull down与质谱分析,发现存在着ELAVL1 (也叫HuR),MATR3和PABPC1,这3个都是已知的mRNA稳定剂(mRNA stabilizers)。以上结果表明IGF2BP2有助于其靶基因稳定翻译。
发现m6A对IGF2BP的作用
MYC是IGF2BP1的靶基因,研究者发现m6A修饰在MYC上积累,且m6A的峰与IFG2BP结合位点一致;CRD区 (不稳定编码区) 的m6A修饰丰度很高,且在METTL14敲降后显著减少;通过RIP和基因特异m6A实验,研究者证明CRD区域内存在IGF2BP结合、m6A修饰、METTL3和METTL13的结合:
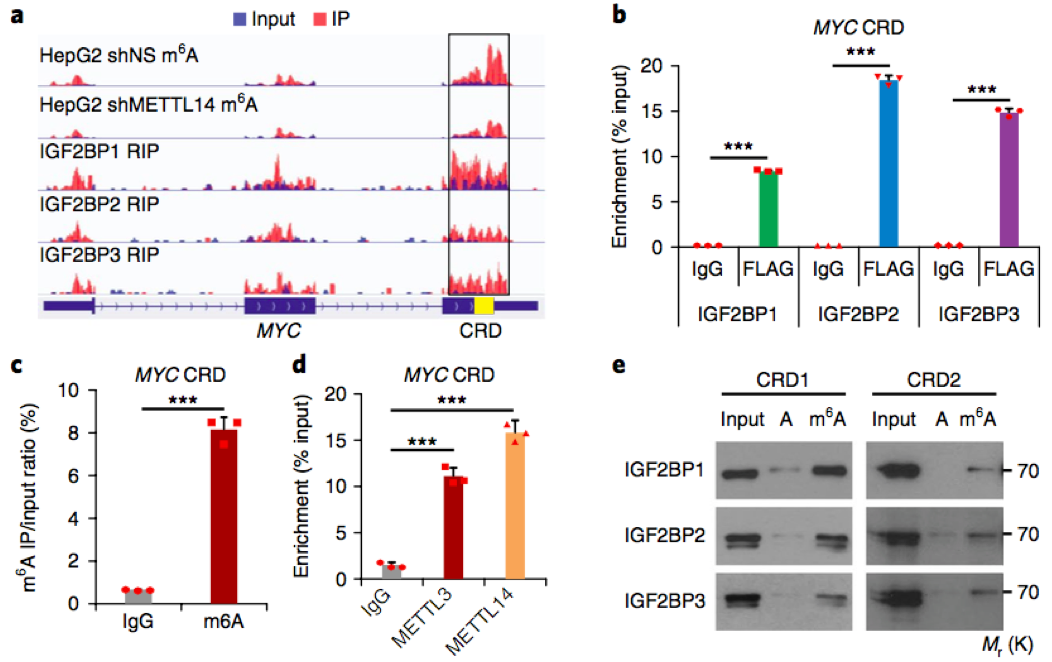
a, m6A-seq和RIP-seq所得的MYC mRNA与m6A峰的分布图 ;b, RIP–qPCR显示MYC CRD中的FLAG-tagged IGF2BPs;c, gene-specific m6A qPCR实验检测MYC CRD的m6A修饰 ;d, RIP–qPCR 显示METTL3和METTL14 结合到m6A CRD. e, RNA pull down,分别用有m6A或者没有m6A修饰 (即图中“A”实验组)的CRD1、CRD2来拉IGF2BP。
接下来,研究者利用荧光素酶报告实验,检测突变CRD序列之后IGF2BP的表达情况;联合RIP-qPCR实验,证明CRD的m6A修饰对IGF2BP结合到MYC以及调节MYC表达是必需的。研究者还发现KH3-4双结构域对IGF2BP与带m6A修饰的mRNA的结合,以及调控靶基因有着重要的作用。
验证IGF2BP的致癌作用
Cancer Genomics cBioPortal与TCGA的数据显示,3种IGF2BP在多种肿瘤中均有异常高表达,联系本研究中它们对致癌基因如MYC的稳定作用,研究者推测IGF2BP具有致癌作用,于是他敲降了HeLa和HepG2细胞的IGF2BP,结果抑制了这些肿瘤细胞的增殖、克隆形成能力和细胞迁移/侵袭,效果如同沉默MYC;利用CRISPR-Cas9技术敲除IGF2BP,再添加KH3-4突变的IGF2BP进行挽救实验,证明IGG2BP的致癌功能依赖于KH3-4,即m6A reader。
Epi老师:值得一提的是,NCB杂志同期还配发了题为“An additional class of m6A readers”的评述文章,对该工作给予了高度评价。这项研究鉴定得新的m6A识别蛋白家族IGF2BP1/2/3,并且发现它有着与已知的YTH结构域蛋白有着截然不同的功能,拓宽了我们对m6A识别蛋白的机制和功能的认识。Epi老师相信这个新发现会让m6A研究进一步升温!
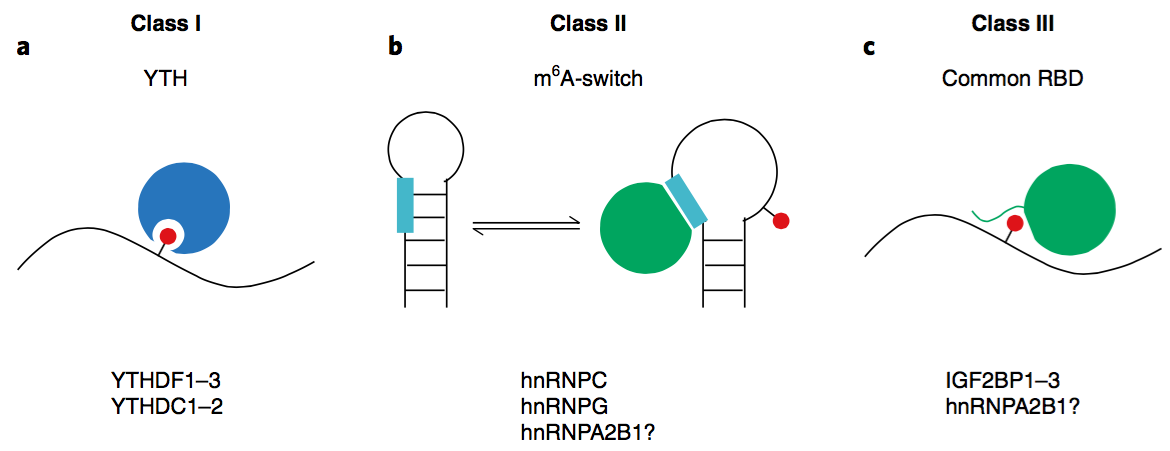
图为3类m6A识别(reader)蛋白。a, 第一类,YTH结构域(蓝色显示)蛋白,与m6A(红色显示)直接结合。b, 第二类,利用m6A开关机制与含m6A的转录本结合:RNA的m6A修饰会破坏碱基互补配对,提高单链RNA基序的可进入性,从而被m6A识别蛋白识别(绿色显示)。RNA基序可以与m6A位点重叠。这类识别蛋白有hnRNPC、hnRNPG,hnRNPA2B1也可能属于这类蛋白。c, 此研究新发现的一类识别蛋白,利用共有的RNA结合结构域(RBD)及其侧翼区(绿色显示)来识别含m6A的转录本。这类识别蛋白包括IGF2BP,利用KH结构域及侧翼区来选择性结合含m6A的RNA;hnRNPA2B1也有可能属于这类蛋白,它的RRM及侧翼区可能有助于m6A选择。
原文: Huang H, et al. Recognition of RNA N6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation. Nat Cell Biol. 2018 Mar;20(3):285-295. PMID: 29476152
杨建华教授个人简介
杨建华教授,中山大学博士生导师,长期致力于开发新算法、平台和实验方法研究非编码RNA基因和RNA修饰及其互作蛋白的结构、功能和作用机制。以通讯作者或第一作者身份在Nature Cell Biology、Cell Research、Nucleic Acids Res.、Cell Reports等杂志发表20多篇研究论文,以合作者身份在Nature Methods、Cell Stem Cell等杂志发表10多篇研究论文。开发starBase、starScan、snoSeeker和ChIPBase等工具被Nature等杂志引用超过1200次,受邀在Springer出版社出版了3篇关于非编码RNA研究方法的论著章节。目前担任Non-coding RNA杂志的编委。
● 振奋人心的3D基因组技术:GRID-seq探索RNA-DNA相互作用
- - - 推荐阅读 - - -

等了四年,miRBase 22 终于来了!
经过漫长的等待,3月13日,我们终于迎来miRBase22的官方发布了!上一次miRBase21的发布已经是2014年的事了。经过近四年的寂静,miRBase22给等待它的人带来了什么惊喜呢?快来了解一下! miRBase序
2018-03-19

年度Top10 | 2017, 超级增强子里程碑式的一年, 必看的10篇高分论文
这项研究找到了42名包含了各种不同亚型室管膜瘤患者进行实验,来寻找可以作为靶点的超级增强子 (SEs),并验证靶向SEs治疗癌症的可行性。 通过ChIP-seq、全外显子测序 (WES)、全基因组测序 (WGS)、转录组分析、DNA复制数分析以及DNA甲基化数据,研究人员从42名室管膜瘤患者的癌细胞中找到了近1700种
2018-02-05
趣谈exo| 特洛伊-外泌体假说 The Trojan Exosomes Hypothesis
相信exosomes研究者们,都接触过一个有趣的词:“特洛伊外泌体”。为什么外泌体会和古希腊的著名战争有关系?今天Epi老师来跟大家科普一下。 1TheTrojanExosomesHypothesis
2018-01-25
