振奋人心的3D基因组技术:GRID-seq探索RNA-DNA相互作用
许多技术可用于在染色质上定位某个RNA,比如ChIRP、CHART和RAP-DNA,它们利用互补序列捕获特定的RNA,然后进行高通量测序,找出染色质靶点,但是这些方法每次只能研究一个已知的RNA,不能研究全基因组范围上全部的RNA-染色质相互作用。
RNA在调节基因表达时还有协调功能DNA元件的作用。我们可以通过Hi-C分析染色质结构,检测所有可能的DNA-DNA相互作用;通过ChIA-PET分析特定因子介导的染色质相互作用,但是这些技术检测的主要都是非细胞类型依赖的拓扑相关结构域 (TAD) 内的,而染色质上的RNA还可能帮助我们找到转录活性相关的相互作用,帮助我们区分超级增强子和普通增强子。
为了解决上述问题,来自加州大学的付向东研究团队,研发了一种无偏差地检测全部染色质-RNA相互作用的技术——GRID-seq。接下来,Epi老师将和大家一起了解这项振奋人心的新技术。
01 GRID-seq技术原理
研究者首先选用了三阴性乳腺癌细胞株MDA-MB-231,构建无偏差的全基因组RNA-染色质相互作用图谱。GRID-seq方法原理如下图:
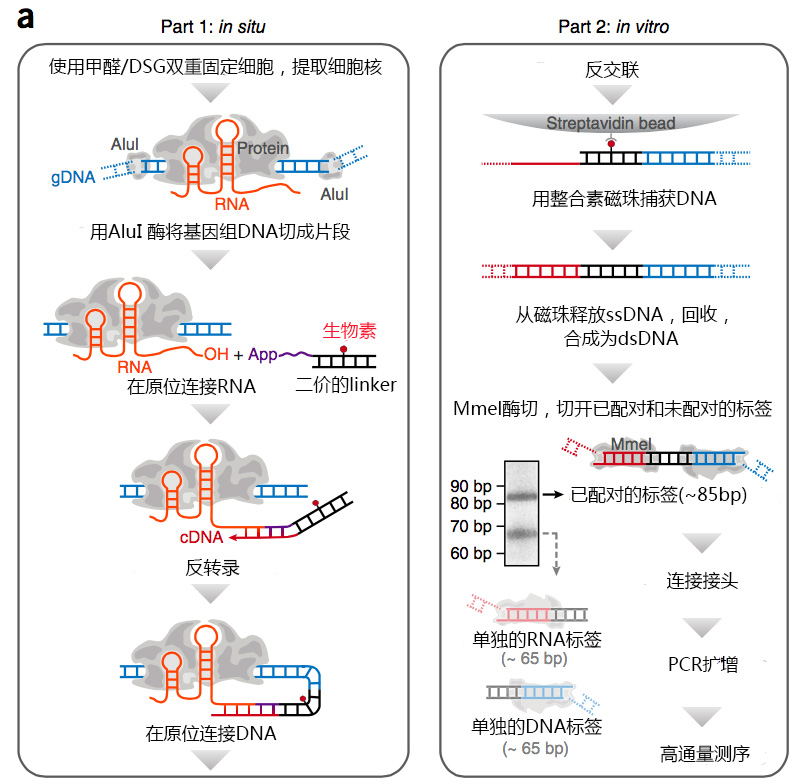
1. 预先设计特殊的linker,一端是单链RNA,另一端是双链DNA
2. 用DSG和甲醛固定RNA-DNA相互作用,提取细胞核。
3. 用AluI 进行酶切
4. 加入linker,linker的RNA与要捕获的RNA连接
5. 反转录,扩增连接的RNA
6. 去除游离的linker
7. linker的DNA与要捕获的DNA连接)
8. 用整合素磁珠捕获DNA
9. 从磁珠释放ssDNA,合成dsDNA,使用MmeI,将linker上设计好的酶切位点切开
10. 跑胶,得到两个DNA片段,85bp的是连接了RNA和DNA的linker,65bp的是只连接了RNA或者DNA的linker。
对人和果蝇基因组的测序结果都表明GRID-seq更倾向于检测染色质上的新生RNA。
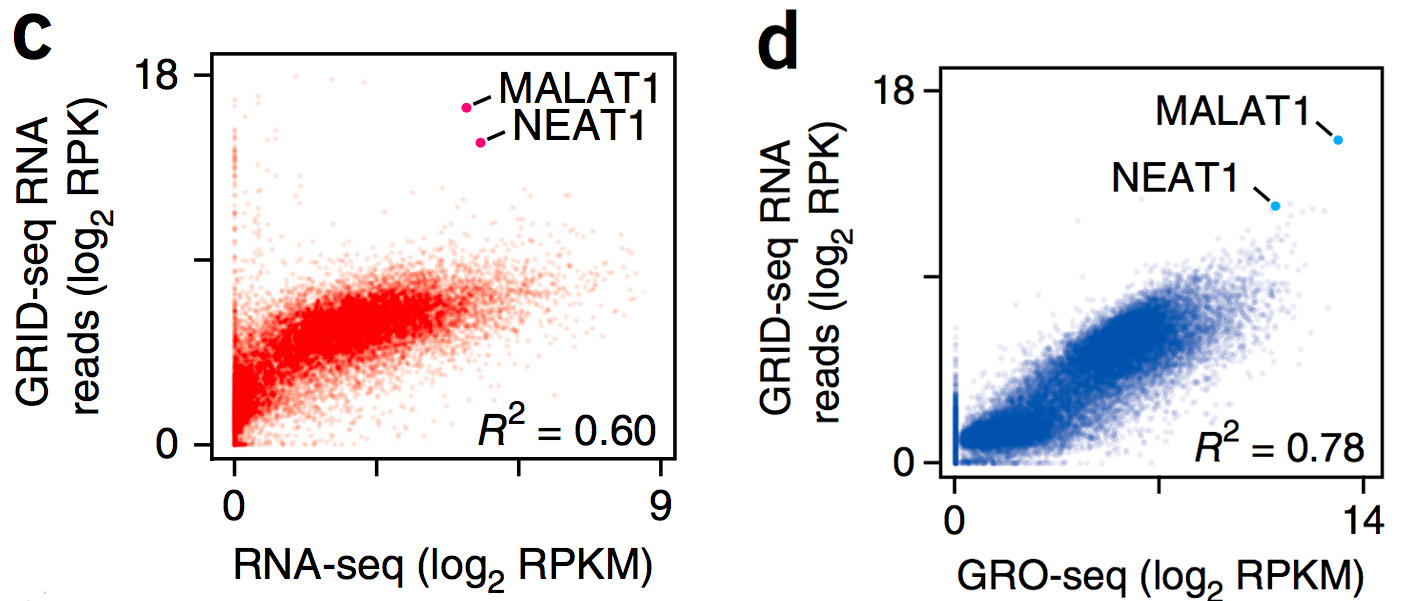
将GRID-seq结果与RNA-seq (c) 以及GRO-seq (d) 比较,发现它与后者检测到的新生RNA相关性更高。
为了排除非特异性的相互作用,研究者构建了一个鉴定特异性相互作用的背景模型:
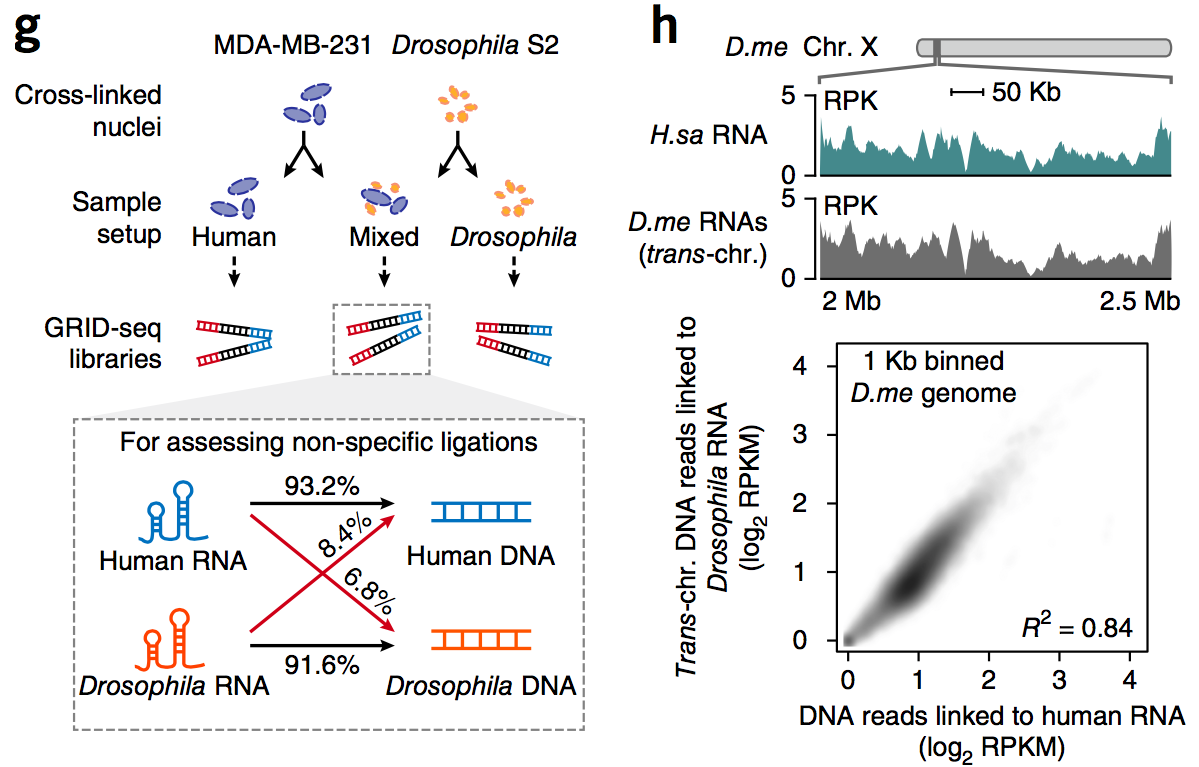
研究者使用人MDA-MB-231、果蝇S2细胞以及组合来构建文库。结果如g图所示,跨物种的结合较少。运用这些交叉物种的reads,研究者可利用小的果蝇基因组 (这样人RNA的read数目就会更多) 来构建一个真正的非特异RNA-染色质相互作用背景 (h图上图第一行) 。接下来,研究者用同一种细胞的mRNA-染色质相互作用做内源背景 (第二行),发现预测的内源背景与外源背景高度一致。
02 GRID-seq数据展示
对GRID-seq作了背景优化之后,数据质量如何呢?
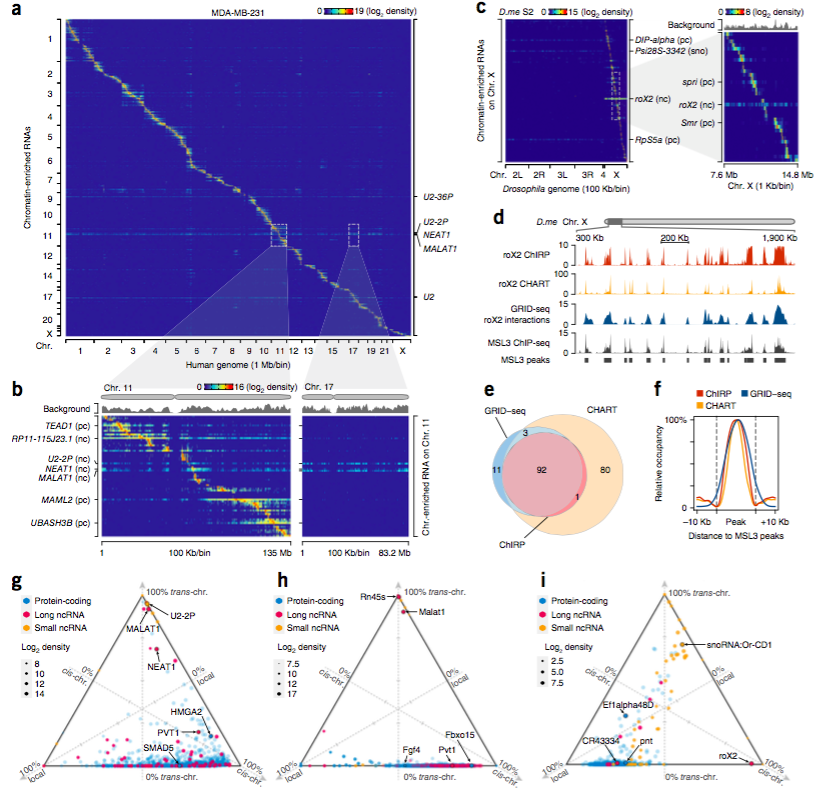
a. 热图显示,在MDA-MB-231细胞中只有少量的RNA与基因组发生反式相互作用;b. 第11条 (左) 和第17条 (右) 染色质上的RNA相互作用情况,可见MALAT1和NEAT1等与染色质上的几个位点相互作用;c. 对果蝇S2细胞的GRID-seq热图显示,roX2 RNA与果蝇X染色质的相互作用;d-f. 将GRID-seq数据与已公布的roX2 ChIRP、CHART数据以及MSL3的ChIP-seq数据相比,十分一致;g-i. 富集染色质的RNA的鉴定峰三角图,分别是人MDA-MB-231细胞,mESC细胞和果蝇S2细胞。
这些数据表明,通过GRID-seq技术能发现已知的特异RNA-染色质相互作用,并具有高特异性、高敏感性的特点。但是与关注单一靶标的捕获技术相比,有着all-to-all 特性的GRID-seq使每个与染色质相互作用的RNA的读数更少。
03 细胞类型特异的相互作用
GRID-seq检测的RNA-染色质相互作用是否为细胞类型特异的呢?研究者对MDA-MB-231和MM.1S细胞 (人多发性骨髓瘤细胞株) 进行联合分析,发现富集染色质的RNA主要都是细胞类型特异的。
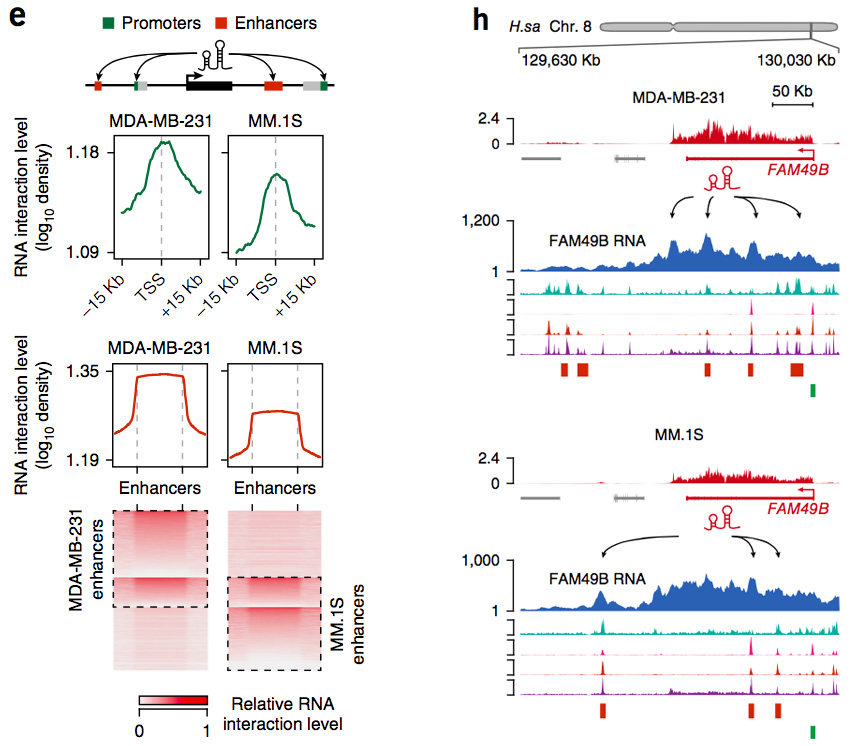
e. 除了未注释的DNA元件,RNA相互作用都以细胞类型特异的方式富集于活性启动子和增强子;h. 许多共有的捕获RNA,比如FAM49B RNA,在两种细胞中有着相似的染色质相互作用丰度,但是与不同的增强子有联系。
04 超级增强子上的RNA
RNA会作用于活性增强子,那么GRID-seq信号能不能反映普通增强子和超级增强子的强度差异呢?研究者用RNA相互作用水平为标准,给10,567个含有H3K27ac标签的活性增强子排序,并比较了超级增强子和普通增强子的染色质富集RNA的密度分布,结果如下:
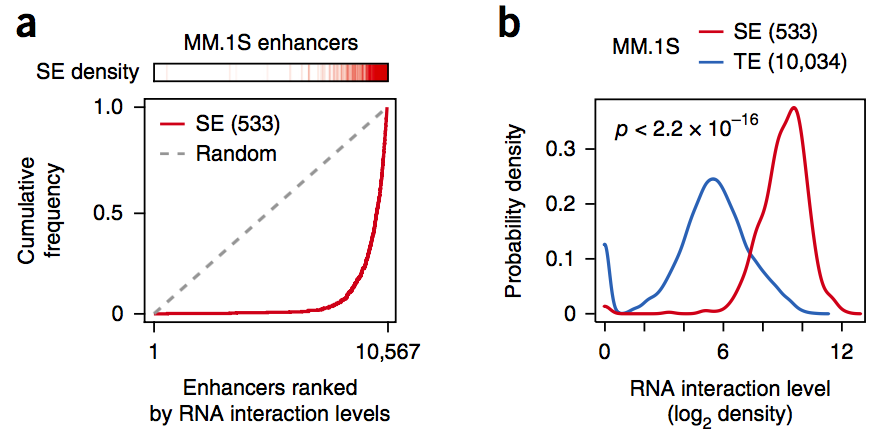
与RNA高度关联的增强子,正是MDA-MB-231和MM.1S细胞中的超级增强子(a),定量分析的结果也能证明这一点(b)。因此,染色质相互作用的RNA可成为鉴定超级增强子的条件。
05 RNA-染色质相互作用与TAD
基因组学的一个基本问题是,在细胞核的三维空间中,不同的DNA分子是如何相互作用的,现有的Hi-C和ChIA-PET技术就应用于研究这个问题。但是,Hi-C或者RNAPII ChIA-PET,难以将活性转录基因和非活性或转录上静止的基因区别开来。Hi-C检测的是全部类型的DNA-DNA相互作用,而GRID-seq捕获的是只生成RNA的基因 (RNA-producing genes ) 与DNA元件的相互作用,所以研究者选取了Hi-C单个基因的部分与GRID-seq的数据作比较:
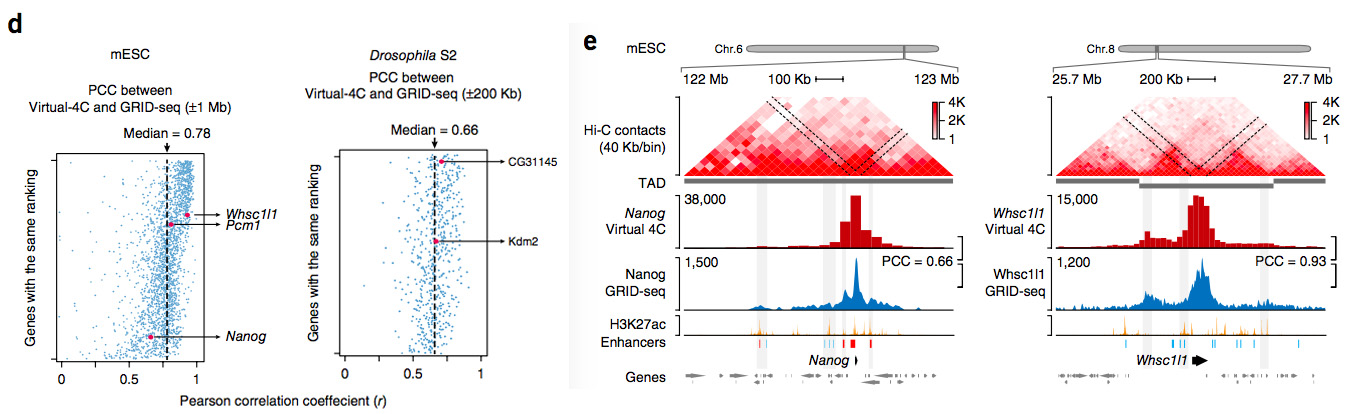
GRID-seq和Hi-C的Pearson相关系数表明它们有着高度一致性 (d),mESC细胞样本实验结果也证实了这一点 (e)。
这种相似性表明,染色质与RNA的相互作用主要存在于细胞核的高级结构中,GRID-seq可应用于预测与RNA生成有关的基因组相互作用,作为现有的3D基因研究技术的补充。
06 启动子与增强子的联系
研究者进一步运用GRID-seq研究转录相关的基因组相互作用,研究一个悬而未决的问题:增强子和活性启动子如何在3D基因组中联系?
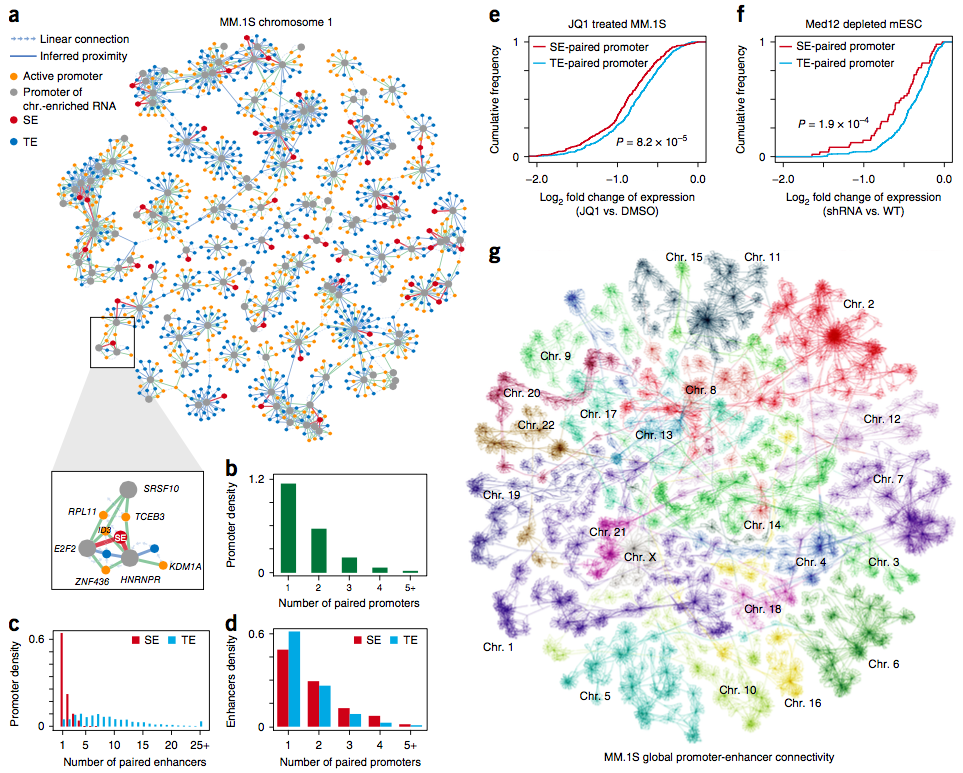
a. 研究者通过Cytoscape的self-organized布局,将启动子-启动子和启动子-增强子的网络直观化。b. 计算启动子-启动子和启动子-增强子相互作用,发现每个启动子关联的RNA最多可达4个,表明一个基因的启动子可能作为其他基因的增强子。c. 每个富集染色质的RNA能与多种普通增强子相互作用,但只和一两个超级增强子发生相互作用。d. 相反,无论普通或者超级增强子,大部分都与一个或两个基因来源的RNA相互作用。e. MM.1S细胞上,与超级增强子有关联的基因,对BRD4抑制剂JQ1干扰的敏感性会比只与一个普通增强子有关系的基因高。f. 用mESC细胞做类似的实验,在删除Mediator后,超级增强子作出的转录应答也更敏感。g. 利用GRID-seq和Cytoscape绘制全基因组相互作用网络,揭示单个染色体的结构,类似染色体涂染技术检测到的核领域。
上述结果表明,在很多增强子控制一个基因的同时,每个增强子,无论是普通还是超级增强子,还调控着一系列高度特异的靶基因;另外,尽管研究者检测到的不同染色体之间的启动子-启动子联系极少,但是确实观察到染色体间的特异相互作用。接下来的工作,需要进一步验证这些预测到的染色体间相互作用,特别是在单细胞水平,这有助于了解细胞核3D空间的基因组结构。
07 讨论
GRID-seq技术通过特殊设计的二价linker连接RNA和DNA,能实现对RNA-DNA相互作用的检测和分析。这项研究中展示的人和果蝇细胞的GRID-seq数据,表明少部分RNA可广泛参与反式染色体相互作用 (trans-chromosomal interactions)。
越来越多的数据表明,lncRNA和新生RNA都参与了染色质上一系列的调控过程,比如募集RNA依赖性的DNA甲基转移酶、转录激活剂或抑制剂。因此,GRID-seq技术的诞生,有望帮助我们发现更多RNA介导的调控活动。
原文:Li X, et al. GRID-seq reveals the global RNA-chromatin interactome. Nat Biotechnol. 2017 Oct;35(10):940-950.
- - - 推荐阅读 - - -
lncRNA研究策略—来自Science一作王品博士的经验分享
表观生物公众号很荣幸邀请到了王博士为广大的 lncRNA研究者分享了他对非编码RNA的研究心得和策略,大家一起来学习交流吧!
2017-11-21
这篇 Cell 文章告诉你如何研究外泌体 miRNA
研究外泌体miRNA的文章越来越多,可是高分的并不多。怎么样的外泌体实验结果在审稿人眼中才具有说服力?Epi老师今天带你们看看这篇最近发表于Cell的外泌体miRNA文章。 肥胖引起的慢性组织炎症是胰岛
2017-11-02

Mol Cell: Spt6利用超级增强子维持细胞多能性
延长因子Spt6如何利用超级增强子、多梳蛋白使ESC细胞保有多能性?10月19日,Mol Cell上发表的一篇来自NIH研究者的文章解答了这个问题。
2017-10-30
