超级增强子研究方案
一、研究背景和研究意义
1.1 增强子
增强子(enhancer),是DNA上一小段可与蛋白质(反式作用因子;trans-acting factor)结合的区域,与蛋白质结合之后,基因的转录作用将会加强。增强子可能位于基因上游,也可能位于下游。且不一定接近所要作用的基因,甚至不一定与基因位于同一染色体[1]。这是因为染色质的缠绕结构,使序列上相隔很远的位置也有机会相互接触。增强子具有以下特征)[1-4]:
(1) 增强子DNA 序列处于染色体疏松的区域,与核小体中组蛋白的修饰,转录因子的结合有关;(2) 增强子活性与其DNA 序列结合的组蛋白H3 的第4 位赖氨酸单甲基化(H3K4me1)和第27 位赖氨酸乙酰化(H3K27ac)修饰程度成正相关[6];(3) 增强子发挥功能需要增强子区域和启动子的区域的直接相互作用,形成三维环状结构(3D-loop)。增强子和启动子的相互作用由多种蛋白介导,如Mediator 复合体、Cohesin 等[5.6]。
在真核生物细胞里,DNA的染色质复合体结构像原核生物的超螺旋一样折叠。所以虽然增强子与基因相距很多核苷酸,但在几何上两者距离很近。使增强子与总转录因子及RNA多聚酶II的相互作用成为可能。增强子可以在被它调控基因的上游或下游。而且增强子不一定靠近转录起始位点才能调控基因转录,已发现有些距离达几十万碱基对。增强子通过与激活蛋白的结合对启动子(不直接对启动子本身)起作用。这些激活蛋白与中介复合物(辅激活物)相互作用,后者通过使用多聚酶II和总转录因子开始基因转录。曾经在内含子内发现增强子。有时增强子的方向颠倒后并不影响它的功能。而且增强子可被切除并插入到染色体的其他位置,仍然影响基因转录。这就是虽然内含子并不转录但其多态性会起作用。
1.2 超级增强子
超级增强子(Super enhancer, SE)是一类具有超强转录激活特性的顺式调控元件,2013年由美国美国白头生物医学研究所(WhiteheadInstituteforBiomedicalResearch)学者Richard A. Young首次提出。与普通增强子(Typical enhancer, TE)相比,超级增强子区域跨度范围通常可达 8-20 Kb,远高于普通增强子的200-300 bp跨度范围。更重要的是,超级增强子比普通增强子具有更高密度的转录激活相关组蛋白修饰(H3K27ac、H3Kme1等)、Mediator复合体和Bromodomain containing 4 蛋白(BRD4,与组蛋白乙酰化修饰位点结合)的结合;辅因子(Mediator等)及转录因子富集密度 [7]。以上特点决定了超级增强子具有强大的调控功能。
(1) 超级增强子具有高密度的H3K27ac 和H3K4me1 修饰,以及Mediator复合体和Bromodomain containing 4 蛋白(BRD4,与组蛋白乙酰化修饰位点结合)的结合;(2) 超级增强子结合的转录因子以及与转录活性相关的染色体的标记比普通增强子高很多;(3) 超级增强子调控的基因比普通增强子调控的基因表达水平高很多;(4) 组成超级增强子的单个增强子也可以像普通增强子一样激活基因转录;(5) 超级增强子可以结合组织中特异的转录因子;(6) 与普通增强子相比,超级增强子活性对于转录因子的阻断更敏感[8,9]。
近些年来研究发现,超级增强子在癌症发生、细胞分化、免疫应答等重要生物学过程中发挥着重要调控功能,其所调控的基因包含原癌及抑癌基因、细胞身份决定基因、炎症通路关键基因等 [10-13]。由于许多肿瘤细胞关键致癌基因由超级增强子驱动,超级增强子有潜力成为理想的抗癌靶点,已成为世界科技前沿的研究热点。
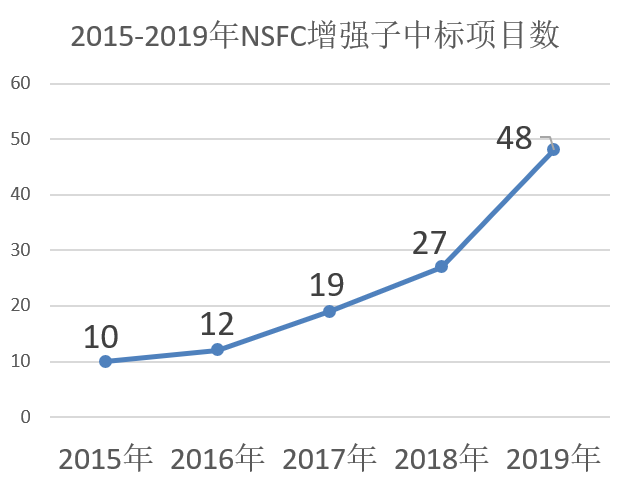
增强子研究逐渐升温
1.3 超级增强子与肿瘤
Richard A. Young最早是在多发性骨髓瘤细胞中发现超级增强子的区域募集了高浓度的 Mediator复合体和BRD4[14]的,并证明超级增强子调控的基因(MYC、IRF4、PRDM1、XBP1)对于多发性骨髓瘤的发生和发展起到关键的促进作用[14]。
目前,超级增强子已被报道与多种疾病发生发展有关,其中包括了不同类型的肿瘤,比如弥漫性大B细胞淋巴瘤、T细胞急性淋巴细胞白血病、默克尔细胞癌、急性髓性白血病、小细胞肺癌、卵巢癌、上皮癌、 鳞状细胞癌、黑色素瘤、乳腺癌、食管鳞状细胞癌和结肠癌等[15]。
大部分研究表明,超级增强子的激活对肿瘤细胞的恶性转化具有促进作用。超级增强子在肿瘤细胞中调控关键的癌基因,从而对肿瘤生成和肿瘤特性维持起到关键作用。超级增强子能够富集肿瘤细胞依赖的信号通路的转录因子,比如在 Wnt 信号通路异常引起的结肠癌细胞中,相关的超级增强子区域富集了很多由 Wnt 信号通路终端的转录因子 4 (tra-nscription factor 4, TCF4),通过激活或者抑制 Wnt 信号通路,可以控制超级增强子调控的基因转录[16,17]。在雌激素受体(estrogen receptor, ER)阳性的乳腺癌细胞中,相关的超级增强子区域聚集了大量的ERα; 而在三阴性乳腺癌细胞中缺少类固醇激素的表达, 与其相关的超级增强子区域富集了完全不同的转录因子[18]。
在另一方面,肿瘤细胞的信号通路可以反过来对超级增强子的活性进行调控。Licht 等人发现 Ras-Erk 活性与超级增强子的活性密切相关:抑制Ras蛋白的活性会导致超级增强子相关的特征(如H3K27ac)消失、活性下降、降低相关基因转录;激活Ras可以增强调控癌基因的超级增强子活性[19]。此外,促癌信号通路可以通过操纵转录机器调节超级增强子的活性,比如通过调节RNA聚合酶II的转录暂停状态[20]:肝癌中YAP(Yes associated protein)蛋白结合到超级增强子上,募集Mediator和CDK9(细胞周期素依赖性激酶9)使暂停的Pol II进入延伸状态,促进癌基因转录。通过激活超级增强子促进癌基因的转录。
综上,超级增强子可以作为连接癌基因信号通路和维持肿瘤细胞特性的基因转录表达的渠道;反过来,信号通路也能够调控超级增强子与转录因子在超级增强子区动态结合:在 NOTCH1 异常导致的 T 细胞白血病(T-ALL) 细胞中,NOTCH1在基因组上具有普遍的结合,但是只有不到10%的基因对于NOTCH1信号通路的改变有应答,而这些应答基因的NOTCH1均结合在对应的超级增强子上,如果这些位点丢失,NOTCH1的结合就会导致超级增强子的特征消失[21]。
除了肿瘤以外,与超级增强子异常相关的疾病还包括有:阿尔兹海默症、类风湿关节炎、克罗恩病[22]。这些与免疫相关的疾病主要是由于T细胞产生了突变,而通过进一步的分析发现,这些突变位点大量分布于超级增强子区域内[22]。
1.4 (超级)增强子与miRNA
研究表明,增强子与microRNA(miRNA)间同样存在复杂的调控关系。Xiao等[23]发现增强子对miR-24-1邻近基因的转录是必需的miR-24-1通过激活增强子RNA(eRNA)表达,改变组蛋白修饰,增加增强子位点p300和RNA Pol II的富集,从而激活转录;Suzuki等证明增强子可以通过募集Drosha和DGCR8,调控成熟的miRNA在细胞中的水平,进而影响基因的表达[24]。
1.5 超级增强子与相分离
物质的相分离是在自然界中普遍存在的物理现象,通常指的是在均匀的混合液/气态物质中自发形成的不同相。生物大分子的相分离(phase separation)凝聚是驱动无膜细胞器形成的主要机制[25]。在体内相分离通常是一个动态的过程,大部分情况下指的是“液–液相分离”(liquid-liquid phase separation)。
2009年,Hyman等人发现线虫胚胎中的RNA和包含蛋白质的结合体P颗粒(P granules)实际上是由蛋白质通过相分离形成的凝聚小球[26]。2011年,该实验室对核仁进行了类似的研究,证实了相分离是核仁发生的基础[27]。2017年,Richard A. Young实验室提出一个解释超级增强子通过相分离参与基因调控的模型[28]。2018年,该实验室通过实验证明了上述模型[29]:胚胎干细胞中的转录共激活因子MED1和BRD4通过相分离形式在超级增强子处成簇分布,并且起到调控基因表达的作用。
如今越来越多的研究者开展了相分离研究。现有的研究成果表明,相分离现象对细胞核的染色质空间结构的组织调控非常重要,探究相分离现象与染色质的结构和功能的关系具有重要的意义。
1.6 超级增强子的临床应用
超级增强子的先驱者Richard A. Young与JQ1/iBET研发者Bradner J.E.这两位科学家曾预言超级增强子具有广阔的研发前景和价值,必将成为下一个药物研发的黄金靶点,有望开发一种精确影响基因调控元件的药物。这两位科学家也因此联手成立Syros公司,研发针对超级增强子的抗癌药。经过了6年的发展,如今以超级增强子为靶点的肿瘤治疗用小分子抑制剂已进入临床研究阶段。
表 1 在肿瘤治疗中以超级增强子为靶点的小分子抑制剂研究现状[30]
|
小分子抑制剂 |
靶点 |
疾病 |
临床研究 |
文献 |
|
JQ1 |
BRD4 |
多发性骨髓瘤 |
— |
31 |
|
Merkel 细胞癌 |
— |
32 |
||
|
iBET151 |
BRD4 |
白血病 |
— |
33 |
|
iBET762 |
BRD2, BRD3 BRD4 |
NUT 中线癌 |
I期临床(NCT01587703) |
33 |
|
BETd-246 |
BRD2, BRD3 BRD4 |
三阴性乳腺癌 |
— |
34 |
|
OTX015 |
BRD2, BRD3 BRD4 |
成神经细胞瘤 |
临床前研究 |
35 |
|
弥漫性大 B 细胞淋巴瘤 |
I期临床(NCT01713582) |
35 |
||
|
急性淋巴细胞白血病 |
I期临床(NCT01713582) |
36,37 |
||
|
NUT 中线癌 |
I期临床(NCT02259114) |
33 |
||
|
多行性成胶质细胞瘤 |
I期临床(NCT02296476) |
33 |
||
|
CPI0610 |
BRD4 |
多发性骨髓瘤 |
I期临床(NCT02157636) |
33 |
|
淋巴瘤 |
I期临床(NCT01949883) |
33 |
||
|
THZ1 |
CDK7 |
食管鳞状细胞瘤 |
— |
38 |
|
成神经细胞瘤 |
— |
38 |
||
|
成人T细胞白血病 |
— |
39 |
||
|
小细胞肺癌 |
|
40 |
||
|
SY-1365 |
CDK7 |
成人晚期实体瘤 |
I期临床(NCT01587703) |
Syros公司 |
|
乳腺癌(与氟维司群联合) |
临床前研究 |
Syros公司 |
||
|
SY-1425 |
RARα |
急性髓样细胞样白血病 |
I期临床(NCT01587703) |
Syros公司 |
|
骨髓增生异常综合征 |
I期临床(NCT01587703) |
Syros公司 |
||
|
THZ531 |
CDK12/13 |
急性T细胞淋巴瘤 |
— |
41 |
|
Lee011 |
CDK4/6 |
尤因肉瘤 |
— |
42 |
2.2 研究内容及关键技术
1. 研究内容:采用H3K37ac ChIP-seq鉴定(超级)增强子
关键技术:
超级增强子的鉴定,依据的是增强子转录活性标记分子结合水平强度的差异,这些分子包括辅因子(如Mediator和cohesin)、组蛋白修饰标记(如H3K27ac和H3K4me1)、染色质修饰分子(如p300)等。在鉴定过程中,首先通过ChIP-Seq分析这些增强子转录活性标记分子在基因组上的富集情况,确定活性增强子位点。之后再对所有活性增强子进行分析,鉴定得到超级增强子。通过与转录组测序数据进行关联分析,可发现超级增强子所调控的靶基因。
技术应用
1. 发现致病驱动基因
2. 分析疾病关联变异位点易感性
3. 开发基于超级增强子复杂性疾病的精准诊断和药物开发
样本要求
样本类型:
1. 甲醛交联的细胞样本,细胞数量≥1×107
2. 组织样本需评估
仅限人、大小鼠,其他物种需评估
样品分组
1. 常规要求至少 2 组样品,包括对照组和实验组
2. 样本数建议:3 VS 3
主要分析内容
1. TE 与 SE 鉴定分析
2. TE 与 SE ChIP-seq 峰图对比
3. TE 与 SE 信号富集图绘制
4. TE 与 SE 差异(长度与数目)分析
5. SE 调控靶基因 GO、KEGG 分析
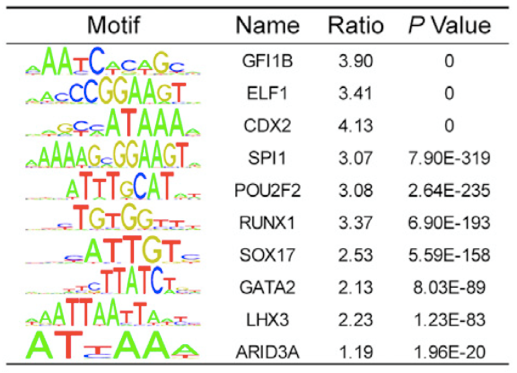
6. SE 结合转录因子预测(motif)
2. 研究内容:采用CAPTURE技术研究增强子相互作用的DNA、RNA和蛋白质
关键技术:
CAPUTURE(CRISPR affinity purification in situ of regulatory elements),即CRISPR亲和纯化原位调控元件,利用CRISPR的特异靶向能力,以及生物素-链霉亲和素的互作优势,建立高分辨率、位点特异性极高的顺式调控元件(如增强子)的蛋白质、RNA和远程的DNA互作网络图谱。
技术原理:
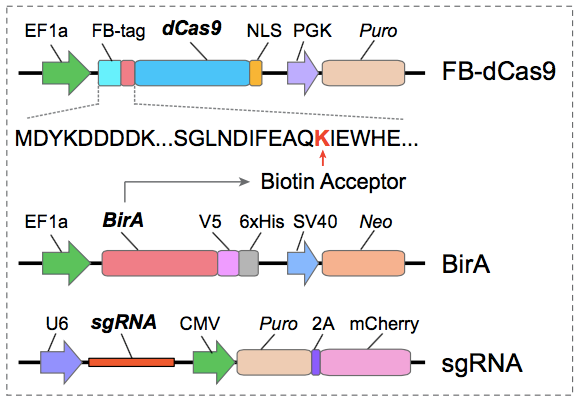
CAPTURE系统三个元件:FB-dCas9(N端含FLAG和生物素接受位点的dCas9)、生物素连接酶BirA,靶标特异性的sgRNA
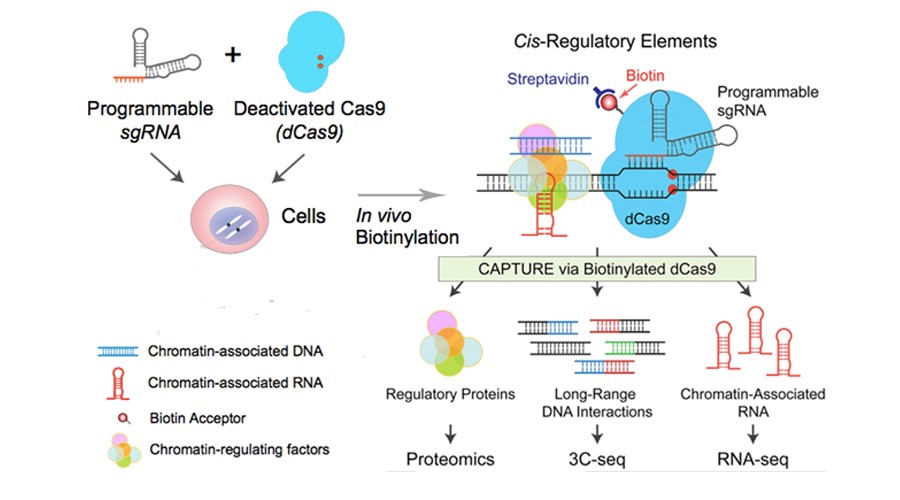
在体内用生物素连接酶BirA给dCas9标记上生物素,加上序列特异的sgRNA,高亲和链霉素就能分离纯化基因组位点相关的大分子。提取到的蛋白、RNA和DNA复合物,分别用质谱和高通量测序研究,获知顺式调控元件的蛋白、RNA和远程的DNA相互作用。
CAPTURE-seq/MS技术有以下优点:
(1) 灵敏度高。生物素和链酶亲和素的亲和力 (affinity) 达到Kd = 10-14mol/L,是抗体介导的相互作用的1000倍以上,这样就可以更高效和更稳定地捕获蛋白—DNA复合物。
(2) 特异性高。这个方法没有使用抗体,能显著减少非特异性结合,此外,生物素—链霉亲和素出众的稳定性,利于进行严格的纯化过程,减少蛋白污染。
(3) 适用于多种方法。研究者可以改变sgRNA序列或组合来操纵dCas9 /sgRNA系统,那就可以作为研究染色质相互作用的高通量测序分析媒介。
示意图:

样本要求
样本类型:活细胞样本,1x108个细胞每样本;
样本物种:仅限人、大小鼠,其他物种需评估。
分析内容:
CAPTURE-seq
1. 去接头污染,去低质量reads和测序质量评估
2. CAPTURE-seq序列与参考基因组序列的比对
3. CAPTURE-seq唯一reads在全基因组的分布
4. Peaks鉴定及基因元件分析
6. Peaks相关基因筛选与GO功能聚类分析、Pathway 分析
7. Peaks可视化
8. Peaks Motif 分析
1. CAPTURE捕获产物蛋白质质谱鉴定结果
1.1 唯一谱图分布
1.2 唯一肽段分布
1.3 蛋白质量分布
1.4 蛋白覆盖度分布
1.5 肽段长度分布
2. CAPTURE捕获产物详细鉴定列表
2.1 谱图鉴定列表
2.2 肽段鉴定列表
2.3 蛋白鉴定列表
3. CAPTURE捕获产物功能分析
3.1 GO注释
3.2 COG注释
3.3 Pathway注释
3. 研究内容:利用ATAC-seq研究染色质开放性,初步判断是否涉及表观遗传调控机制
关键技术:
染色质开放性是指真核生物染色质DNA和转录因子等蛋白能否结合的开放程度,这一特性反映了染色质转录活跃程度,结合其他DNA修饰(如甲基化)信息,特定条件下的染色质开放性变化可以提供大量的基因表达调控信息,为各种蛋白结合新位点的发现指明方向。还可分析初生组织和细胞(如胰岛β细胞)、临床患者样本,基于染色质开放性特征对患者和样品进行分类。
ATAC-seq(Assay of Transposase Accessible Chromatin with High-throughput sequencing),即染色质开放性检测,利用转座酶研究染色质可接近性的高通量测序技术 [9]。2013年,美国斯坦福大学William Greenleaf教授报道了其研发的ATAC-seq,利用DNA转座酶取代传统研究开放染色质区域使用的DNA酶,准确率与传统方法一致,且重复性更高,只需少量细胞/组织,就能完成高质量的测序,是目前研究染色质开放性的最优策略。
技术原理:

图1-12. ATAC-seq技术原理
A, ATAC-seq 文库构建示意图。转座子只能插入开放的染色质 DNA,所以利用 Tn5 转座酶,将携带测序标签的转座复合物加入到细 胞核中,后续利用标签进行 PCR 扩增,即可识别开放染色质; B, 转座后成为 DNA 片段。在 72° C 延长,与 adapter 连接,再扩增,随 后 PCR 扩增出来的序列就包含了 adapter、共有序列末端、测序标签。
技术应用:
对于未知表观遗传是否在细胞系统的应答发挥作用的情况,或者不清楚哪种组蛋白修饰、转录因子是最重要的, 或者细胞数量很少的情况前提下,ATAC-seq是比ChIP-seq更适合的一种研究技术。
1. 初步判断所研究的课题是否涉及表观遗传调控机制
2. 结合基序分析,识别参与基因调控的转录因子
3. 识别转录因子调控的靶基因和功能元件
4. 与超级增强子鉴定联合分析,明确活性超级增强子的范围
5. 通过开放的染色质图谱,可以更好地了解药物或疾病的基因调控和细胞应答机制;识别驱动细胞命运、疾病或应答相关的转录因子
技术优势
1. 灵敏性高,低细胞起始量,每个样本 50,000 个细胞即可
2. 实验步骤简化,实验重复性好,成功率高
3. 能同时揭示开放染色质的基因组位置、DNA结合蛋白和转录结合位点的相互作用
样本要求
样本类型:
1. 活细胞样本,50,000 个细胞每样本
2. 组织样本,质量≥ 100mg
仅限人、大小鼠,其他物种需评估
样品分组
1. 常规要求至少 2 组样品,包括对照组和实验组(临床样本为正常人组和患者组)
2. 样本数建议:3 VS 3
分析内容
1. 基因组比对分析
2. 基因组富集分析
2.1 富集区域基本信息
2.2 富集区域分布特征
2.3 Peak 相关基因 GO 分析
2.4 Peak 相关基因 KEGG 分析
3. 富集区域 motif 分析
4. 研究内容:采用HiChIP技术研究与增强子相互作用的蛋白质
关键技术:
HiChIP(in situ Hi-C followed by chromatin immunoprecipitation)是一种用于解析染色质构象的全新技术,该方法由Howard Chang 实验室建立,2016 年11 月份发表于Nature Methods[7]。HiChIP 结合了Hi-C 与ChIA-PET 两大技术的特点,并进一步与转座酶介导文库构建技术结合起来,用更小的数据量获取更高分辨率的染色质三维结构信息。利用HiChIP技术,可在三维构象水平研究DNA 和蛋白接触点,挖掘一维测序数据(ChIP-seq 和ATAC-seq)无法揭示的相互作用。
表1-1. Hi-C 和ChIA-PET 及HiChIP 技术的比较
|
|
优 点 |
不 足 |
||
|
Hi-C |
解析所有染色质构象 |
深度测序需要完全解析染色质特征,染色质构象数据量庞大;特异性不好;信号背景比低 |
||
|
ChIA-PET |
产生感兴趣蛋白因子有关的长范围联结 |
细胞需要量大;产生小片段的reads;感兴趣基因的reads 很少;低效,有假阳性 |
||
|
HiChIP |
细胞需要量小;目标基因reads 多;信号背景比高;特异性好;快速,高效,方便 |
仅生成感兴趣蛋白因子结合的染色质构象 |
||
技术原理
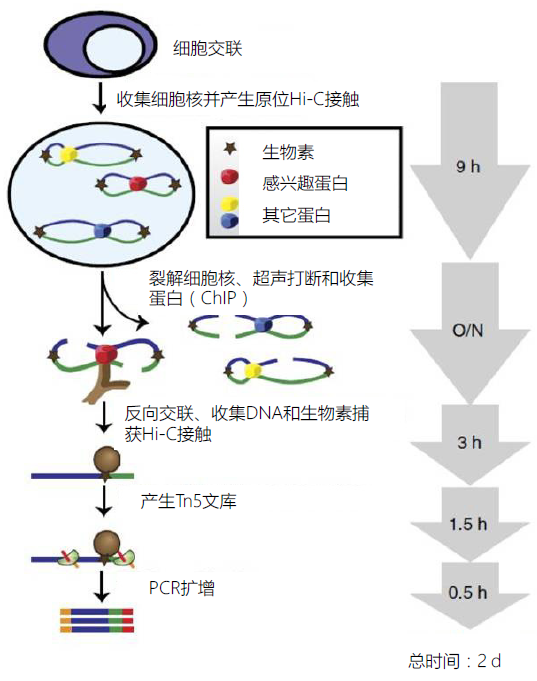
图1-9. HiChIP-seq 技术流程
HiChIP 在裂解细胞前就在细胞核中交联,从而降低假阳性,最大化提高DNA contact 的捕获效率。然后收集细胞核,在原位产生Hi-C 交联,利用生物素标记DNA 末端,裂解细胞核,超声打断DNA,后续用特异性的抗体进行ChIP 实验。得到DNA 蛋白复合物后,进行DNA 洗脱和反向交联。随后,利用生物素捕获Hi-C 交联和文库制备, 上机测序。
• 技术应用
1. 针对特定蛋白的染色质互作构象分析
2. 转录因子作用机制研究
3. 表观修饰对基因调控的机制研究
• 技术优势
1. 构象信息读取的产量提高了10 倍以上
2. 相对于ChIA-PET,样本要求降低了100 倍以上
3. 所需细胞数量低于Hi-C 且比Hi-C 具有更大的信噪比
4. 可获取特定的染色质三维结构信息
• 样本要求
样本类型:活细胞样本,交联细胞数量≥ 1.5×107
样本物种:仅限人、大小鼠,其他物种需评估
其他注意事项:
1. 前期准备需要固定细胞,提供细胞沉淀,通过干冰运输。细胞固定方案请咨询技术支持
2. 不具备前期准备条件的,需提供活细胞及配套培养基, 常温运输
5. 研究内容:采用GRO-seq技术研究新生RNA、eRNA
关键技术:
GRO-seq(Global nuclear Run-On sequencing),是一种专门测量新生RNA的技术。新生RNA的变化直接反映了转录调控的多种机理。利用GRO-seq技术,可以知道RNA聚合酶所在位点,从而检测基因组上正在进行的转录;还能高效检测到目标样本中的eRNA分子。
增强子相关RNA(eRNA)是由基因功能元件增强子转录产生的一类长链非编码RNA,在基因转录调控和疾病的发生发展中发挥重要的作用,是近年来非编码RNA领域的新热点。多数eRNA具有转录双向性和非多聚腺苷酸反应性,并且eRNA 的产生通常和近端基因诱导相关联。eRNA主要通过和其目标启动子相连并相互作用来实现对下游基因的调控,利用分子关联分析和DNA 圈连均证明了eRNA这一作用机制。目前,eRNA已被证明和前列腺癌、乳腺癌和甲状腺癌等多种癌症相关,将有望作为疾病治疗的创新靶点。
由于eRNA在结构上稳定性低,存在周期短,因此常规的转录组测序方法难以灵敏的捕捉到。利用表观生物创新的GRO-seq测序技术可以检测eRNA分子,为疾病的分子机制研究开拓全新的研究领域。全基因组转录的“快照”(snapshot),直接检测启动子近端POL暂停现象
技术原理:
先提取细胞核,添加Run on buffer,然后加入核糖核苷酸类似物BrUTP,使新生RNA带上BrU标记。利用针对BrUTP的抗体,免疫提取出带标记的新生RNA,构建文库,进行高通量测序。
技术应用
1. 获得全基因转录的“快照”(snapshot),定位RNA聚合酶位点和转录位点
2. 可直接观察启动子近端RNAP II暂停等现象,进一步探索增强子等远程调控元件的转录调控机制
2. 鉴定eRNA
样本要求
样本类型:细胞样本,细胞数量≥1×107
仅限人、大小鼠,其他物种需评估
分析内容
1. 去接头污染,去低质量reads和测序质量评估
2. 测序序列与参考基因组序列的比对
3. Reads在全转录组的分布;
4. Reads在转录起始位点(TSS)附近的meta分析;
5. 转录暂停基因分析
6. eRNA分析
7. 差异基因筛选与GO功能聚类分析、Pathway 分析
6. 研究内容:利用dCas9-p300提高目的基因H3K27ac水平,激活目的基因
关键技术:
dCas9-p300是基于CRISPR/Cas 9基因调控系统的一种靶向乙酰化工具,把dCas9蛋白融合到人乙酰转移酶p300的催化核心而成。dCas9-p300能催化它靶位点的H3K27乙酰化,有效激活启动子、近端增强子和远端增强子上的靶基因。它能够实现靶向乙酰化,是表观基因组调控编辑的有力工具。
技术原理:
dCas9,即dead cas9,工程化缺陷型核酸酶,是Cas9蛋白的突变体,Cas9蛋白的RuvC1和HNA两个核酸酶活性区域同时发生突变。因此,dCas9蛋白的内切酶活性完全消失,只保留由gRNA引导进入基因组的能力。dCas9可以与不同蛋白形成融合蛋白,再由特定的gRNA导向而结合于特定的基因组位点,继而经由不同融合蛋白行使不同于Cas9的其他功能。
人类E1A相关蛋白p300是组蛋白乙酰化的关键成分,p300的催化核心与dCas9融合,组成了dCas9-p300。当gRNA靶向编码区或启动子区时,这个系统可成功诱导基因高表达。特别是,当靶向启动子或增强子时,基因表达可增强50至10,000倍。通过转录组分析评估得到,这种靶向的乙酰转移酶对基因的激活在基因组上是高度特异的,该系统可以精确调控某段染色质的变化。
技术应用
1. 增加靶向增强子和其下游基因启动子的H3K27ac水平
2. 研究调控元件和靶基因表达背后的机制。
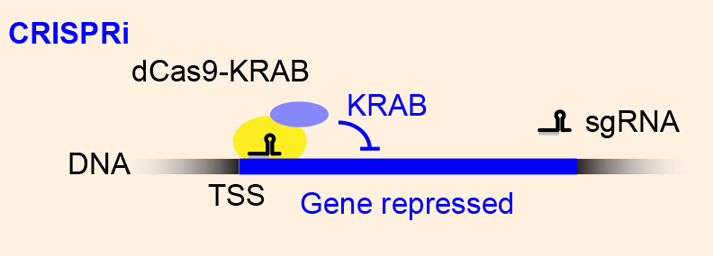
7. 研究内容:利用dCas9-KRAB使目的基因三甲基化,抑制目的基因
关键技术:
dCas9融合KRAB(Krüppel-associated box)后,作为一种CRISPRi,可以高度特异地诱导H3K9三甲基化(H3K9me3),从表观遗传修饰层面可逆地抑制靶基因的表达。对哺乳动物细胞系而言,dCas9-KRAB比单独的dCas9的抑制能力更优异,且对细胞生长影响小,是一种无毒且高效的基因沉默方法。
技术原理
KRAB结构域能募集内源染色质修饰复合物,包括KAP1共抑制复合物和核小体重构及去乙酰化酶(NuRD)复合物。KAPI复合物里含有一个组蛋白甲基化转移酶SERDB1,因而能够使组蛋白H3K9发生三甲基化、去乙酰化,是使用最广泛的抑制子,有着强大的抑制能力,常用于高通量基因筛选。
dCas9-KRAB融合蛋白对靶位点的作用是短暂的,当转录修饰蛋白去除后,可以重新建立转录,也就是可逆的。可设计gDNA结合到靶基因转录起始位点(TSS),抑制转录起始,沉默靶基因的表达。
技术应用
1. 瞬时转染到细胞,即可降低gDNA靶基因的表达。
2. 可靶向基因启动子区或转录起始位点,显著抑制基因。
3. 设计合适的gRNA,可降低miRNA等非编码RNA的表达。
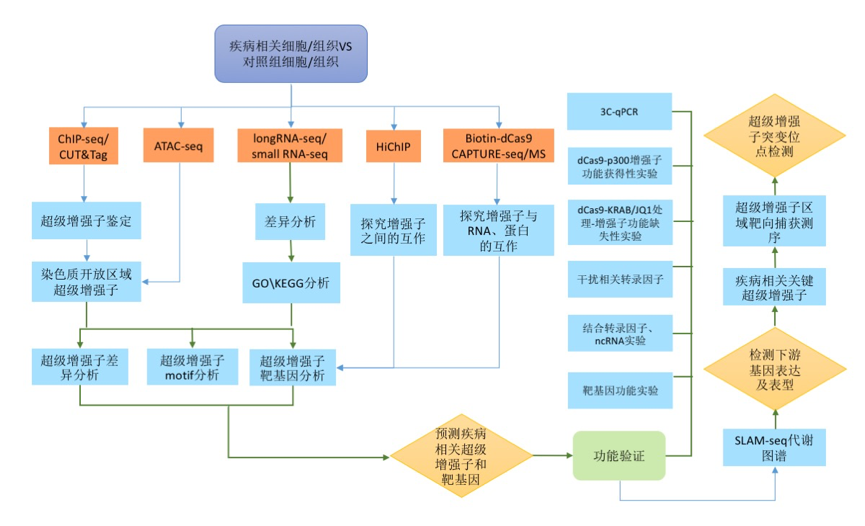
三、研究技术路径图
四、参考文献
[1] Blackwood EM, Kadonaga JT. Going the distance: acurrent view of enhancer action. Science, 1998, 281(5373):60–63.
[2] Bulger M, Groudine M. Functional and mechanistic diversity of distal transcription enhancers. Cell, 2011, 144(3): 327–339.
[3] Sun CB, Zhang X. Advance in the research on super enhancer. Hereditas(Beijing), 2016, 38(12): 1056–1068. 孙长斌, 张曦. 超级增强子研究迚展. 遗传, 2016, 38(12): 1056–1068.
[4] Levine M. Transcriptional enhancers in animal development and evolution. Curr Biol, 2010, 20(17): R754–763.
[5] Sengupta S, George RE. Super-enhancer-driven transcriptional dependencies in cancer. Trends Cancer, 2017, 3(4): 269–281.
[6] Ing-Simmons E, Seitan VC, Faure AJ, Flicek P, Carroll T, Dekker J, Fisher AG, Lenhard B, Merkenschlager M. Spatial enhancer clustering and regulation of enhancerproximal genes by cohesin. Genome Res, 2015, 25(4): 504–513.
[14] Lovén J, Hoke HA, Lin CY, Lau A, Orlando DA, Vakoc CR, Bradner JE, Lee TI, Young RA. Selective inhibition of tumor oncogenes by disruption of super-enhancers. Cell, 2013, 153(2): 320–334.
[15]中文综述:超级增强子在肿瘤研究中的进展
[16] Hnisz D, Schuijers J, Lin CY, Weintraub AS, Abraham BJ, Lee TI, Bradner JE, Young RA. Convergence of developmental and oncogenic signaling pathways at transcriptional super-enhancers. Mol Cell, 2015, 58(2): 362–370.
[17] Super-enhancers facilitate gene regulation by signaling pathways. Cancer Discov, 2015, 5(5): OF11.
[18] Wang Y, Zhang T, Kwiatkowski N, Abraham BJ, Lee TI, Xie S, Yuzugullu H, Von T, Li H, Lin Z, Stover DG, Lim E, Wang ZC, Iglehart JD, Young RA, Gray NS, Zhao JJ. CDK7-dependent transcriptional addiction in triple-negative breast cancer. Cell, 2015, 163(1): 174–186.
[19] Nabet B, P óB, Reyes JM, Shieh K, Lin CY, Will CM, Popovic R, Ezponda T, Bradner JE, Golden AA, Licht JD. Deregulation of the ras-erk signaling axis modulates the enhancer landscape. Cell Rep, 2015, 12(8):1300–1313.
[20] Adelman K, Lis JT. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nat Rev Genet, 2012, 13(10): 720–731.
[21] Wang H, Zang C, Taing L, Arnett KL, Wong YJ, Pear WS, Blacklow SC, Liu XS, Aster JC. NOTCH1-RBPJ complexes drive target gene expression through dynamic interactions with superenhancers. Proc Natl Acad Sci USA, 2014, 111(2): 705–710.
[22] Vahedi G, Kanno Y, Furumoto Y, et al. Super-enhancers delineate diSEaSE- associated regulatory nodes in T cells[J].Nature, 2015, 520(7548):558-562.
[23] Xiao M, Li J, Li W, et al. MicroRNAs activate gene transcription epigenetically as an enhancer trigger[J]. RNA Biol, 2017,14(10):1326-1334.
[24] Suzuki H I, Young R A, Sharp P A. Super-enhancer- mediated RNA processing revealed by integrative mi⁃ croRNA network analysis[J]. Cell, 2017,168(6):1000- 1014.
[25] Uversky VN. Intrinsically disordered proteins in overcro- wded milieu: membrane-less organelles, phase separation, and intrinsic disorder. Curr Opin Struct Biol, 2017, 44: 18–30.
[26] Brangwynne CP, Eckmann CR, Courson DS, Rybarska A, Hoege C, Gharakhani J, Jülicher F, Hyman AA. Germline P granules are liquid droplets that localize by controlled dissolution/condensation. Science, 2009, 324(5935): 1729– 1732.
[27] Brangwynne CP, Mitchison TJ, Hyman AA. Active liquid-like behavior of nucleoli determines their size and shape in xenopus laevis oocytes. Proc Natl Acad Sci USA, 2011, 108(11): 4334–4339.
[28] Hnisz D, Shrinivas K, Young RA, Chakraborty AK, Sharp PA. A phase separation model for transcriptional control. Cell, 2017, 169(1): 13–23.
[29] Sabari BR, Dall'Agnese A, Boija A, Klein IA, Coffey EL, Shrinivas K, Abraham BJ, Hannett NM, Zamudio AV, Manteiga JC, Li CH, Guo YE, Day DS, Schuijers J, Vasile E, Malik S, Hnisz D, Lee TI, Cisse II, Roeder RG, Sharp PA, Chakraborty AK, Young PA. Coactivator condensation at super-enhancers links phase separation and gene control. Science, 2018, 361(6400): eaar3958.
[30]中文综述:超级增强子在肿瘤研究中的进展
[31] Lovén J, Hoke HA, Lin CY, Lau A, Orlando DA, Vakoc CR, Bradner JE, Lee TI, Young RA. Selective inhibition of tumor oncogenes by disruption of super-enhancers. Cell, 2013, 153(2): 320–334.
[32] Sengupta D, Kannan A, Kern M, Moreno MA, Vural E, Stack B, Jr., Suen JY, Tackett AJ, Gao L. Disruption of BRD4 at H3K27Ac-enriched enhancer region correlates with decreased c-Myc expression in Merkel cell carcinoma. Epigenetics, 2015, 10(6): 460–466.
[33] Shin HY. Targeting super-enhancers for disease treatment and diagnosis. Mol Cells, 2018, 41(6): 506–514.
[34] Bai L, Zhou B, Yang CY, Ji J, McEachern D, Przybranowski S, Jiang H, Hu J, Xu F, Zhao Y, Liu L, Fernandez-Salas E, Xu J, Dou Y, Wen B, Sun D, Meagher J, Stuckey J, Hayes DF, Li S, Ellis MJ, Wang S. Targeted degradation of BET proteins in triple-negative breast cancer. Cancer Res, 2017, 77(9): 2476–2487.
[35] Odore E, Lokiec F, Cvitkovic E, Bekradda M, Herait P, Bourdel F, Kahatt C, Raffoux E, Stathis A, Thieblemont C, Quesnel B, Cunningham D, Riveiro ME, Rezai K. Phase I population pharmacokinetic assessment of the oral bromodomain inhibitor OTX015 in patients with haematologic malignancies. Clinical Pharmacokinetics, 2016,3(55): 397–405.
[36] Berthon C, Raffoux E, Thomas X, Vey N, Gomez-Roca C, Yee K, Taussig DC, Rezai K, Roumier C, Herait P, Kahatt C, Quesnel B, Michallet M, Recher C, Lokiec F, Preudhomme C, Dombret H. Bromodomain inhibitor OTX015 in patients with acute leukaemia: a dose- escalation, phase 1 study. The Lancet Haematology, 2016, 3 (4):186–195 .
[38] Chipumuro E, Marco E, Christensen CL, Kwiatkowski N, Zhang T, Hatheway CM, Abraham BJ, Sharma B, Yeung C, Altabef A, Perez-Atayde A, Wong KK, Yuan GC, Gray NS, Young RA, George RE. CDK7 inhibition suppresses super-enhancer-linked oncogenic transcription in MYCN- driven cancer. Cell, 2014, 159(5): 1126–1139.
[39] Wong RWJ, Ngoc PCT, Leong WZ, Yam AWY, Zhang T, Asamitsu K, Iida S, Okamoto T, Ueda R, Gray NS, Ishida T, Sanda T. Enhancer profiling identifies critical cancer genes and characterizes cell identity in adult T-cell leukemia. Blood, 2017, 130(21):2326–2338.
[40] Christensen CL, Kwiatkowski N, Abraham BJ, Carretero J, Al-Shahrour F, Zhang T, Chipumuro E, Herter-Sprie GS, Akbay EA, Altabef A, Zhang J, Shimamura T, Capelletti M, Reibel JB, Cavanaugh JD, Gao P, Liu Y, Michaelsen SR, Poulsen HS, Aref AR, Barbie DA, Bradner JE, George RE, Gray NS, Young RA, Wong KK. Targeting transcriptional addictions in small cell lung cancer with a covalent CDK7 inhibitor. Cancer Cell, 2014, 26(6): 909–922.
[41] Zhang T, Kwiatkowski N, Olson CM, Dixon-Clarke SE, Abraham BJ, Greifenberg AK, Ficarro SB, Elkins JM,
Liang Y, Hannett NM, Manz T, Hao M, Bartkowiak B, Greenleaf AL, Marto JA, Geyer M, Bullock AN, Young RA, Gray NS. Covalent targeting of remote cysteine residues to develop CDK12 and CDK13 inhibitors. Nat Chem Biol, 2016, 12(10):876–884.
[42] Kennedy AL, Vallurupalli M, Chen L, Crompton B, Cowley G, Vazquez F, Weir BA, Tsherniak A, Parasuraman S, Kim S, Alexe G, Stegmaier K. Functional, chemical genomic, and super-enhancer screening identify sensitivity to cyclin D1/CDK4 pathway inhibition in Ewing sarcoma. Oncotarget, 2015, 6(30): 30178–30193.

