SLAM-seq动态转录组(RNA代谢)

项目简介
SLAM-seq服务(RNA代谢测序)
通过高通量测序技术绘制基因表达图谱,可以揭示 RNA 在一个稳定状态下质和量上的变化,但是这仅仅是基因表达的一个“快照”,无法观察到RNA转录、加工和降解的细胞内动态过程。
SLAM-seq,即thiol(sh)-linked alkylation for the metabolic sequencing of RNA,可以理解为“s4U烷基化RNA代谢测序技术”,可观察转录的实时情况和特征。通过研发成熟的代谢RNA标记方法和高通量测序技术,SLAM-seq能检测整合到总RNA中的核苷类似物4-硫尿苷(s4U),绘制RNA聚合酶II依赖性的基因表达动态,是一种操作性强、稳定、可靠的基因表达调控机制研究方法。
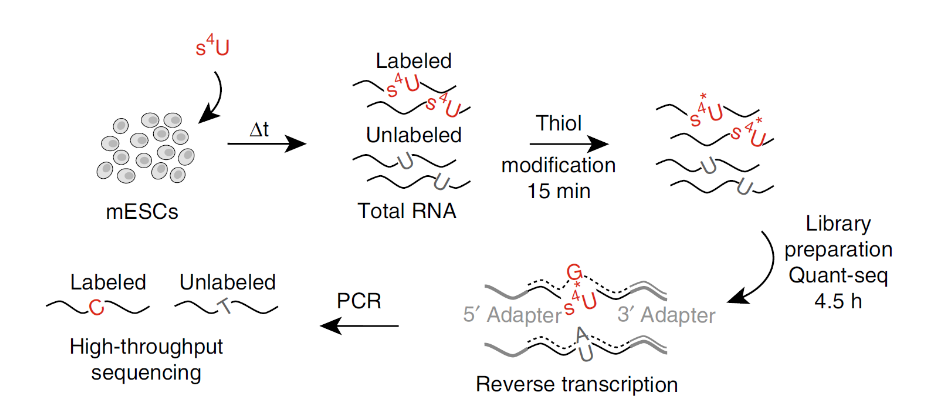
SLAM-seq技术流程图
技术原理
向合成中的RNA链中掺入4-thiouridine (S4U),使原本的碱基T被S4U修饰,从而在反转录中被错认为C,导致原本应该为碱基A的位置错配成为G。通过测序分析这些错配的G,就可以对新合成mRNA或其他longRNA进行直接的定量。
应用与分析
技术应用
1. 检测基因转录水平的变化
2. 研究RNA半衰期、稳定性
3. 深入研究基因转录调控机制
4. RNA修饰如m6A、m1A、ac4C等作用机制的研究
分析内容
1. 序列比对
2. 文库质控
3. Mapped Reads过滤
4. 背景T>C SNP校正
5. mRNA或longRNA*异表达分析
6. 靶基因GO富集分析
7. 靶基因KEGG富集分析
8. mRNA或longRNA转录本水平T>C counts计数与表达量化
9. mRNA或longRNA转录本水平Total reads计数与表达量化
10. mRNA或longRNA稳定性分析
11. mRNA或longRNA半衰期计算
送样要求
需要在培养的活细胞中掺入S4U碱基,可免费提供
1x10^6
细胞数量
人、大小鼠
样本物种
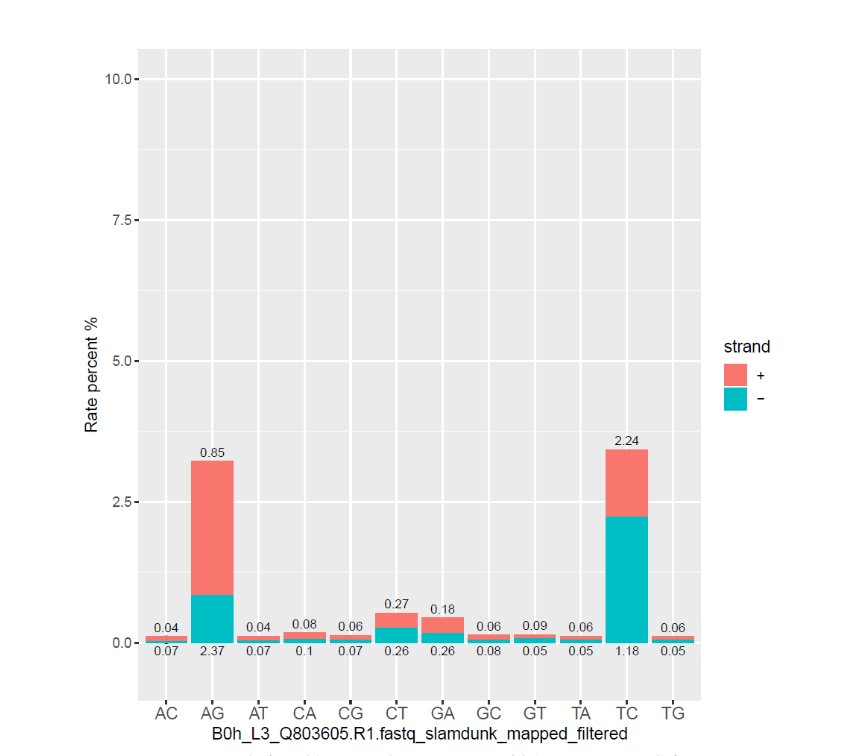
图2. Slam-seq建库质控(检测T>C和A>G转化是否明显升高)
表观生物实测数据

图3. 所有转录本整体稳定性分析

图4. 不同类型转录本稳定性比较分析
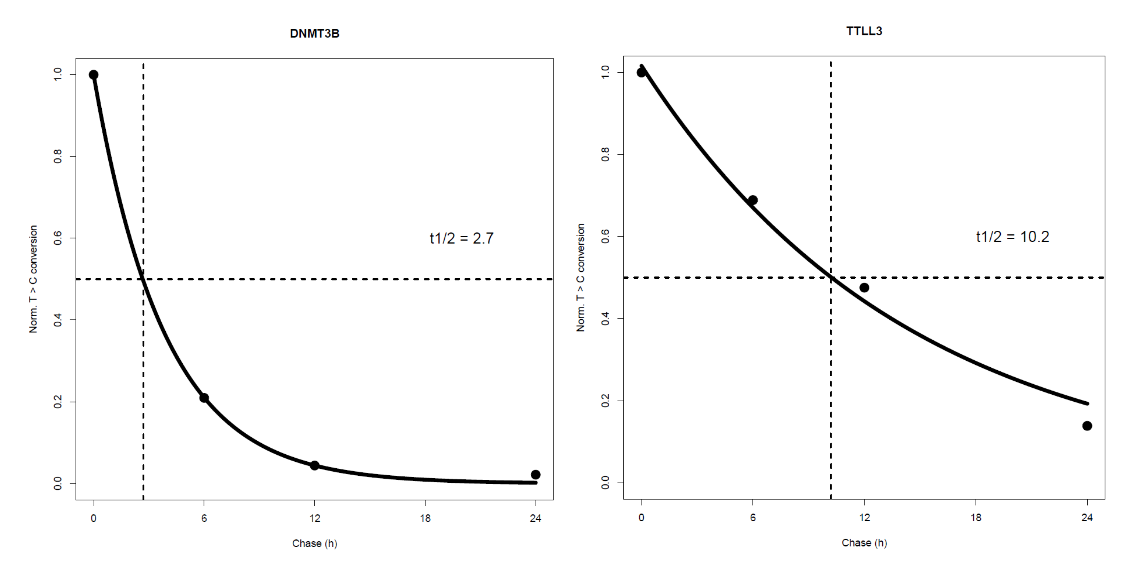
图5. 单个基因的稳定性分析
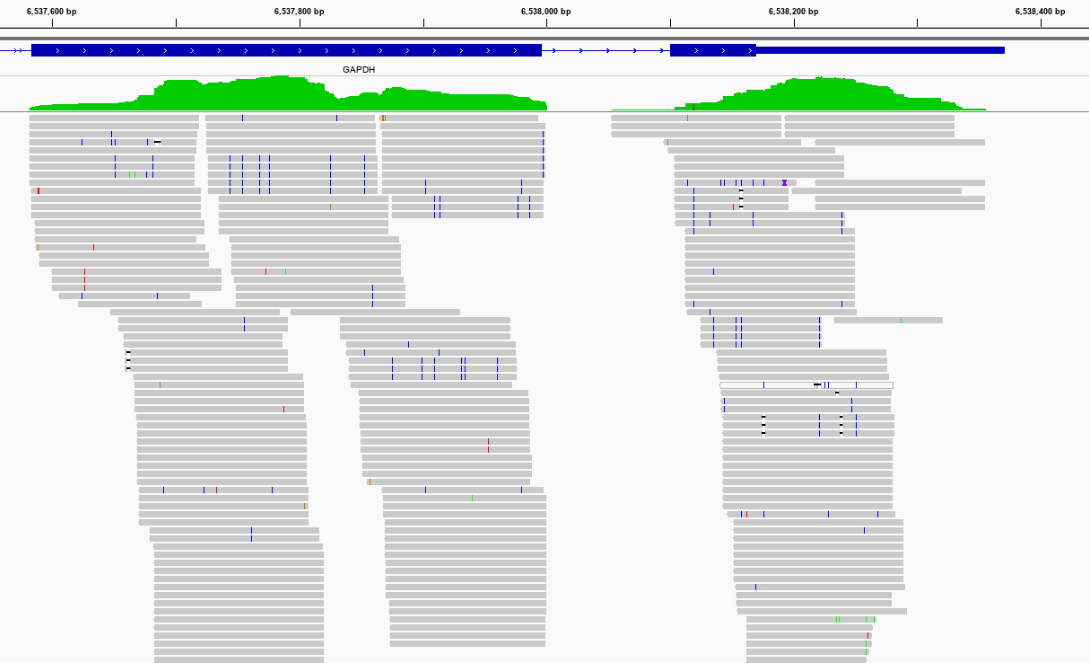
图6. 单个基因T>C突变查看(蓝色为T>C突变的碱基)

图7. 多组学分析揭示H3K9la调控机制
客户文章
1、Adv Sci:组蛋白H3K9乳酰化通过LUC7L2介导的MLH1内含子保留促进胶质母细胞瘤对替莫唑胺的耐药性²
这项研究揭示了组蛋白H3K9乳酰化修饰(H3K9la)在胶质母细胞瘤(GBM)替莫唑胺(TMZ)耐药中的作用机制。研究发现H3K9la通过激活LUC7L2转录,进而介导MLH1内含子7滞留,最终导致GBM产生TMZ耐药。同时研究者还发现临床上用于治疗癫痫的药物stiripentol可以通过抑制乳酰化来增强TMZ的敏感性。
在研究方法上,研究团队采用了多组学联合分析策略。主要使用了CUT&Tag测序鉴定H3K9la的全基因组结合位点、SLAM-seq定量新合成的mRNA、RNA-seq分析转录组变化,并将这些数据与CGGA数据库进行整合分析。CUT&Tag分析显示H3K9la在LUC7L2启动子区域富集,而SLAM-seq与RNA-seq联合分析确定了14个潜在靶基因。多组学整合分析进一步证实LUC7L2是H3K9la的重要靶基因,其表达受H3K9la调控。RNA-seq的KEGG和GO分析则显示错配修复通路显著富集。
为验证这些发现,研究团队进行了一系列关键实验。通过ChIP-qPCR验证了H3K9la与LUC7L2启动子的结合,RIP和CLIP实验证实了LUC7L2与MLH1 pre-mRNA的结合。体内外实验则证实stiripentol可以通过抑制乳酰化增强TMZ的治疗效果。
这项研究不仅从表观遗传修饰、RNA剪接调控等多个层面深入阐明了GBM TMZ耐药的新机制,还提出了一种潜在的联合治疗策略,具有重要的临床转化价值。研究通过系统的多组学分析方法为理解和克服GBM的TMZ耐药提供了新的视角和治疗思路。

图8. SLAM-seq分析结果
2、Circ Res:NAT10通过ac4C介导的转录组调控参与心脏重塑³
这项研究系统揭示了NAT10介导的RNA ac4C修饰在心脏重构中的作用机制。研究采用了多组学整合分析策略,包括acRIP-seq用于鉴定ac4C修饰位点和修饰转录本、SLAM-seq分析转录本稳定性和半衰期、Ribo-seq评估翻译效率以及RNA-seq分析基因表达水平。通过acRIP-seq分析展示了ac4C修饰在人类和小鼠心脏重构过程中的变化模式及其富集的生物学过程。SLAM-seq分析表明NAT10过表达显著延长了mRNA的半衰期,在NMCMs中从8.75h延长至11.11h,在NMCFs中从3.36h延长至4.98h,特别是对ac4C(+)转录本的影响更为显著。研究通过多组学数据整合分析,在NMCMs中整合acRIP-seq、RNA-seq和SLAM-seq数据鉴定出62个靶基因,在NMCFs中鉴定出70个靶基因,并通过整合Ribo-seq、acRIP-seq和RNA-seq数据鉴定出7个关键基因。研究发现NAT10通过增加CD47和ROCK2 mRNA的ac4C修饰水平来提高其稳定性和翻译效率,从而促进心脏重构。特别是,使用NAT10抑制剂Remodelin可以通过降低ac4C水平来减轻心脏重构,提示了潜在的治疗策略。这项研究首次系统地展示了ac4C修饰在心脏重构中的调控作用,为心力衰竭的治疗提供了新的靶点。
参考文献
[1]Herzog VA, Reichholf B, Neumann T, et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nat Methods. 2017;14(12):1198-1204.
[2]Yue Q, Wang Z, Shen Y, et al. Histone H3K9 Lactylation Confers Temozolomide Resistance in Glioblastoma via LUC7L2-Mediated MLH1 Intron Retention. Adv Sci (Weinh). 2024;11(19):e2309290.
[3]Shi J, Yang C, Zhang J, et al. NAT10 Is Involved in Cardiac Remodeling Through ac4C-Mediated Transcriptomic Regulation. Circ Res. 2023;133(12):989-1002.
