Cell案例| lncRNA研究无从下手?你试过这些方法吗
在刚过去不久的6月底,Cell 期刊同时发表了两篇研究lncRNA TERRA的文章,一篇是关于TERRA如何实现优先修复短端粒的机制,国内众多生物媒体均对此研究进行了报道。今天,Epi老师和大家谈的是和它一起发表的另一篇TERRA文章。
这篇标题为 “TERRA RNA Antagonizes ATRX and Protects Telomeres” 的文章,主要就是发现了TERRA不仅与端粒结合,还与端粒以外的染色体结合,并且影响附近基因的转录,保护端粒完整性。这么说来好像很简单,RNA和染色质的相互作用,做做FISH,做做pull down不就好了?But,咱们的研究对象可是TERRA,TERRA有着高度重复的序列,所以不能用普通的覆瓦式(tiling)探针,一般来说ChIRP用的就是一组tiling核苷酸探针;特异位点TTAGGG在所有染色体末端都存在,又会很容易把端粒DNA拉下来而不是想要的TERRA RNA,造成高背景。那么作者是怎么解决这些技术问题的呢?我们一起来看一下吧。
1. CHIRT-seq
作者将现有的ChIRP和CHART (capture hybridization analysis of RNA targets) 技术结合,发明了一项新技术并命名为CHIRT,用以捕获染色体上的TERRA结合位点:
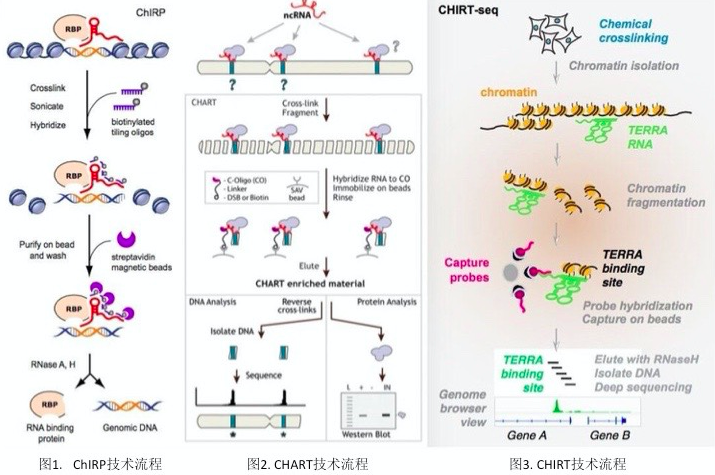
图中分别是三种技术的流程图。ChIRP使用的是一组全链覆盖式探针(又称平铺探针);CHART使用的互补核酸探针数量较少,序列较长。作者将两者结合,并做了优化:在CHIRT中使用了一个单独的核酸探针,靶向端粒重复序列,与多探针相比,脱靶的可能性减少;剪切强度减弱,保护lncRNA;将RNaseH洗脱使用的buffer改为NP40,保护RNaseH活性;建库前设置spike-in对照DNA,作为内参。更多的技术对比可见本文末延伸阅读。
通过CHIRT提取到TERRA基因结合位点,进行测序,再配合CEAS软件分析,TERRA RNA FISH验证,作者证实了他的猜想:TERRA不仅仅与端粒结合,还和邻近的其他基因结合。
2. LNA GapmeR
除了CHIRT,本文还有一个技术亮点。在此之前,对TERRA的研究一直苦于缺乏合适的工具,删除TERRA的效果也不是很稳定,因为TERRA是从必需的端粒结构转录而来的,无法进行基因敲除;而且TERRA高度重复,序列还与端粒相似,很难使用RNA干扰方法。
于是作者使用了特殊锁核酸 (LNA) 化学修饰的,并添加了gapmer设计的单链ASO,使之更适合用于RNaseH介导的TERRA转录本体内降解。切割TERRA后做RT-qPCR和RNA-seq,发现TERRA对它的靶基因有负调节作用。解决了这两个技术难题,接下来各种实验都可以顺利开展了。

A. Northen blot,B.RNA FISH,检测LNA gapmer切除效果;C & D,将RNA-seq数据做成累积频数图 (C) 和Bland-Altman散点图 (D);E.切割TERRA后进行RT-qPCR。
3.相互作用蛋白研究
不严谨不Cell,作者还嫌一般的蛋白质组研究常常提取到hnRNP等RNA结合蛋白,所以用了iDRIP (identification of direct RNA interacting proteins) 技术,做定量质谱,获得TERRA蛋白相互作用组 (interactome),发现了134个新的TERRA蛋白伴侣,涉及很多不同的细胞过程。
作者将自己的CHIRT数据与前人的ESC ChIP数据做比较,发现ATRX与TERRA的结合浓度最高,顺理成章地选了ATRX作为研究对象,进行了更多的实验:
a. ChIP、siRNA、metagene分析显示,TERRA的结合对ATRX的靶基因有正调节作用,与ATRX的作用是相反的,两者是拮抗的。
b. EMSA检测到TERRA和ATRX直接相互作用;ATRX免疫染色和端粒DNA FISH发现TERRA通过竞争结合到端粒DNA,调控ATRX的端粒定位。
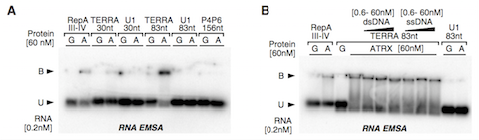
RNA EMSA。A图,ATRX蛋白使TERRA RNA迁移,表明两者直接结合。B图,添加双链或单链的端粒重复序列,都没有竞争掉ATRX与TERRA的相互作用。 B, 结合探针(shifted);U, 未结合。RepA, 阳性对照;U1和P4P6, 阴性对照;G, GST,对照;A, ATRX蛋白.
c. RNA-seq和RT-qPCR显示,切割TERRA后,端粒酶RNA元件Terc增加为2倍;pull down、RNA FISH证明TERRA与Terc体内结合。
d. 切割TERRA,TIF(telomere-induced DNA damage foci,端粒功能异常的时候就会出现)增加,证明TERRA调控端粒酶活性,维持端粒完整性。
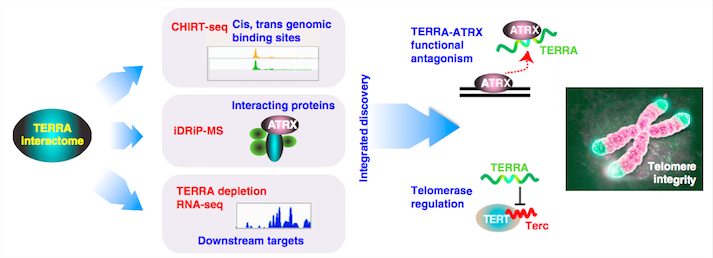
至此,文章就完美地结束了,其中有很多实验技术咱们都可以参考看看能否解决自己研究lncRNA与蛋白互作时遇到的问题,比如ChIRP-seq、LNA GapmeR、iDRiP。这篇文章发掘了TERRA修复端粒以外的新功能——调控邻近的其他基因转录,还与很多蛋白结合,为TERRA的研究开辟了更多的道路。
原文:Chu, et al. TERRA RNA Antagonizes ATRX and Protects Telomeres. Cell. 2017.06.017.
延伸阅读:ChIRP与CHART技术比较
相同点:
ChIRP与CHART都是研究RNA的技术,使用探针沉淀染色质,揭示lncRNA在基因组上的结合位点。操作方面,均是交联后用生物素(或链酶亲和素)反义探针捕获,用链霉亲和素磁珠回收,将沉淀的蛋白、RNA、DNA分别收集,后两者可以继续进行RT-PCR或高通量测序。ChIRP提取到的蛋白可以进行质谱分析。
不同点:
1. 探针的大小与设计不同。ChIRP能覆盖lncRNA序列的全长,靶向所有潜在的位点,探针序列较短(~20bp),易于合成,可以减少脱靶;而CHART通过RNaseH试验寻找合适的靶标位点,设计探针(18~28bp)。
2. 交联方法不同。ChIRP使用的是戊二醛,CHART使用的是甲醛,已证明较大的交联试剂(戊二醛)效率更高。
|
|
优点 |
缺点 |
|
CHART |
使用更少量的探针,减少背景信号 |
交联使用甲醛溶液,效率不高 |
|
不需要已知特异结构域 |
探针合成需要经过RNaseH试验,耗费时间 |
|
|
ChIRP |
揭示全基因组范围的lncRNA DNA结合位点 |
与dChIRP相比,信噪比较高 |
|
需要已知特异结构域,探针覆盖lncRNA全长 |
不能揭示特异的lncRNA结构域 |
|
|
探针短,合成方便,脱靶效应少 |
|
- - - 推荐阅读 - - -

【推荐】RNA甲基化修饰(m6A)研究技术及方案设计
RNA甲基化是表观遗传学内容的重要内容之一,其中m6A(N6-methyladenosine,6-甲基腺嘌呤)较为常见的一种修饰方式。m6A是一种动态可逆的修饰方式,在转录后调控中发挥作用,其在调控基因表达、剪接、RNA 编辑、RNA 稳定性、控制mRNA寿命和降解、介导环状RNA翻译[1]等方面扮演重要角色,具有重要的研究意义.
2017-06-29

【推荐】肿瘤相关超级增强子的研究方案及应用前景
增强子是调控细胞基因时空表达关键的顺式作用元件。2013年,Richard A. Young 实验室基于当时增强子的研究,提出了超级增强子(Super-enhancers, SEs)概念.
2017-06-22

如何快速发现肿瘤特异性外泌体RNA分子标志物
在当前精准医学的大背景下,液体活检作为一项重要的创新技术,有望在癌症早期筛查与诊断、治疗方案指导、疗效评估及预后测评领域发挥作用,具有极高的临床应用价值和市场前
2017-03-16
