备战2019NSFC| 肿瘤相关Lncrna深度分析与功能预测
TCGA(The Cancer Genome Atlas),即癌症基因组图谱项目,始于2005年,由美国政府出资,美国国家癌症研究所和美国人类基因组研究所共同监督,旨在应用高通量的基因组分析技术,以帮助人们对癌症有个更好的认知,从而提高对于癌症的预防、诊断和治疗能力。目前,利用TCGA数据,进行深度数据挖掘,再设计实验验证而发表的文章越来越多,其中包括癌症亚型、driver mutation、假基因等新型研究领域。

数字展示出TCGA数据之丰富
但是TCGA数据库中只提供数据的下载,后续的分析任务才是更重要的部分。表观生物通过整合挖掘TCGA 7,332套RNA-seq数据、拷贝数变化数据及临床相关数据,将15,931个高可信度的长非编码RNA(下称lncRNA)与18种肿瘤进行关联分析,鉴定了异常表达的lncRNA,发生频繁拷贝数变化的lncRNA以及显著预后相关的lncRNA。同时,表观生物评估了与这些lncRNA共表达的蛋白编码基因,并预测了其可能的生物学功能。这些数据有望为研究癌症相关lncRNA奠定坚实基础和提升研究效率!
表1. 涉及肿瘤类型及分析的样本数量
|
类型 |
肿瘤样本 数量 |
正常对照数量 |
|
THCA(甲状腺癌) |
504 |
59 |
|
CHOL(胆管癌) |
36 |
9 |
|
UCEC(子宫内膜癌) |
180 |
23 |
|
READ(直肠癌) |
92 |
10 |
|
HNSC(头颈癌) |
518 |
44 |
|
COAD(结肠癌) |
287 |
41 |
|
STAD(胃癌) |
413 |
36 |
|
LIHC(肝癌) |
369 |
50 |
|
KICH(肾嫌色细胞癌) |
66 |
25 |
|
PRAD(前列腺癌) |
494 |
51 |
|
LUAD(肺腺癌) |
513 |
59 |
|
LUSC(肺鳞癌) |
498 |
50 |
|
BRCA(乳腺癌) |
1092 |
113 |
|
KIRP(肾乳头状细胞癌) |
288 |
32 |
|
ESCA(食管癌) |
181 |
13 |
|
KIRC(肾透明细胞癌) |
530 |
72 |
|
BLCA(膀胱癌) |
407 |
19 |
|
GBM(胶质瘤) |
153 |
5 |
数据应用
1. 快速获取在肿瘤中异常表达及存在拷贝数变异的lncRNA,为验证肿瘤相关lncRNA奠定基础;
2. 为相关研究论文的发表提供重要的数据支撑;
3. 可作为申请课题的基础工作,降低前期投入的成本;
4. 为lncRN的功能高通量筛选结果提供参照,指导精确锁定肿瘤相关的lncRNA。
技术优势
1. 国内首个TCGA数据挖掘服务!
2. 表观生物承诺,每个癌种的全部相关数据均只出售一次,售完即止;18组肿瘤的数据,先到先得!
分析内容
1. 差异表达分析
我们通过利用Limma包中的voom算法对每种癌症类型中差异表达的长链非编码RNA进行鉴定,统计显著的长链非编码RNA被定义为倍数变化大于或等于2倍,假阳性发现率(FDR)小于0.05。以胃癌为例,我们用上述的方法分析了15931个长链非编码RNA在413例肿瘤样本与36例癌旁组织样本之间的表达水平,发现2581个长链非编码RNA在癌与癌旁组织之间显著差异表达(见图1)。例如,lnc_ENSG00000236081和lnc_ENSG00000274979在胃癌中显著上调(见图2)。
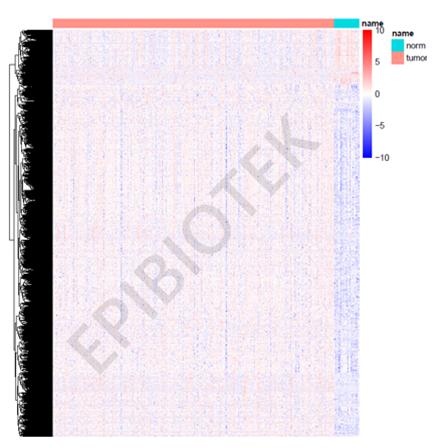
图1 热图显示了在胃癌中显著失调的长链非编码RNA
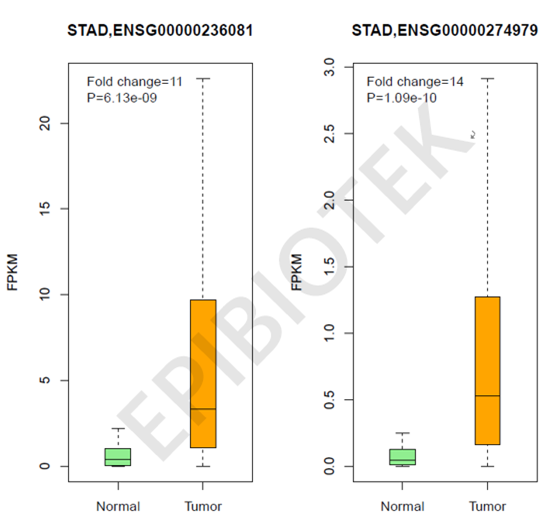
图2 箱线图显示了lnc_ENSG00000236081和lnc_ENSG00000274979在胃癌中显著上调
2. 拷贝数变化分析
基于已经鉴定的显著失调的长链非编码RNA,我们进一步探索了拷贝数变化对其变化的影响。通过整合分析拷贝数变化和表达数据,我们发现一批显著失调的长链非编码RNA的DNA拷贝数发生了高频的变化。例如,lnc_ENSG00000274979在胃癌中均发生了频繁的扩增,且明显与它们升高的表达显著相关(见图3)。
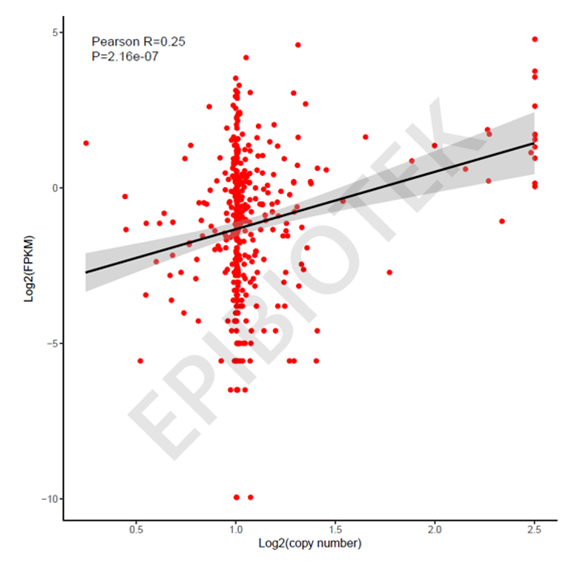
图3 lnc_ENSG00000274979在胃癌中扩增,且表达与拷贝数显著正相关
3. 生存分析
我们也基于临床数据,评估了这些差异表达的长链非编码RNA与病人生存之间的关联。基于多因素cox回归模型(以年龄和性别作为协变量),我们鉴定了一批长链非编码RNA与病人生存显著相关。例如,高表达lnc_ENSG00000223949的胃癌病人比那些低表达的病人拥有更短的生存时间(见图4)。
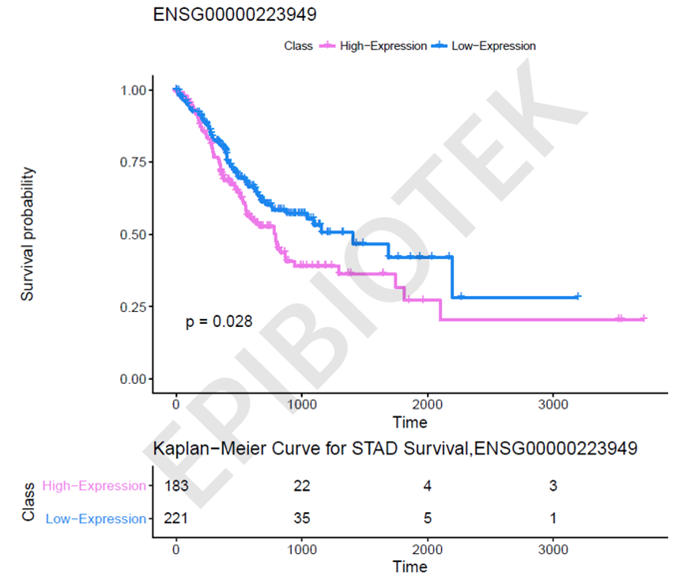
图4 lnc_ENSG00000223949的表达与胃癌病人的生存显著相关
3. 鉴定与lncRNA表达相关的蛋白编码基因
为了研究lncRNA与蛋白编码基因之间的调控关系,我们分析了lncRNA与近20000个蛋白编码基因之间的表达相关性。以经典的癌相关长非编码RNA PVT1为例(在胃癌中显著上调),我们鉴定了800多个与PVT1显著相关的蛋白编码基因(FDR<0.05)。图5显示了与PVT1相关的前50个蛋白编码基因,其中包括了已报道的PVT1与MYC的调控关系。基于表达相关性的研究,将有助于我们发现lncRNA介导的调控关系。
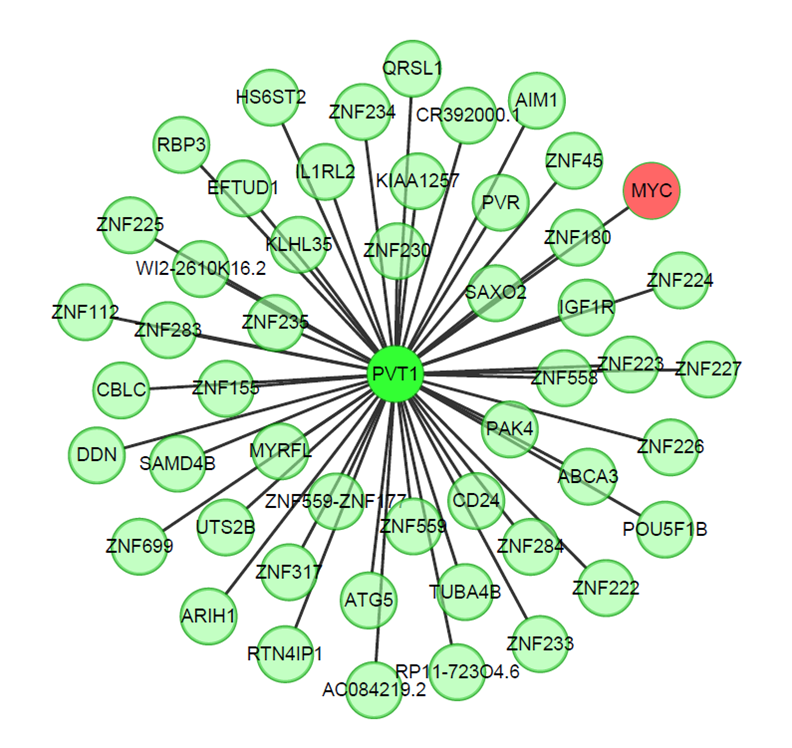
图5 与PVT1显著相关的前50个蛋白编码基因
4. lncRNA功能预测
通过分析与lncRNA显著相关的蛋白编码基因集,我们能够推断lncRNA可能的功能。继续以PVT1为例,通过对与之相关的蛋白编码基因集进行GO分析,我们发现这些基因显著富集在细胞周期,DNA复制以及修复相关的生物学过程中。同时,KEGG Pathway分析也显示了这些基因显著富集在细胞周期相关的信号通路中,这些结果与已报道的PVT1的功能相一致。
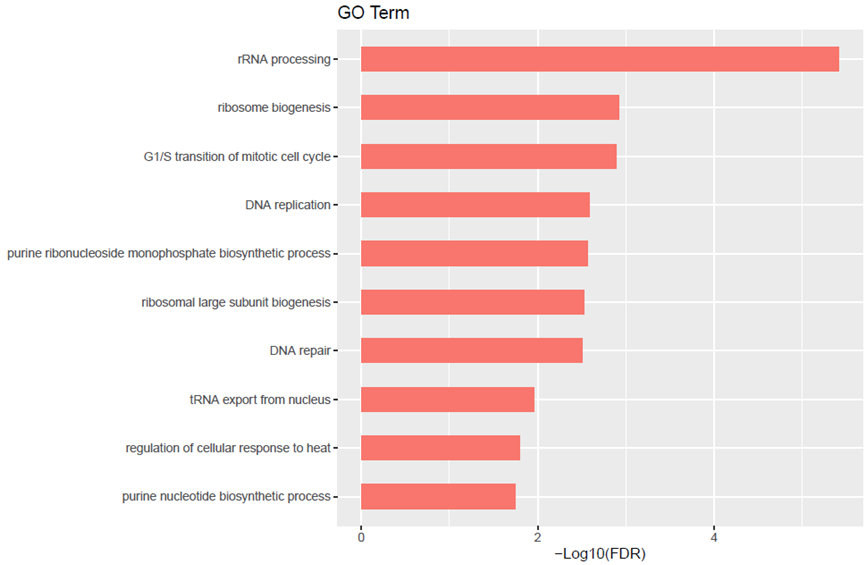
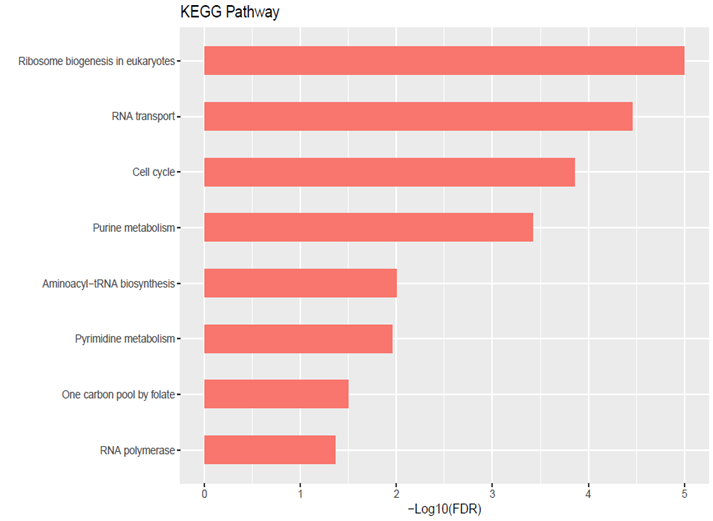
图6 GO和KEGG Pathway分析
注:客户可通过分析结果总表挑选最多10个重点目标的长链非编码RNA分子,由表观生物进行差异表达箱线图、表达与拷贝数变化关系图、表达与生存关系图,共表达网络图以及GO和KEGG分析图绘制。
- - - 推荐阅读 - - -
9折优惠| 2018全国细胞外囊泡研究大会,ISEV首次走进中国
由中国研究型医院学会细胞外囊泡研究与应用专委会(CRHA-CSEV)主办的“2018全国细胞外囊泡功能与临床应用进展学术会议暨第二届中国研究型医院学会细胞外囊泡研究与应用专业委员会学术会议”(简
2018-10-30

首届医学表观遗传学前沿技术培训班圆满落幕!
本届培训班浓厚的学术氛围、干货满满的课程安排,几位讲者专业严谨的学术态度、细致认真的技术讲解、风趣幽默的讲课过程以及对于中国生物医学基础科研现状的深刻理解,得到了参会学员的一致好评。 培训班现场座无虚席,直到培训结束,都是人头济济的 本次培训班邀请到了中科院广州生物医药与健康研究院姚红杰研
2018-10-16

喜讯!广州表观生物与上海烈冰科技签订战略合作协议,共建表观医学云平台
广州表观生物科技有限公司宣布与上海烈冰科技签订战略合作协议,以NovelBrain云计算平台为基础,构建表观医学云平台(EpimedCloud)。
2018-09-21
