Ribo-seq核糖体印迹测序服务
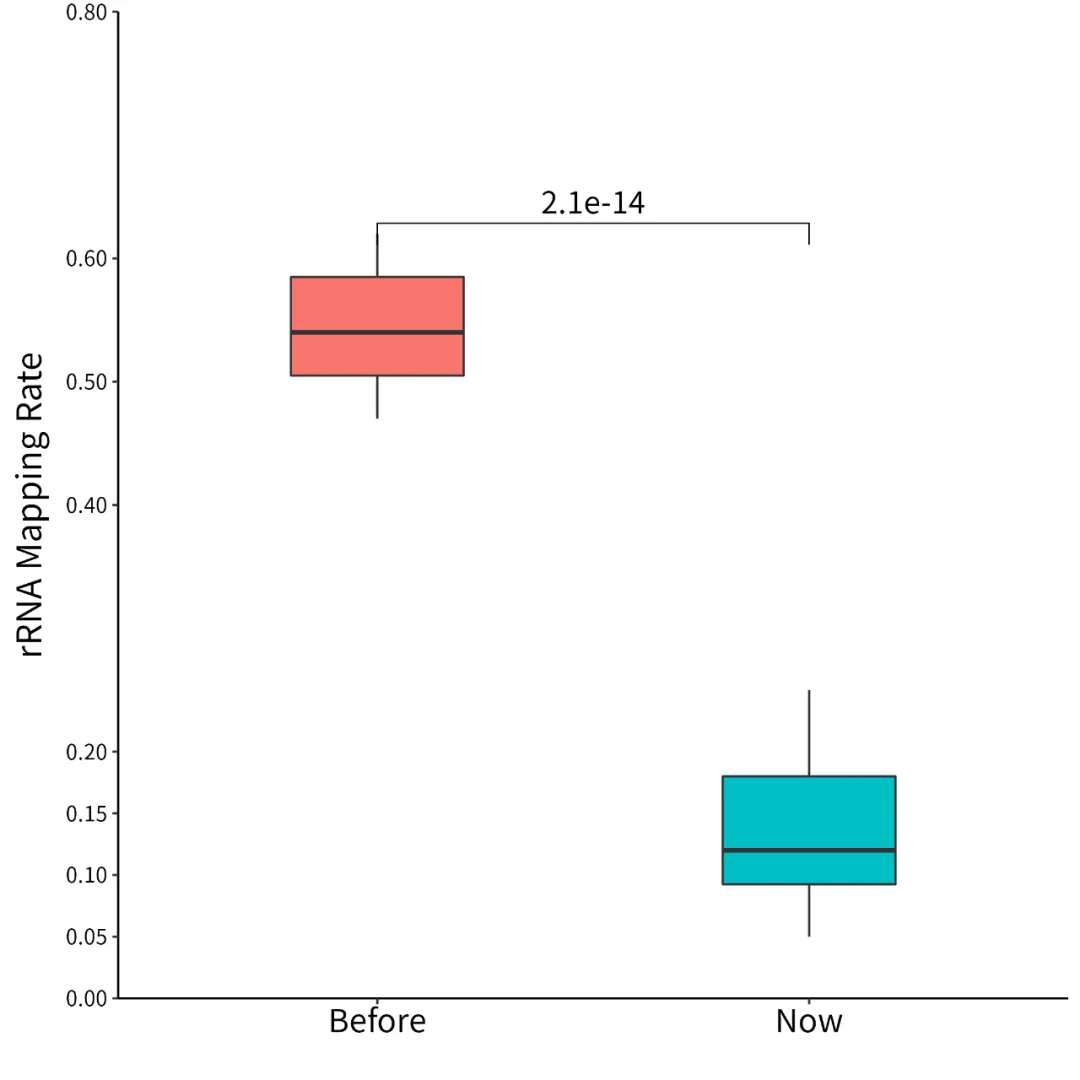
表观生物Ribo-seq实测数据,rRNA平均占比从55%降至14%
项目简介
Ribo-seq (Ribosome profiling),即核糖体印迹测序技术,系由 Weissman 课题组于 2009 年首次发表的翻译组学研究技术[1]。利用 Ribo-seq,研究者能从基因组水平检测蛋白质的翻译状况,获得全面的、高质量的蛋白质翻译速度情况,了解蛋白质表达情况及其丰度,还能直接对翻译过程进行研究。
表观生物通过技术优化,将Ribo-seq数据的rRNA占比降至平均14%,最低达到5%!测序效率更高,质量更优,获得的翻译组信息更准确,助力客户获得更多有价值的研究结果。
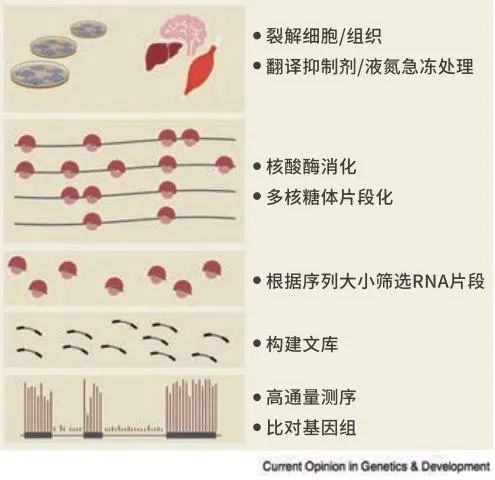
Ribo-seq 技术流程[2]
技术原理
利用核酸酶降解没有核糖体覆盖的 mRNA 片段,高通量测序获得 ribosome footprints,及核糖体分布的位置信息。而 footprints 密度越高的位点,表明翻译延长速率越慢。
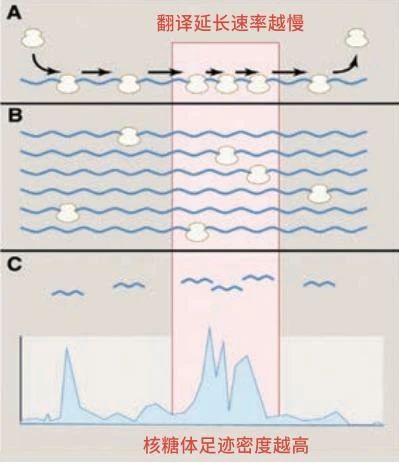
Ribo-seq 检测翻译延长速率的原理[2]
技术应用
联合 longRNA-seq,研究转录本上正在进行的翻译情况,深入了解基因调控最重要层面:
1. 了解转录本上的核糖体分布、翻译活性
2. 推测翻译起始位点、ORF 位置
3. 确定蛋白翻译效率
4. 探究翻译调控和基因表达情况
5. 鉴定新蛋白 / 新短肽
送样要求
样本物种:仅限人、大小鼠,其他物种需评估
样本类型:
1. 细胞,≥ 1×107 个细胞 / 样本
2. 组织,≥ 50mg/ 样本
样品分组
1. 常规要求至少 2 组样品,包括对照组和实验组(临床样本为正常人组和患者组)
2. 每个样品均进行 Ribo-seq 和 longRNA-seq
3. 样本数建议:3 VS 3
分析内容
Ribo-seq 组基本分析
1. 原始数据过滤与测序质量评估
2. rRNA、tRNA 去除,reads 长度过滤
3. 参考基因组比对、参考转录组比对
4. Ribosome 实验质量质控(reads长度分布、RFs分布、3nt周期性特征)
5. 差异翻译基因分析
6. 差异翻译基因热图(仅限有生物学重复)
7. 差异翻译基因GO KEGG分析
8. 基因翻译效率计算(需要用 mRNA-seq 或 longRNA-seq)
9. 差异翻译效率分析
10. 差异翻译效率基因GO KEGG分析
11. 密码子使用频率分析
12. ORF(开放阅读框)预测
13. 翻译暂停分析
对照 RNA-seq 或 longRNA-seq 分析
1. 测序原始 reads 去接头,质量 控制
2. 参考基因组比对
3. 基因表达定量分析
4. 基因注释
5. 差异表达分析
6. 差异基因热图绘制 (仅限有生物学重复)
7. 差异基因GOKEGG分析
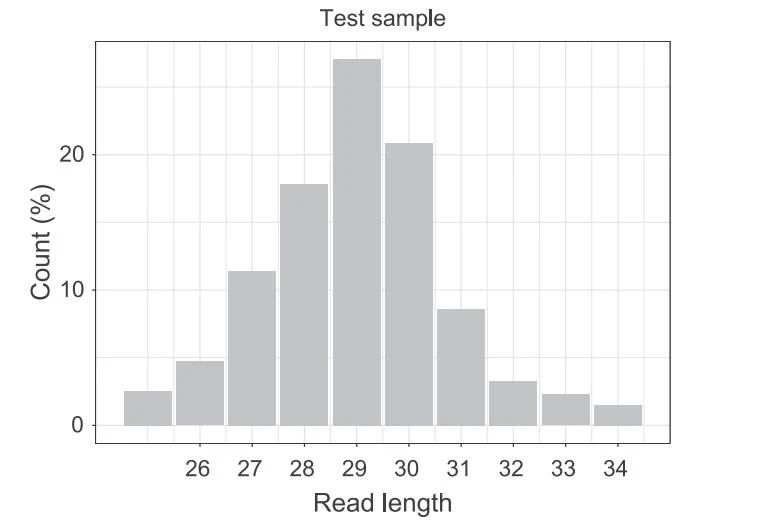
图1. Ribo-seq 比对到基因组的 read 的长度分布情况
实测数据及分析结果示例 1
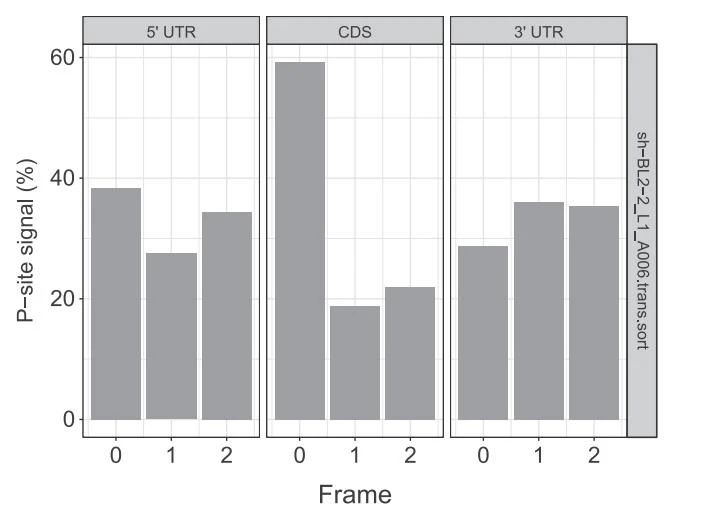
图2. P-site 信号在 5'UTR、CDS、3'UTR 区间的分布
实测数据及分析结果示例 2
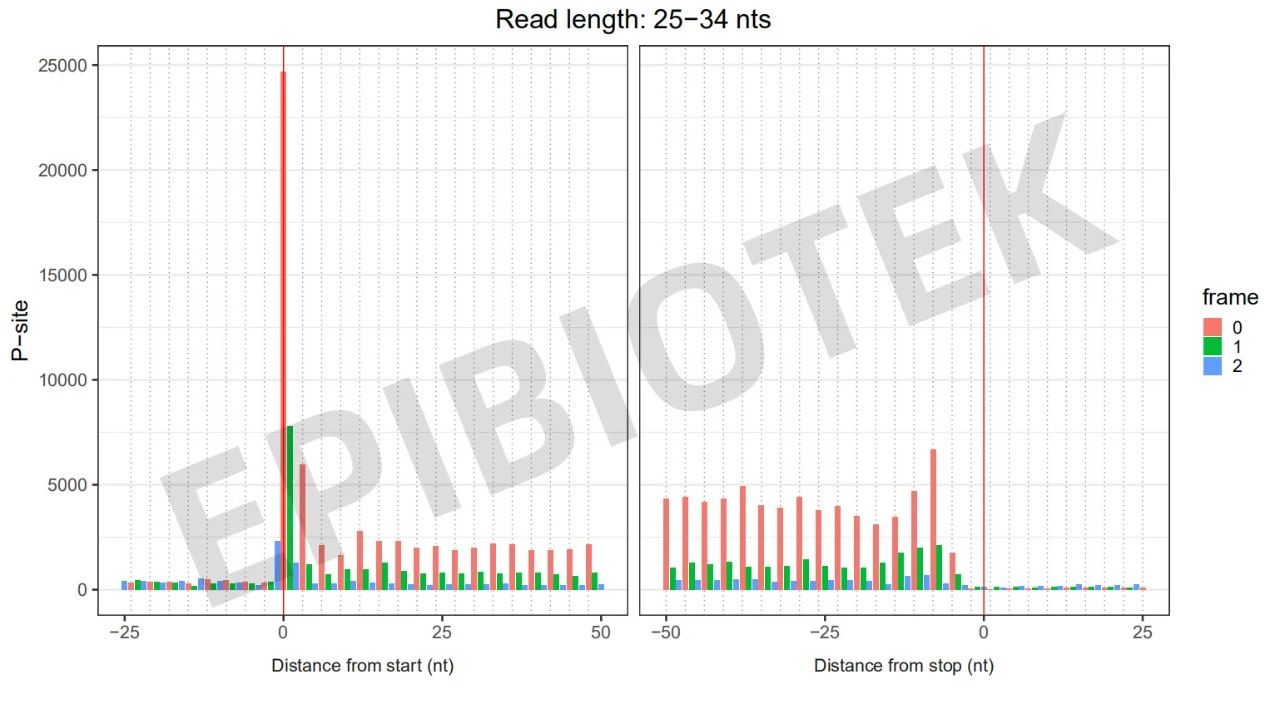
图3. 细胞沉淀样本的3nt质控图
实测数据及分析结果示例 3
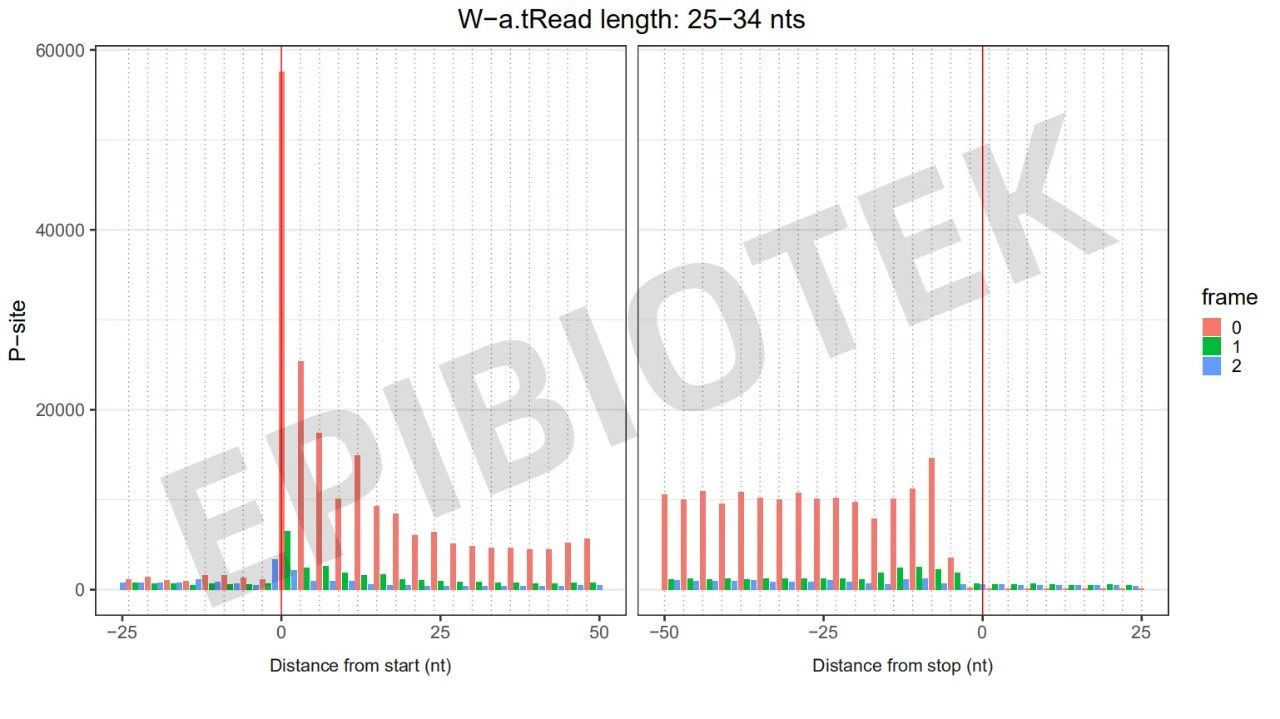
图4. 贴壁活细胞样本的3nt质控图
实测数据及分析结果示例 4
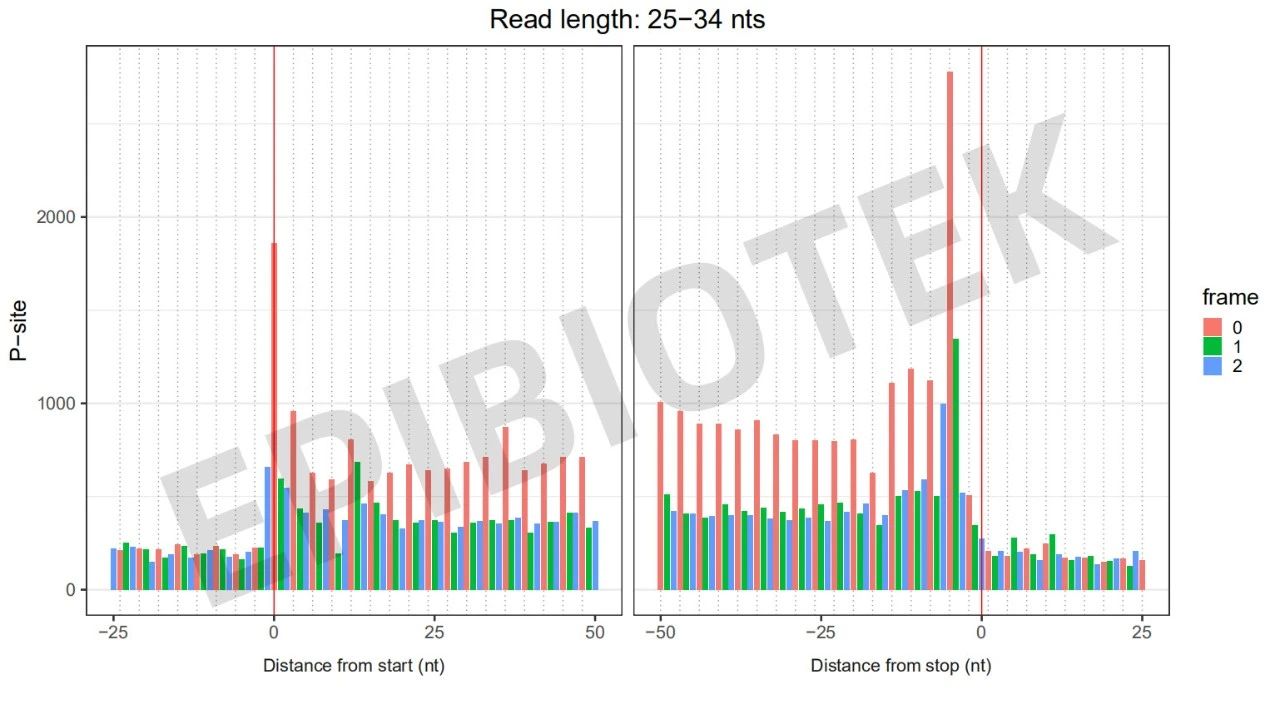
图5. 组织样本的3nt质控图
实测数据及分析结果示例 5
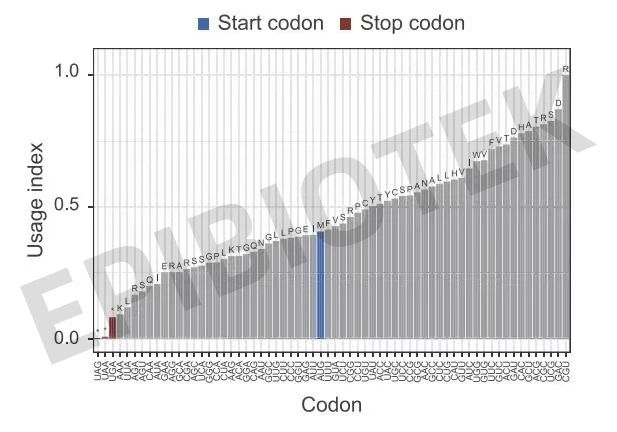
图6. 不同密码子使用频率分析
实测数据及分析结果示例 6
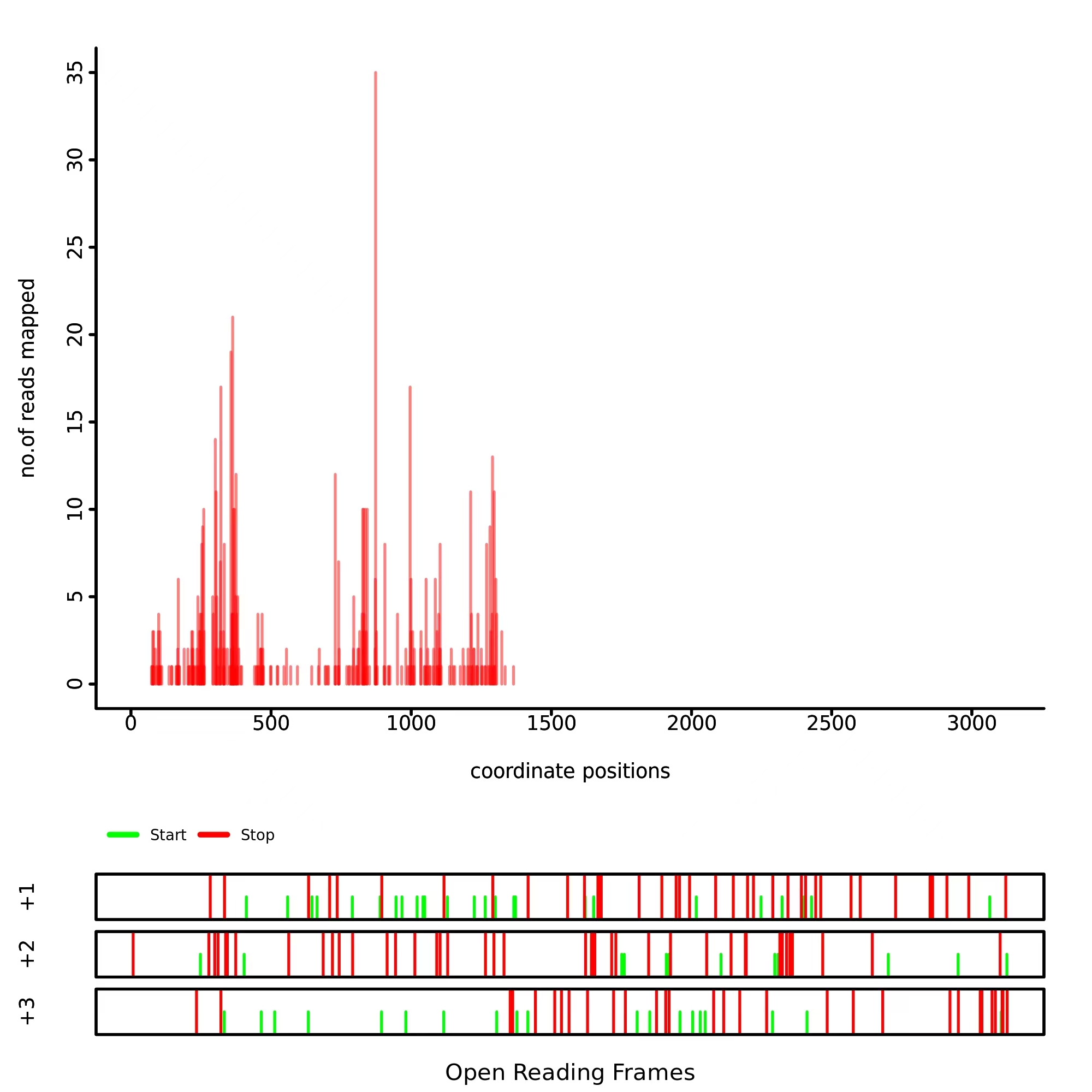
图7.翻译暂停分析
实测数据及分析结果示例 7
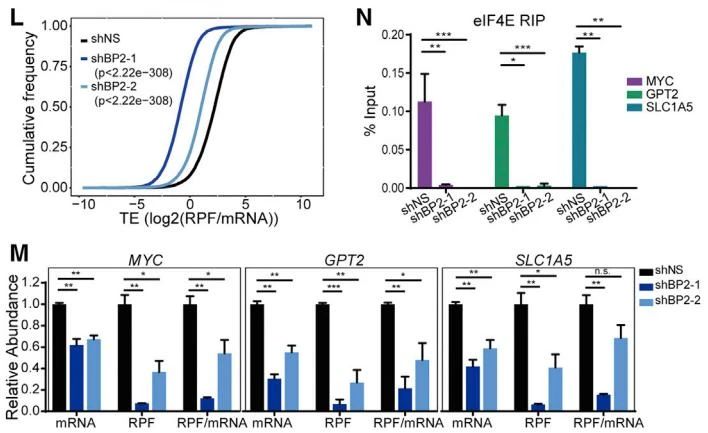
图7. 利用Ribo-seq发现IGF2BP2敲减导致全局翻译效率下降,该服务为表观生物提供。
客户文章
Cancer Cell:m6A甲基化“阅读器”IGF2BP2塑造AML谷氨酰胺代谢依赖性展[3]
此研究发现IGF2BP2作为m6A的阅读器通过调控下游基因GPT2、SLC1A5和MYC的mRNA稳定性和翻译从而促进急性髓性白血病(AML)的谷氨酰胺代谢过程,在AML的发生、发展、维持及干性中发挥了重要作用,并进一步开发以IGF2BP2为药物靶点的小分子抑制剂(CWI1-2),为AML的治疗提供新的靶点和策略。
![Blood:circMYBL2 通过募集 PTBP1 调控 FLT3 翻译,促进 FLT3-ITD 型急性髓系白血病进展[4]](http://openapi.whaleng.com/file-server/ep/2024/04/857c020419d94a8e81443af19ecc4b0a.png)
Blood:circMYBL2 通过募集 PTBP1 调控 FLT3 翻译,促进 FLT3-ITD 型急性髓系白血病进展[4]
此研究发现 circMYBL2 在 FLT3-ITD 突变型 AML 患者中的表达水平更高;在体外和体内实验中,敲降 circMYBL2 可抑制 FLT3-ITD AML 细胞的增殖,并促进其分化。在机制上,circMYBL2 可通过增加 PTBP1 与 FLT3 mRNA 的结合,提高FLT3 激酶的翻译效率。敲降 circMYBL2 可破坏对抑制剂耐药的 FLT3- ITD 阳性细胞的细胞活性,显著降低 FLT3 激酶的表达水平,进而使下游通路失活。本研究提示 circMYBL2 或可成为 FLT3-ITD AML 的潜在治疗靶点。
图8. 表观生物提供了 Ribo-seq 测序及生信分析服务。D. Ribo-seq 结果显示,circMYBL2 敲降后,MOLM-13 细胞基因的核糖体占位(翻译效率)发生变化;E. 敲降 circMYBL2 后,Ribo-seq 显示 FLT3 mRNA 的翻译效率下降。
参考文献
[1] Ingolia NT, Ghaemmaghami S, Newman JR, et al. Genome-wide analysis in vivo of translation with nucleo- tideresolution using ribosome profiling. Science, 2009, 324(5924): 218-223
[2] Gobet C, Naef F. Ribosome profiling and dynamic regulation of translation in mammals. Curr Opin Genet Dev. 2017Apr;43:120-127.
[3] Weng H, Huang F, Yu Z,et al. The mA reader IGF2BP2 regulates glutamine metabolism and represents a therapeutic target in acute myeloid leukemia.Cancer Cell 2022 Oct 26 (客户文章)点此阅读文章解读
[4] Sun YM, Wang WT, Zeng ZC, et al. circMYBL2, a circRNA from MYBL2, regulates FLT3 translation by recruiting PTBP1to promote FLT3-ITD AML progression. Blood. 2019 Oct 31;134(18):1533-1546.](客户文章)
[5] ChenH, Gao S, Liu W,et al. RNA N-Methyladenosine Methyltransferase METTL3 Facilitates Colorectal Cancer by Activating the mA-GLUT1-mTORC1 Axis and Is a Therapeutic Target. Gastroenterology 2021 03;160(4). (客户文章)
[6] Wei R, Cui X, Min J, et al. NAT10 promotes cell proliferation by acetylating CEP170 mRNA to enhance translation efficiency in multiple myeloma. Acta Pharm Sin B. 2022 Aug; 12(8): 3313–3325. (客户文章)
