SELECT 复旦表观团队揭示组蛋白修饰与m6A修饰共同调控小鼠胚胎干细胞异染色质形成
SELECT-m6A单位点定量检测技术自问世以来,非常火爆,使用了该技术的研究也陆续见刊,其中不乏高分文章。今天,Epi老师就通过其中一篇文章,跟大家分享SELECT技术的更多应用。
m6A修饰的功能研究,离不开三种甲基化相关的酶(老生常谈Writers、Erasers、Readers,见图1),所以目前的研究主要都集中于这几种相关基因/蛋白。其中,METTL3/METTL14复合物具有催化m6A修饰的作用,关于METTL3/METTL14对于细胞质中mRNA的调控研究更是数不胜数,但是METTL3是否参与以及如何参与调控染色质功能的研究相对较少。

图1. Writers(甲基转移酶)、Erasers(去甲基酶)、Readers(m6A结合蛋白)Nombela et al. Molecular Cancer (2021)
2021年1月,来自复旦大学生物医学研究院的研究团队在Nature发表了题为METTL3 regulates heterochromatin in mouse embryonic stem cells的研究成果。这篇文章研究的机制包括了染色质组蛋白修饰H3K9me3和H4K20me3以及m6A修饰,发现METTL3对m6A修饰的催化,对小鼠干细胞异染色质的完整性十分重要。

ChIP-seq发现组蛋白修饰与METTL3相关
一开始,研究者通过ChIP-seq技术发现METTL3在染色质上与异染色质修饰H3K9me3、H3K20me3在染色质上共定位,呈现很强的正相关;进一步的分析发现METTL3主要结合在内源性逆转录病毒(Endogenous retrovirus)IAPEz转座子亚群上。
通过构建Mettl3敲除细胞系以及METTL3 WT和METTL3 APPA酶活突变体挽救细胞等试验, 研究者发现敲除后METTL3结合位点的H3K9me3/H4K20me3水平显著降低,并且这种降低可以被METTL3 WT挽救,而不能被METTL3 APPA挽救,提示METTL3调控异染色质依赖于m6A修饰。
m6A修饰调控可变剪切?还是调控RNA稳定性?
那么m6A修饰是通过什么机制调控IAPEz元件的呢?从图1可以看到,常见的m6A修饰调控机制有:调控基因可变剪切(Splicing)、RNA稳定性(Stablity)、基因翻译效率(Translation)等。这些可以通过以下技术进行研究:
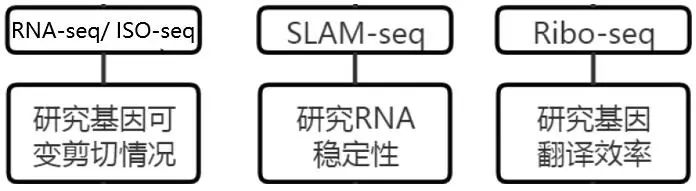
mRNA上的m6A修饰主要通过YTHDF1/2/3蛋白促进RNA降解(即调控RNA稳定性),研究者为了探究降解途径是否也参与IAPEz RNA的转录后修饰,进行了RNA半衰期实验(研究RNA稳定性),发现IAPEz RNA的半衰期在Mettl3 敲除以后并没有显著变化,而对照mRNA Nxt1的半衰期则显著上升,提示METTL3调控IAPEz RNA含量主要通过转录调控, 而不是转录后调控。研究者进一步分析了公共数据库的Ythdf1/2/3敲除细胞的RNA-seq数据,也发现IAPEz RNA的含量不会随着Ythdf1/2/3敲除而上升,进一步支持m6A修饰不是通过转录后调控来促进IAPEz的降解。
meRIP-seq发现m6A修饰集中于IAPEz
接下来,研究者使用meRIP-seq确定全转录组的m6A修饰图谱,发现在Mettl3 KO细胞中,编码基因的mRNA 3′端m6A富集被消除:

图2. IAPEz-int转录本上的m6A修饰:母细胞(parental cell)和Mettl3 KO细胞编码基因上的m6A平均富集水平;
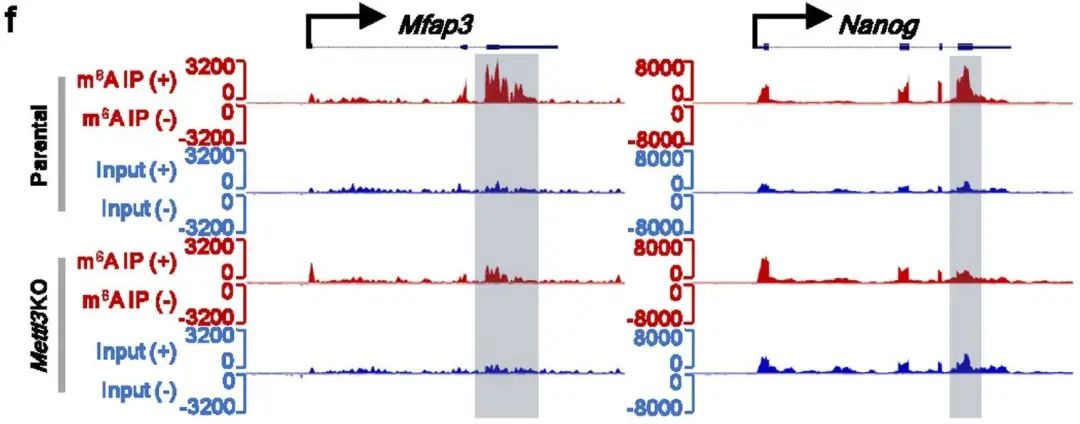
图3. IGV峰图显示m6A富集于编码基因3’端,但敲除Mettl3后的细胞中就没有富集了。
值得注意的是,依赖于IAP转录本的METTL3的m6A在其5′端而非3′端特异性富集(104个m6A峰定位于86个IAPEz元件):
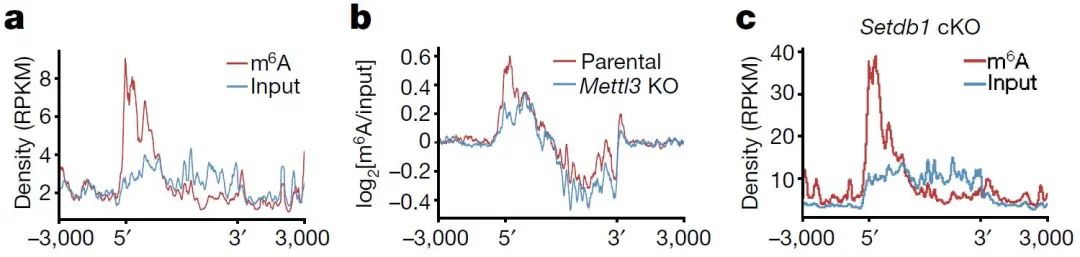
图4. 母细胞、mettl3 KO细胞和Setdb1 cKO细胞的m6A修饰在RNA结构上的分布情况
SELECT检测低表达转录本的m6A修饰单位点
考虑到meRIP–seq对表达水平低的转录本(比如IAPEz)的m6A修饰检测能力可能有限,而IAPEz RNA含量在Setdb1(H3K9me3甲基转移酶)敲除细胞可以大幅度上升,为了排除meRIP实验中使用的m6A抗体引起的潜在偏差,研究者使用SELECT验证了IAPEz-int共有序列5′端的五个腺苷位点上的m6A修饰:
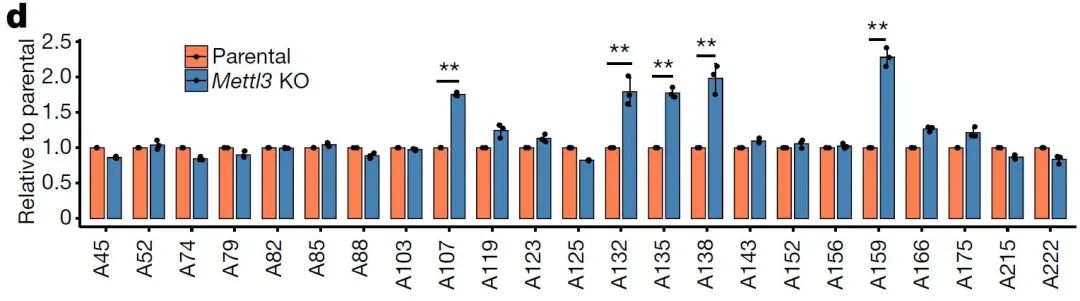
图5. SELECT结果显示母细胞和Mettl3 KO细胞的IAPEz-int共有序列腺苷位点上的m6A修饰
H3K9me3修饰与m6A修饰共同参与调控机制
研究者通过ChIP-qPCR发现METTL3和YTHDC1在染色质上有很好的共定位,YTHDC1结合染色质依赖于RNA的m6A修饰,失去m6A结合能力的YTHDC1的突变株在染色质的富集水平也会大幅度丢失,进一步支持YTHDC1结合染色质依赖于m6A修饰的假设。研究者还发现甲基催化酶SETDB1反过来也可以促进METTL3和YTHDC1在IAPEz转座子元件的富集。
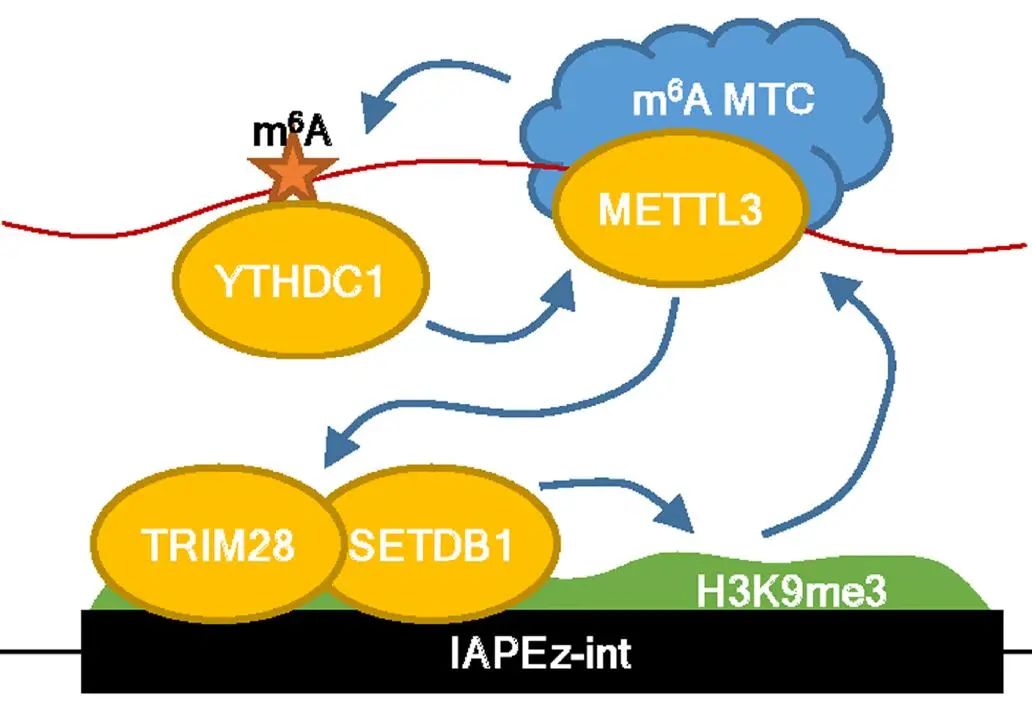
图6. 此研究发现METTL3可以结合小鼠胚胎干细胞ERV中的IAPEz转座子,通过招募SETDB1/TRIM28维持IAPEz转座子上的异染色质状态
已发表的SELECT应用文章(部分)
[1] Xu W, Li J, He C, et al. METTL3 regulates heterochromatin in mouse embryonic stem cells[J]. Nature, 2021, 591(7849): 317-321.(本文原文)
[2] Zhang, Z., Chen, T., Chen, HX. et al. Systematic calibration of epitranscriptomic maps using a synthetic modification-free RNA library. Nat Methods, 2021,18, 1213–1222.[3] Worpenberg L, Paolantoni C, Longhi S, et al. Ythdf is a N6-methyladenosine reader that modulates Fmr1 target mRNA selection and restricts axonal growth in Drosophila. EMBO J. 2021 Feb 15;40(4):e104975. [4] Dong L, Mao Y, Zhou A, et al. Relaxed initiation pausing of ribosomes drives oncogenic translation. Sci Adv. 2021 Feb 17;7(8):eabd6927.[5]Wang Y, Xiao Y, Dong S, et al. Antibody-free enzyme-assisted chemical approach for detection of N 6-methyladenosine[J]. Nat Chem Biol, 2020, 16(8): 896-903.
[6] Liu X M, Zhou J, Mao Y, et al. Programmable RNA N 6-methyladenosine editing by CRISPR-Cas9 conjugates[J]. Nat Chem Biol, 2019, 15 (9): 865-871.[7] Xiao Y, Wang Y, Tang Q, et al. An Elongation- and Ligation-Based qPCR Amplification Method for the Radiolabeling-Free Detection of Locus-Specific N6 -Methyladenosine Modification. Angew Chem Int Ed Engl. 2018 Dec 3;57(49):15995-16000.
SELECT-m6A修饰单位点定量检测
表观生物SELECT检测试剂盒填补了市场上特定位点m6A常规检测技术的空白,协助研究者鉴定m6A相关蛋白的作用靶点,深入挖掘m6A修饰的作用机制,现有跨年大促,欢迎点击了解!SELECT技术与我司meRIP-seq技术、ONT Direct RNA全长转录组测序技术、SLAM-seq技术结合,构建完备的m6A修饰研究解决方案;还可与ChIP-seq、HiChIP等表观基因组学进行创新组合,打破组学壁垒,冲刺高分文章!