表观研习班第一期130个免费学员名单全部出炉!
RNA修饰研究火爆,但是对它不够了解,无从下手?
m6A我知道,但这个ac4C又是什么?
如何利用现有技术研究疾病与RNA修饰的关系?
m6A生信分析有哪些好用的工具?
表观生物研习班帮你一次性解决上述所有问题!
研习班内容包括
RNA修饰与肿瘤和干细胞的研究课题设计
组蛋白修饰与RNA修饰共调控研究经验分享
m6A修饰的生信分析工具教学
更重要的是
本期研习班全体学员
!!学费全免!!


林水宾研究员
中山大学
2012年在美国佛罗里达大学医学院获得博士学位,随后加入哈佛大学医学院附属波士顿儿童医院从事博士后研究。2017年加入中山大学附属第一医院,任研究员、博士生导师。多年研究聚焦在RNA修饰的调控机理,揭示了RNA修饰对肿瘤发生、组织发育和再生疾病的调控功能,阐明了调控基因表达和疾病发生发展的RNA修饰组学和翻译组学新机理。以(共同)第一/通讯作者发表论文13篇,包括Nature、Mol Cell、Nat Commun 和Nat Rev Cancer 等。论文被引用次数超过一千次。作为课题负责人主持国家自然科学基金面上项目,Damon Runyon Cancer Research Foundation和Alex's Lemonade Stand Foundation等多项国内外科研基金。

周克任博士
中山大学
2014年于中山大学生命科学学院本科毕业,后保送中山大学博士研究生,师从屈良鹄教授,聚焦于基因的转录调控和RNA修饰的规律与功能研究。在博士期间,设计并建立了ChIPBase、dreamBase和starBase等RNA调控和功能研究平台,发现并解析组蛋白H3K36me3修饰指导m6A修饰的调控机制。目前已在Nature 和Nucleic Acids Research 杂志以第一作者或共同第一作者发表论文3篇,总引用率63。

王泽林博士
表观生物生信技术总监
2018年于中山大学获得博士学位,导师为屈良鹄教授,主要研究方向为非编码RNA与癌基因组学,一直专注于基因组学大数据分析和挖掘,擅长各种基因组学层面的大数据分析,精通基因组信息与临床表征信息的整合分析以及各种数据的可视化。熟悉机器学习和深度学习算法,设计并开发数据库和研究平台。其研究成果以第一作者或共同第一作者发表于European Urology、Cell Reports 等具有较高影响力的学术期刊上。
9:30
|
10:20
林
水
宾
课程名称:RNA表观遗传修饰对肿瘤和干细胞的调控功能和机制
1. RNA表观遗传修饰研究进展概述
2. RNA表观遗传修饰对肿瘤和干细胞的调控功能和机制研究举例
3. RNA表观遗传修饰研究课题设计和基金申请经验交流
10:25
|
11:15
周
克
任
课程名称:解析组蛋白H3K36me3修饰指导m6A修饰的调控机制
结合已发表文章,阐述组蛋白修饰与RNA修饰共调控机制及其研究技术、研究思路。
11:20
|
11:50
表观
生物
技术讲座:表观转录组研究相关测序技术及应用
13:30
|
14:15
王
泽
林
课程名称:m6A修饰研究的生物信息学方法
1. m6A常见分析内容解读
2. m6A常见数据库介绍和使用(MeT-DBV2.0和m6Avar数据库使用与介绍)
2.1 m6A和GWAS SNP关联分析
2.2 m6A与miRNA结合位点关联分析
2.3 m6A与剪切因子结合位点关联分析
2.4 m6A与RBP结合位点关联分析
2.5 m6A与肿瘤关系分析
14:20
|
15:05
3. m6A常用软件介绍和使用
3.1 GO和KEGG分析(DAVID WEB TOOL)
3.2 GSEA功能富集分析
3.3 M6A修饰位点预测(常见预测工具介绍和使用,SRAMP,m6A Finder,WHISTLE等)
3.4 IGV介绍和使用
3.5 韦恩图工具介绍和使用(JVENN)
3.6 常用R包介绍和使用
15:10
|
16:30
4. 学员实操练习
时间
导师
课程简介
11:50-13:30
午餐(科学城总部酒店餐厅,学员免费)
上课地点时间
广州科学城总部国际酒店3楼会议室,2019年7月6日(周六)。位于广州市科学大道,距离地铁6号线暹岗站B出口约200米。

第二批学员
- - - 推荐阅读 - - -

TCGA数据挖掘服务| 肿瘤相关表观遗传调控基因深度分析
TCGA(TheCancerGenomeAtlas),即癌症基因组图谱项目,始于2005年,由美国政府出资,美国国家癌症研究所(NationalCancerInstitute)和人类基因组研究所(NationalHumanGenomeResearchInstitute)共同监督,
2019-05-07

第二届医学表观遗传学前沿技术培训班圆满落幕!
2019年3月24日,为期三天的第二届医学表观遗传学前沿技术培训班在广州浙江大厦圆满落幕。 培训班现场 本次培训班邀请到了德克萨斯大学
2019-04-01
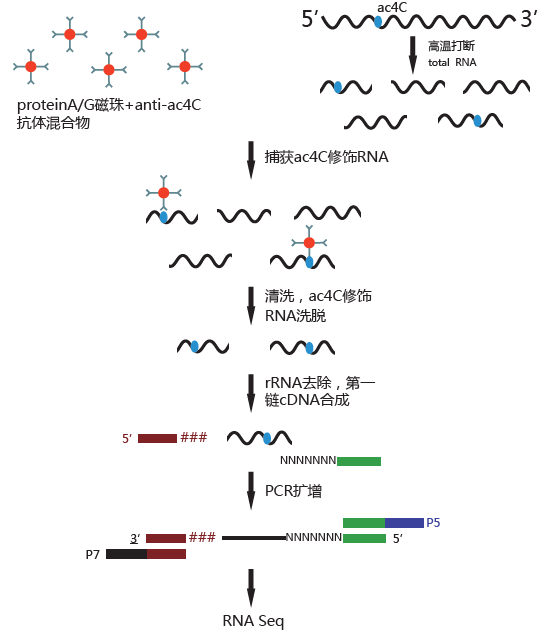
率先推出!RNA乙酰化ac4C测序acRIP-seq
N4-acetylcytidine(ac4C),N4位乙酰胞嘧啶,是真核原核生物中保守的化学修饰,早期研究认为ac4C主要存在tRNA和18SrRNA上[1]。而近期研究显示,mRNA上也存在大量的ac4C,该修饰在促进
2019-03-27
