【重磅发布】ChIRP-seq整体服务正式上线!
ChIRP( Chromatin Isolation by RNA Purification) [1]是一种同时分析lncRNA/circRNA[2] 、蛋白及DNA三者互作关系的实验方法。利用ChIRP-seq技术可用于在全基因范围内定位lncRNA/circRNA的结合位点和可能结合的其他RNA分子,是揭示位于细胞核内lncRNA/circRNA 生物学机制的关键实验手段。
表观生物结合自身团队技术优势,在国内率先为生物医学研究提供整体的ChIRP-seq技术服务,推动lncRNA/circRNA研究领域的发展。
项目流程:
ChIRP-seq流程包括ChIRP探针设计合成、lncRNA复合物交联、超声打断、探针杂交、DNA回收纯化、测序分析。
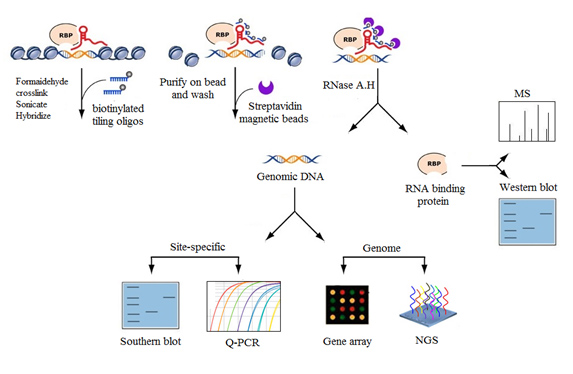
图1. ChRIP实验流程图
ChIRP-seq实验分组:
1、IP组:目标lncRNA/circRNA Odd组和Even组(即实验组,用于鉴定lncRNA/circRNA作用基因组位置)
2、lacZ组:外参对照组,证明ChIRP探针的特异性;
3、Input组:捕获前分离提取的基因组DNA,作为内参对照组,证明探针特异性;
4、Positive组:阳性对照组,通过已知验证有效的探针,通过WB检测已知结合蛋白质,证明整个ChIRP实验体系的有效性;
5、目标lncRNA/circRNA qPCR:证明ChIRP探针对目标lncRNA的正确结合及有效捕获。
样本要求:
细胞数量:细胞数量需要达到108个;提供活细胞或者提前做好交联处理的细胞样本。
生物信息分析:
1. 去接头污染,去低质量reads和测序质量评估
2. ChIRP测序序列与参考基因组序列的比对
3. ChIRP测序唯一reads在全基因组的分布
4. Odd探针组与Even探针组Common Peaks合并分析
5. Common Peaks鉴定及基因原件分析
6. Common Peaks相关基因筛选与GO功能聚类分析、Pathway分析
7. Common Peaks 可视化
8. Common Peaks Motif分析
实验周期:
60个工作日。
公司提供:
实验报告(材料、试剂、仪器、方法、数据分析、实验结果)。
其他注意事项:
1、目标lncRNA/circRNA的表达丰度:CT值最好在23以内(ACTB 16-17/GAPDH 18 );
2、如果目标lncRNA/circRNA表达丰度较低,需要进行过表达以确保ChIRP成功;
3、实验前需要确定目标lncRNA/circRNA定位于细胞核内发挥功能。
[1] Chu C, Qu K, Zhong F L, et al. Genomic maps of long noncoding RNA occupancy reveal principles of RNA-chromatin interactions[J]. Molecular cell, 2011, 44(4): 667-678.
[2] Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature structural & molecular biology, 2015, 22(3): 256-264.
- - - 推荐阅读 - - -

外泌体透射电镜检测服务上线通知
项目简介 在外泌体研究中,外泌体鉴定是继分离之后的又一关键步骤。你分离到的“外泌体”是否有较典型的外泌体结构?是否表达特异性的外泌体表面Marker?大小是否落在外泌体常规的粒径范围?这些都是外泌体研究者
2017-07-24
2017全国细胞外囊泡研讨会最终议程发布,诚邀参会!
欢迎莅临表观生物展台咨询和参与抽奖活动!我们将带来最新的外泌体 longRNA测序技术服务和 Nanosight外泌体浓度粒径检测服务等信息,同时现场还有精美抽奖礼品等您拿. 注:会议期间晚餐地点--珠江大酒楼一楼龙凤宫 表观生物展位:A7展台 欢迎莅临表观生物展台咨询和参与抽奖活动! 我们将带来最新的外泌体longRNA测序技术服务和Nanosight外泌体
2017-06-01
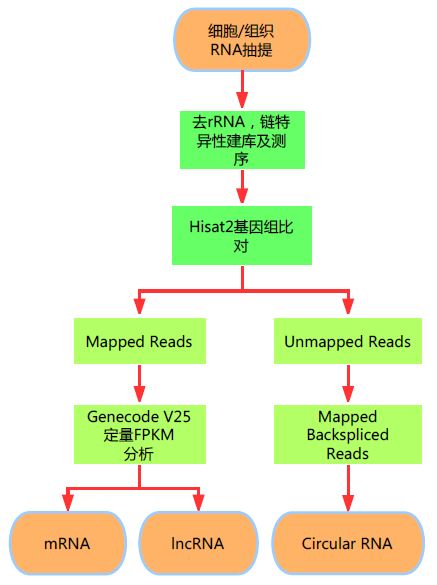
LongRNA-seq服务上线通知
即日起,表观生物LongRNA-seq技术服务正式上线了,详情请参阅下方内容! 项目简介 LongRNA-seq是一种全面检测细胞和组织长片段RNA(>200nt)的转
2017-05-12
